2. 模式识别国家重点研究室 (中国科学院 自动化研究所), 北京 100190
2. National Laboratory of Pattern Recognition (Institute of Automation, Chinese Academy of Sciences), Beijing 100190, China
医疗文本的信息抽取研究具有重要的理论意义和应用价值, 其在医疗领域的众多应用中如智慧医疗[1]、大数据诊断[2]和智能问诊[3]中都扮演着非常重要的上游任务角色. 由于医疗文本往往以非结构化的形式出现且包含大量具有特殊含义的医疗命名实体(例如“疾病”“症状”“身体部位”等), 而信息抽取的下游任务如医疗对话系统[4]、医疗知识图谱构建[5]等都依赖于对文本中的命名实体的识别结果. 因此如何自动、准确地抽取命名实体成为医疗信息分析和挖掘的一项关键技术.
对于医疗文本领域来说, 其文本中所包含的医疗实体构词形式复杂多样, 往往以陈述性短语形式出现(如症状类实体). 而中文又以字为语素的最小书写单位[6], 这就造成了中文医疗文本中经常会出现实体的嵌套现象. 如图1所示, 可以看到实体类别为“检查程序”的“X线”医疗实体被整个嵌套在了类别为“症状”的“X线心影显著增大”医疗实体的内部, 因此我们称“X线”和“X线心影显著增大”互为各自的内外层嵌套实体.
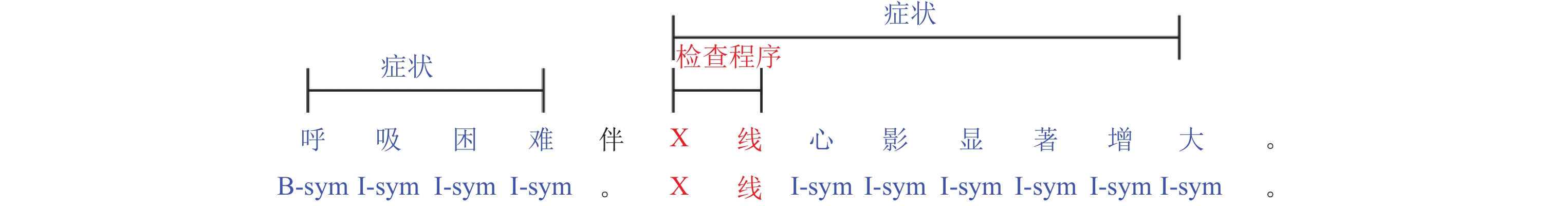
|
图 1 嵌套命名实体序列标注示例 |
近些年, 受益于深度神经网络的发展, 命名实体识别系统的性能得到了大幅提升[7-9]. 传统实体识别模型多使用基于文本序列标注的方法, 如encoder+CRF模型[10-12]来对命名实体进行识别. 由于序列标注方法需要将词语边界和词语类型同时进行输出(图1中“B”“I”“O”为边界, “-”后为实体类别), 因此这种方法只适用于实体边界没有重叠的非嵌套的命名实体, 如图1中的症状类实体“呼吸困难”. 而在处理“X线”这种和其他实体有边界嵌套的情况时, 仅依靠序列标注模型就难以进行处理. 为了解决对上述边界出现嵌套的实体识别问题, 本文提出将实体识别任务转化为实体的边界识别与边界首尾关系识别的联合任务. 如图2所示, 对于待识别的文本, 首先识别文本中所包含的所有实体的边界首字和尾字, 之后按照关系抽取的方法, 将每个具有边界配对关系(即首字和尾字隶属同一个实体)的首字和尾字组合进行关系抽取, 如实体“X线”与“X线心影显著增大”的识别可以转换为对“(首字, 尾字, 实体类别)”关系三元组的抽取, 即“(X, 线, 检查程序)”和“(X, 大, 症状)”.
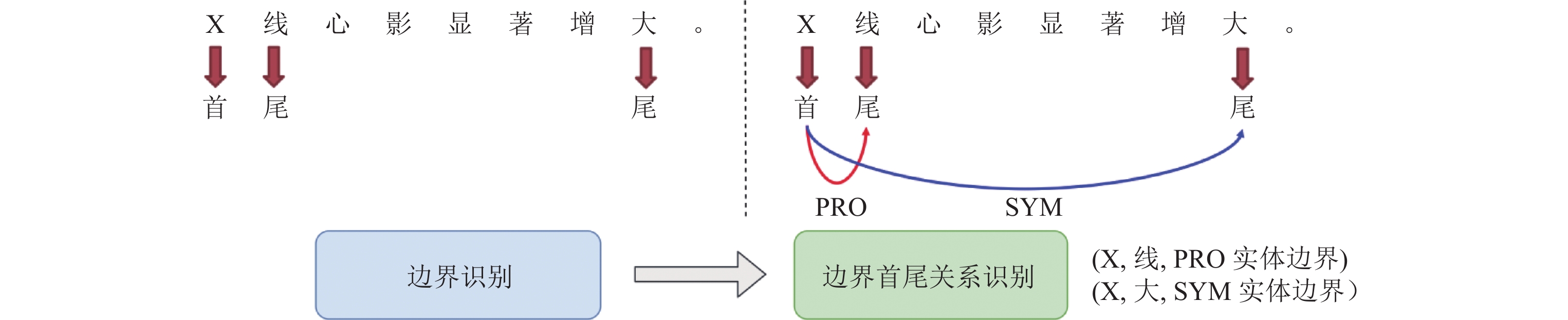
|
图 2 实体的边界识别与边界关系识别示例 |
Bekoulis等人[13]提出将关系抽取视为一个多头选择问题(multi-head selection), 借此来解决实体关系抽取中的实体关系重叠问题, 即每一个实体都可能与其他多个实体存在关系. 这与本文中所需要解决的实体边界字直接的关系抽取有共同之处. 因此本文借鉴多头选择机制的思想, 按照每一个实体的首字都可能与多个实体尾字拥有关系链接的顺序, 提出了改进后的多头选择机制. 与针对实体关系抽取任务所设计的初始多头选择机制不同, 我们设计的多头选择机制所针对的实体边界的首尾字关系和实体之间的关系在文本组成规则上有着较大的区别. 我们依照实际文本中的实体嵌套规则, 对实体首尾字之间的关系组合进行了严格限制, 例如, 同一组首尾字之间只能组成一种实体类别, 每个字都只能对其自身或相对位置靠后的字组成首尾关系等. 我们将在本文的第3节对其进行详细介绍.
和我们的想法近似, Yu等人[14]将biaffine机制引入实体识别任务, 并借鉴了依存关系解析(dependency parsing)的思想将实体识别任务看成识别首尾字的问题, 并对首尾字所形成的span赋予实体类别. 其方法本质上也是对实体中的“首-尾”组合进行关系抽取, 不过, Yu等人[14]的方法并没有具体考虑在一些特定文本领域, 如中文医疗文本中, 不同实体与实体之间存在着诸多特有的嵌套限制. 例如“药物”类型的实体不可能作为内层实体嵌套在“科室”类型的实体之中, 而“微生物”类实体则不可能成为“医学检测项目”的外层嵌套实体. 类似的文本规则往往由于其类型在训练数据中占比过低而无法被模型自动学习到, 因此我们提出利用层次过滤的方法, 将医疗实体的嵌套规则融合进实体识别模型的解码过程中, 从而达到提升模型准确率的目的.
此外, 针对以往的span-based方法往往忽略了实体边界信息的缺点[15], 我们提出了对实体边界字进行序列标注任务进行联合训练来提高模型对实体span信息的利用率. 我们将实体识别分为两部分, 首先对输入的文本进行实体边界首尾字的序列识别, 然后基于已识别出的首尾字特征信息, 再进行实体首尾字之间的关系抽取任务两项任务共享编码层并进行联合训练.
本文的主要贡献总结如下.
(1) 提出一种可以有效识别嵌套实体的新方法. 采用对实体边界识别和首尾字的边界关系识别的多任务联合训练的方式, 强化系统对实体边界信息的利用率.
(2) 提出一种基于医疗领域知识规则的过滤方法. 利用医疗文本领域中的实体嵌套规则来对实体识别结果进行过滤筛查.
(3) 在公开的中文医疗实体识别数据集上的实验评估显示, 本文所提方法在嵌套类型实体识别性能上显著优于已有的方法, 在整体准确率方面比最先进的方法提高了0.5%.
本文第2节对医疗嵌套实体识别的相关工作进行介绍. 第3节对我们所提出的模型的各个部分进行具体介绍. 第4节对我们所进行的实验进行介绍, 并进行实验分析. 第5节对本文进行总结.
2 相关工作命名实体识别(named entity recognition, NER)是信息抽取应用的重要上游任务, 其旨在将给定文本中所包含的具有特定意义的词语进行抽取. 早期的命名实体识别系统采用统计学的方式对文本进行自动识别, 如采用支持向量机(support vector machine, SVM)[16-18], 隐含马尔柯夫模型(hidden Markov model, HMM)[19-21]以及条件随机场(conditional random fields, CRF)[22, 23]等方式. 近些年, 越来越多的研究开始将深度学习技术应用于命名实体识别方法中并取得了不错的效果. Collobert等人[24]首先提出将卷积神经网络(convolutional neural network, CNN)作为编码器应用于NER系统中. Huang等人[10]和Lample等人[8]采用Bi-LSTM和CRF组成编码器-解码器框架解决长距离依赖问题. 语言模型技术也对NER研究提供了帮助. 利用预训练语言模型对不同NER任务进行finetune可以得到很好地识别效果, 如ELMo (embeddings from language models)[25]、BERT (bidirectional encoder representations from Transformers)[26]等.
对嵌套实体(nested entity)的识别一直是NER研究中的一个关键问题. 早期的研究工作采用人工制定的规则来辅助NER系统来处理嵌套实体的问题[27-29]. 当前对于嵌套实体的识别方法主要可以分为layer-based和span-based两种类型. 前者是解决嵌套实体识别最直观的方法. 根据嵌套命名实体结构的层次性质, layer-based模型通常包含多个层级. 每一层用于标识一组特定级别或长度的命名实体. Ju等人[30]提出通过由内向外动态叠加LSTM-CRF模型的层来识别嵌套实体. Wang等人[31]提出了采用堆叠NER多层结构抽取嵌套命名实体的模型, 每个层预测特定长度的文本区域是否为实体, 由底向上层层聚合的方式对实体边界信息进行识别. span-based方法通常将嵌套NER任务视为一个多分类问题, 并设计多种策略以在分类得到实体子序列文本边界的潜在表示. Zheng等人[32]提出boundary-aware模型, 通过使用序列标注模型识别实体候选边界从而定位实体位置, 然后基于候选实体边界来预测实体类别标签. Yu等人[13]使用基于图的依赖解析的思想来识别命名实体. 其首先利用Bi-LSTM来获得上下文表示, 然后应用两个独立的全连接层来表示实体边界的首尾边界信息并使用Bi-affine模型来预测句子中的命名实体. Su等人[33]利用全局归一化的思路, 将多个实体类型的识别视为Multi-head机制, 将每一个head视为一种实体类型识别任务, 并显式注入了相对位置信息. 上述的这些方法在识别嵌套实体的过程中, 都没有考虑在医疗文本特定领域下实体与实体之间的嵌套存在着不同的类别限制. 本文所提出的MTS-NER系统基于改进后的多头选择机制, 将从实际中文医疗文本数据的分析得到的实体之间的嵌套过滤规则融合进对实体的识别过程, 进一步提升了模型对中文医疗文本中的实体识别效果.
3 系统介绍本节将具体对基于多头选择的中文医疗实体识别系统(MTS-NER)的工作原理进行介绍. 不同于传统的基于序列标注方法的实体识别模型, 本方法将实体识别任务转化为实体的边界识别与边界首尾关系识别的联合任务. 如图3所示, 对于待识别的中文医疗文本首先进行Encoder层的编码生成word embedding (第3.1节), 然后通过CRF层对实体的边界信息进行序列生成, 并将word embedding和span embedding进行合并(第3.2节). 然后, 通过多头选择机制计算出每个字所链接的尾部边界字和其对应的实体类别(第3.3节). 最后, 系统根据中文医疗文本嵌套实体特征对最终结果进行进一步的筛选后输出实体识别结果(第3.4节). 以下将对系统各组成部分进行细节介绍.
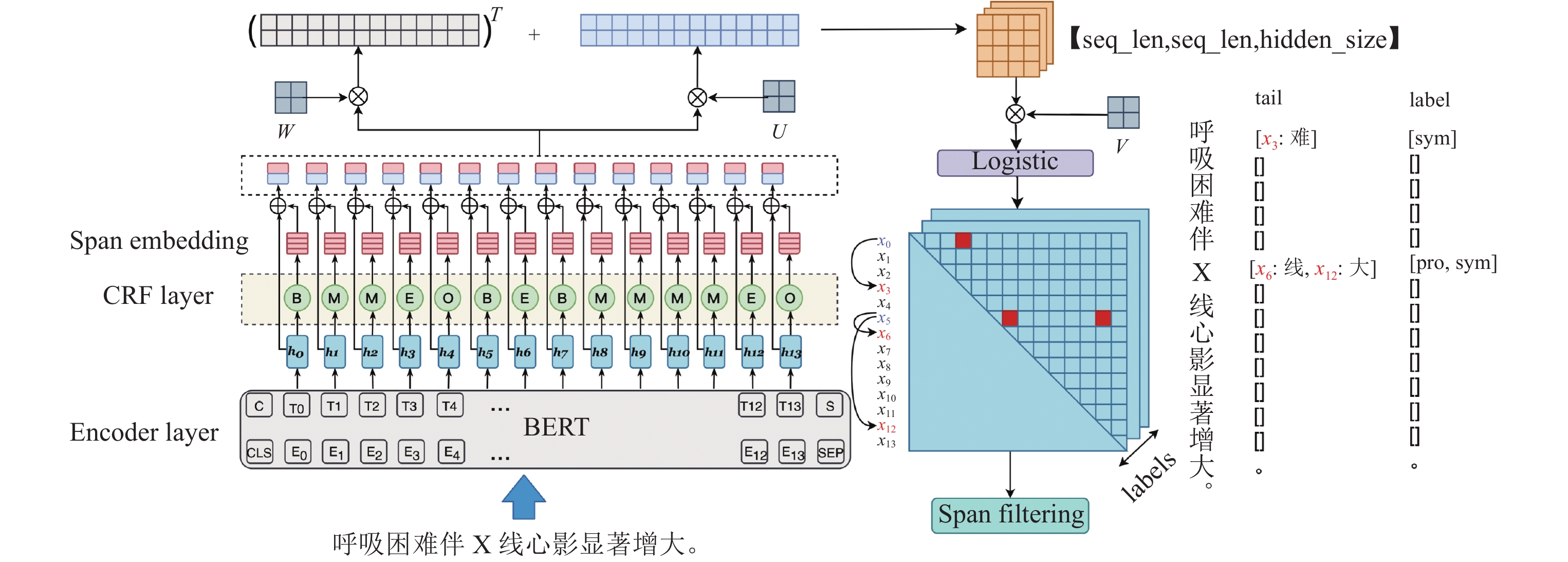
|
图 3 MTS-NER系统概览 |
3.1 编码层设计
本系统对中文文本输入进行字级别的编码操作, 采用BERT (bidirectional encoder representations from Transformers)预训练语言模型对输入进行编码. BERT模型使用双向注意力机制在大规模的无监督语料库上进行预训练从而获得预训练语言模型, 其结构包括自注意力编码器和下游任务层. 本系统利用已有的BERT预训练模型在命名实体任务上进行fine-tune操作, 从而优化句子中每个单词的上下文表示信息. 我们在句首和句尾分别用[CLS]和[SEP]标记, 并在句尾添加[PAD]标记使其长度等于最大序列长度. 给定一个输入序列
| $ {h}_{i}={\rm BERT}\left({x}_{i}\right) $ | (1) |
在具体实验中, 由于医疗数据文本与通用领域文本分布存在较大差异, 且中文医疗词汇存在长尾分布很难从普通数据集中学习, 因此我们使用基于医学的预训练模型MC-BERT[34]作为预训练语言模型对系统的BERT编码层进行初始化设置. 区别于以往的BERT模型采用全词掩码(whole word masking)来随机掩码普通词的做法, MC-BERT采用了全实体掩码(whole entity masking)和全跨度(whole span masking)来分别针对医学实体(如: “腹痛”)词和医疗短语(如: “腹部一阵一阵痛”)进行掩码操作, 从而将实体与语言领域知识注入表达学习模型中.
3.2 边界识别为了强化系统对实体边界信息的识别能力, 我们将单一的实体识别问题转化为实体边界和实体边界首尾字的关系联合训练问题. 此部分介绍本系统对实体边界的识别设计. 我们采用传统的序列标注方法对实体边界进行识别. 如图4所示, 我们对待识别文本中的实体字进行BMESO标注, 其中标签“B”表示此字为某类型实体的边界首字; 标签“E”表示此字为某类型实体的边界尾字; 标签“M”表示此字位于某类型实体内部且为非首尾字; 标签“S”表示此单一字构成实体; 标签“O”表示非实体字.

|
图 4 边界标注示例 |
需要注意的是, 这里的序列标注结果并不对不同实体之间的首尾字的隶属关系进行体现, 如图4中“大”同时为两个实体的边界尾字, 但这里都只用相同的尾字符号“E”进行标注. 我们采用传统的encoder+CRF序列标注结构对实体边界标签进行识别. 这种序列标注结构简单有效, 由上节所述编码层进行特征编码, 之后使用CRF层作为解码器进行序列解码. 具体而言, 给定长度为n的输入序列X, 解码器需要预测出其对应的标签向量
| $ p\left({y}\right|{x})=\frac{{\rm e}^{\mathit{Score}({x}, {y})}}{\displaystyle\sum _{{{y'}}\in {Y}_{x}}{\rm e}^{\mathit{Score}({x}, {{y'}})}} $ | (2) |
这里的
| $ \mathit{Score}({x}, {y})=\sum _{i=0}^{n}A[{y}_{i-1}, {y}_{i}]+\sum _{i=i}^{n}{P}_{i}\left[{y}_{i}\right] $ | (3) |
其中,

|
图 5 Score 计算示例 |
| $ \begin{array}{cc}los{s}_{\rm span} =-\log\left(\dfrac{{\rm e}^{\mathit{Score}({x}, {y})}}{\displaystyle\sum _{{{y'}}\in {Y}_{x}}{\rm e}^{\mathit{Score}({x}, {{y'}})}}\right) =\displaystyle\sum _{{{y'}}\in {Y}_{x}}{\rm e}^{\mathit{Score}({x}, {{y'}})}-\mathit{Score}({x}, {y})\end{array} $ | (4) |
在得到实体边界标签的预测序列
我们将嵌套实体的识别任务定义为实体边界的多头选择问题, 即对于每一个待识别文本中的字
| $ g({{\textit{z}}}_{i}, {{\textit{z}}}_{j}, {r}_{ij})=Vf(W{{\textit{z}}}_{i}+U{{\textit{z}}}_{j}+b) $ | (5) |
其中,
| $ \Pr({r}_{k}, {x}_{j}|{x}_{i})=\mathit{Logistic}(g({{\textit{z}}}_{i}, {{\textit{z}}}_{j}, {r}_{k})) $ | (6) |
具体到实际操作过程中对张量的计算, 如图3中红色虚线框中所示, 在通过全连接层(图3中W, U)分别得到对头字和尾字的状态表示后, 我们将其中一方进行转置操作, 然后利用张量维度广播机制将两个张量进行加和操作, 从而得到维度为[seq_len, seq_len, hidden_size]的输出张量, 其中seq_len表示待识别文本序列的长度n, hidden_size表示全连接层隐层维度dr. 然后再将其通过全连接层V生成维度为[seq_len, seq_len, label_size]的输出张量G, 其中label_size为实体类别的数目. G即为所有
| $ los{s}_{\rm MTS}=\sum _{{i}}^{{n}}\sum _{j}^{m}-\log \Pr(\hat{{x}_{j}}, {r}_{ij}|{x}_{i}\theta ) $ | (7) |
其中, m为
| $ los{s}_{\rm joint}=los{s}_{\rm span}+los{s}_{\rm MTS} $ | (8) |
与实体关系抽取或依存句法分析任务不同, 实体边界首尾字匹配问题对首尾字之间的相对位置有着一定的限制条件, 例如第3.3节中已提及的边界首字和尾字之间存在
| 表 1 实体之间嵌套规则表览 |
| 表 2 CMeEE数据集中不同实体类型之间的嵌套关系数量统计 |
算法1. 基于实体嵌套规则的层级式过滤算法.
输入:
输出:
初始化: 按照
1. while
2.
3. while
4.
5.
6.
7.
8. if is_nested
9. if not is_matched
10.
11. 过滤掉
12. end if
13. else if is_flat
14. continue
15. else
16.
17. 过滤掉
18. end if
19. end while
20. end while
21. return
在本节中, 我们将通过对实际应用中的中文医疗命名实体数据集进行大量实验来证明我们所提出的系统框架的有效性, 并针对框架结构的不同组成部件进行具体的分析来显示其作用和优势.
4.1 数据设置我们使用中文医疗命名实体抽取数据库(CMeEE), 其数据来源于中文医疗信息处理评测基准CBLUE(Chinese biomedical language understanding evaluation)下属子任务评测. 其共包含了20000条中文医疗领域语句, 通过统计, 数据中共包含9大类医疗实体类别, 具体如后文表3所示. 其中, 具有嵌套关系的实体对(外层实体-内层实体)数量共4855对. 可以看出, 大部分外层实体为sym类型, 单一字构成实体大部分为bod类型. 在具体的实验中, 我们发现实体类别嵌套关系出现次数小于3次的情况基本上都为数据的错标漏标所造成, 因此为了避免对第3.4节所介绍的span filtering产生干扰, 我们对出现次数小于3次的类别约束规则进行了删除. 在实验中, 我们将数据集按照训练集:开发集:测试集为15000:2500:2500的数量随机进行切分, 并采用3折交叉验证方式进行测试. 我们采用精确率, 召回率和F1值对系统的实体识别结果进行评估.
| 表 3 数据集实体类别统计 |
4.2 实验设置
主要参数设置如表4所示, 我们使用中文医疗预训练模型MC-BERT (https://github.com/alibaba-research/ChineseBLUE)作为预训练语言模型对系统的BERT编码层进行初始化设置, 其layer大小为12, hidden size大小为768. 我们采用Adam作为优化器, learning rate大小为
| 表 4 主要参数设置 |
4.3 基线设置
为了证明我们所提出的MTS-NER模型在嵌套命名实体识别的结果上具有优势, 我们采用了以下几种模型作为基线系统进行结果对比.
● BiLSTM-CRF[8]: 使用Bi-LSTM作为编码器, CRF作为解码器的基础命名实体识别模型, 不具备对嵌套实体的识别能力.
● Pyramid[31]: 一种layer-based模型, 采用堆叠NER多层结构抽取嵌套命名实体的模型, 由底向上层层聚合的方式对实体边界信息进行识别.
● Bi-affine[14]: 一种span-based模型, 采用双仿射注意力机制对实体首尾边界信息进行交互打分, 具有识别嵌套命名实体的能力.
● GlobalPointer[33]: 一种span-based模型, 利用全局归一化的思路, 将多个实体类型的识别视为multi-head机制, 将每一个head视为一种实体类型识别任务. 兼容了嵌套实体和非嵌套实体的识别, 在多个数据集上取得SOTA成绩.
4.4 整体结果表5列出了MTS-NER和对比方法的实验结果.
| 表 5 不同模型在CMeEE数据集上的性能表现 |
从表5中可以看出, 相较于其他方法, MTS-NER在多个单独实体类别和总体的F1值上都取得了最高得分0.642, 对比于传统方法BiLSTM-CRF、有着7.3%的提升, 和同样具备嵌套实体识别能力的Pyramid、Bi-affine和GlobalPointer方法对比, F1值分别有着1%、2.4%和0.1%的提升, 准确率方面分别有着4%、5.5%和0.5%的提升. 与同为span-based模型的GlobalPointer方法结果对比中可以发现, 尽管在总的F1值上差距不明显, 但在一些如“bod”“pro”和“ite”这种多为内部嵌套的实体类别对比上, MTS-NER具有明显优势, 准确率分别有着3%、8.3%和2.2%的提升. 其中, “bod”类别实体的F1值较之GlobalPointer有着3.7%的提升. 对比表2中所统计不同类别实体嵌套关系数量可以发现, “bod”类别实体作为其他类型实体的内部嵌套实体的情况明显高于其余实体类别, 这印证了我们的方法所制定的针对不同实体嵌套模式和实体类型的过滤方式的有效性. 在第4.5.2节我们还将进一步讨论过滤规则的影响.
4.5 系统影响因素分析在本文中, 我们提出了实体边界识别和嵌套规则过滤两个机制来提升多头选择机制对实体识别的效果. 为了具体验证系统中不同模块对系统整体性能的影响, 我们进行了消融实验来对每个模块的作用进行具体分析. 我们对系统模块进行了如下实验: (1)完整包含实体边界识别联合训练以及实体嵌套规则过滤; (2)不进行边界识别联合训练, 只采用编码层中的隐层信息单独训练多头识别模型并进行实体嵌套规则过滤; (3)进行实体边界识别和多头识别模型的联合训练但去除实体嵌套规则过滤; (4)只进行多头识别模型的训练, 去除实体边界识别联合训练以及实体嵌套规则过滤步骤. 实验结果如表6所示. 可以看到, 本文所提出的两种针对实体边界的改进机制都对模型有着正向的性能影响. 对比于原始的多头选择模型, 加入实体边界识别信息联合训练和加入实体嵌套规则过滤的模型分别有着0.4和0.7的F1值提升. 接下来我们将对这两个机制进行具体的分析.
| 表 6 MTS-NER消融实验结果 |
4.5.1 实体边界标注信息影响
在具体的实验中, 我们一共设计了两种针对实体首尾边界的标注方式, 除了第3.2节中所提到的“BMEOS”方式外, 我们还对“BMEOS+type”方式进行了实验, 其标注方式如图6所示.
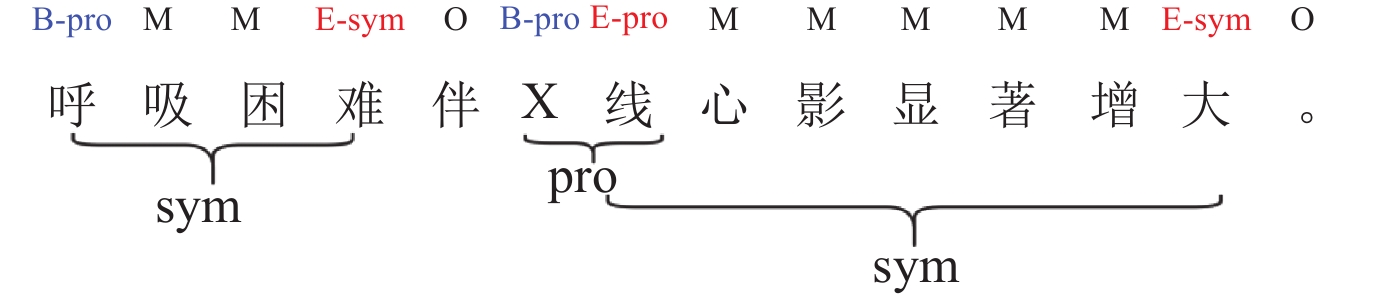
|
图 6 “BMEOS+type”方式边界标注示例 |
在“BMEOS+type”标注方式下, 模型的序列标注模块所识别出的标签同时包含了实体边界信息和实体边界字所隶属的内层实体类别信息.
表7给出了模型在两种边界字标注方式下的F1值表现(未进行实体嵌套规则过滤), 其中“span-F1”和“entity-F1”分别表示模型的序列标注模块对字边界的识别性能和融合边界信息后模型整体对实体的识别性能. 可以看出, 尽管采用“BMEOS+type”方式进行实体边界字标注可以包含更多的实体类别信息, 但是在最终的F1值性能体现上和“BMEOS”方式具有较大差距. 我们分析造成此种现象的主要原因是, 过多的标签种类造成序列标注的准确率下降, 继而导致将大量的噪声数据错误传递给了接下来的多头选择模块, 从而使得模型整体性能进一步下降. 因此我们选择较为简约的“BMEOS”标注方式对实体边界字进行标注并联合训练.
| 表 7 不同边界字标注方式下的性能表现 |
4.5.2 过滤规则影响
在实验过程中, 我们对中文医疗命名实体抽取数据库(CMeEE)的数据所包含的嵌套实体类别数目进行了统计分析, 结果如表2所示. 其中, “entity1”和“entity2”分别表示外层实体和内层嵌套实体. 从统计结果可以看出, 绝大部分的外层实体的类别为“sym”, 而内层嵌套实体多由“bod”“dis”和“ite”等构成. 结合表5中所列的对应的“bod”“dis”和“ite”的性能表现可以发现, 本文所提出的方法在对应实体的精确率P上都有着相对其他方法较大的提升. 我们分析这是由于各个类型的实体其对应的可选外层嵌套实体类型范围相对较小, 例如表7中所示实体类型为“bod”“equ”“mic”“pro”和“sym”的内层实体其对应的外层嵌套实体类型只有“sym”一种类型. 因此当系统基于嵌套规则的过滤方法在解码过程中一旦内层实体类型确定之后, 不属于其对应范围的外层噪声实体解码结果将被直接过滤掉.
例如表8中例子1所示, 过滤之前的结果错误地将两个独立的“(肝, bod)、(肾, bod)”识别为了一组嵌套实体“(肝、肾, bod)、(肾, bod)”, 而通过系统的嵌套规则过滤机制, 由于内层嵌套实体为“bod”类型且获得比外层实体更高的条件概率得分, 因此别错误识别的外层嵌套实体候选“(肝、肾, bod)”就被删除, 因而提升了实体识别的准确率. 而如实体类型“sym”则由于其常和多种类型的实体都能组成嵌套实体对, 因而所受规则过滤的影响相对有限. 另一方面, 由于原始的多头选择机制在获取条件概率时所使用的函数为Logistic函数, 因而在同一组识别的实体首尾字所组成的实体可能解码出现多种实体类别. 如表8中的例子2所示, 可以看到, 原始的系统将实体“血凝块”同时识别出了两种类型“(血凝块, bod)”和“(血凝块, dis)”, 这显然并不符合实体识别任务的实际情况. 而如第3.4节所介绍的, 我们的嵌套规则过滤方法会对同组实体首尾边界组合进行排序且在非嵌套实体的情况只保留最高得分组合, 因此在保留候选词多样性的基础上避免了Logistic函数带来的噪声类别问题, 从而进一步提升了模型的识别准确率.
| 表 8 医疗嵌套实体识别结果示例 |
5 总 结
本文提出了一种融合实体嵌套规则的中文实体识别方法MTS-NER, 将实体边界的识别任务转化为了实体的边界识别与边界首尾关系识别的联合训练任务. 通过对实体关系抽取所常用的多头选择机制进行改进使其能够适用于嵌套实体识别的任务. 在解码阶段, 我们提出了基于实体嵌套规则的过滤方法, 对不符合医疗实体边界嵌套规则和实体类别嵌套规则的识别结果进行层级过滤, 从而使得识别结果能够符合真实医疗文本中的内外层实体嵌套组合的组成规律. 我们在公开医疗实体识别数据集上的实验结果表明了该方法的有效性. 在未来我们将继续对我们的规则方法进行优化以适用于更多不同类型的实体识别任务.
| [1] |
Cowie MR, Blomster JI, Curtis LH, Duclaux S, Ford I, Fritz F, Goldman S, Janmohamed S, Kreuzer J, Leenay M, Michel A, Ong S, Pell JP, Southworth MR, Stough WG, Thoenes M, Zannad F, Zalewski A. Electronic health records to facilitate clinical research. Clinical Research in Cardiology, 2017, 106(1): 1-9.
[doi:10.1007/s00392-016-1025-6] |
| [2] |
Denaxas SC, Morley KI. Big biomedical data and cardiovascular disease research: Opportunities and challenges. European Heart Journal-quality of Care and Clinical Outcomes, 2015, 1(1): 9-16.
[doi:10.1093/ehjqcco/qcv005] |
| [3] |
Li I, Pan J, Goldwasser J, Verma N, Wong WP, Nuzumlalı MY, Rosand B, Li YX, Zhang M, Chang D, Taylor RA, Krumholz HM, Radev D. Neural natural language processing for unstructured data in electronic health records: A review. Computer Science Review, 2022, 46: 100511.
[doi:10.1016/j.cosrev.2022.100511] |
| [4] |
Li M, Xiang L, Kang XM, Zhao Y, Zhou Y, Zong CQ. Medical term and status generation from Chinese clinical dialogue with multi-granularity transformer. IEEE/ACM Trans. on Audio, Speech, and Language Processing, 2021, 29: 3362-3374.
[doi:10.1109/TASLP.2021.3122301] |
| [5] |
Sun J, Zhou Y, Zong CQ. One-shot relation learning for knowledge graphs via neighborhood aggregation and paths encoding. ACM Trans. on Asian and Low-resource Language Information Processing, 2021, 21(3): 52.
[doi:10.1145/3484729] |
| [6] |
Zhou YH. About modern Chinese morphemes. Journal of Southwest University for Nationalities (Philosophy and Social Sciences), 2001, 22(7): 202–205 (in Chinese with English abstract).
|
| [7] |
Chiu JPC, Nichols E. Named entity recognition with bidirectional LSTM-CNNs. Trans. of the Association for Computational Linguistics, 2016, 4: 357-370.
[doi:10.1162/tacl_a_00104] |
| [8] |
Lample G, Ballesteros M, Subramanian S, Kawakami K, Dyer C. Neural architectures for named entity recognition. In: Proc. of the 2016 Conf. of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies. San Diego: ACL, 2016. 260–270.
|
| [9] |
Dong CH, Zhang JJ, Zong CQ, Hattori M, Di H. Character-based LSTM-CRF with radical-level features for Chinese named entity recognition. In: Proc. of th 5th CCF Conf. on Natural Language Processing and Chinese Computing (NLPCC 2016), and the 24th Int’l Conf. on Computer Processing of Oriental Languages. Kunming: Springer, 2016. 239–250.
|
| [10] |
Huang ZH, Xu W, Yu K. Bidirectional LSTM-CRF models for sequence tagging. arXiv:1508.01991, 2015.
|
| [11] |
Sheikhshab G, Birol I, Sarkar A. In-domain context-aware token embeddings improve biomedical named entity recognition. In: Proc. of the 9th Int’l Workshop on Health Text Mining and Information Analysis. Brussels: ACL, 2018. 160–164.
|
| [12] |
Li XN, Yan H, Qiu XP, Huang XJ. FLAT: Chinese ner using flat-lattice transformer. In: Proc. of the 58th Annual Meeting of the Association for Computational Linguistics. Online: ACL, 2020. 6836–6842.
|
| [13] |
Bekoulis G, Deleu J, Demeester T, Develder C. Joint entity recognition and relation extraction as a multi-head selection problem. Expert Systems with Applications, 2018, 114: 34-45.
[doi:10.1016/j.eswa.2018.07.032] |
| [14] |
Yu JT, Bohnet B, Poesio M. Named entity recognition as dependency parsing. In: Proc. of the 58th Annual Meeting of the Association for Computational Linguistics. ACL, 2020. 6470–6476.
|
| [15] |
Shen YL, Ma XY, Tan ZQ, Zhang S, Wang W, Lu WM. Locate and label: A two-stage identifier for nested named entity recognition. In: Proc. of the 59th Annual Meeting of the Association for Computational Linguistics and the 11th Int’l Joint Conf. on Natural Language Processing (Vol. 1: Long Papers). ACL, 2021. 2782–2794.
|
| [16] |
Isozaki H, Kazawa H. Efficient support vector classifiers for named entity recognition. In: Proc. of the 19th Int’l Conf. on Computational Linguistics. Taipei: ACL, 2002. 1–7.
|
| [17] |
Lee KJ, Hwang YS, Kim S, Rim HC. Biomedical named entity recognition using two-phase model based on SVMs. Journal of Biomedical Informatics, 2004, 37(6): 436-447.
[doi:10.1016/j.jbi.2004.08.012] |
| [18] |
Ju ZF, Wang J, Zhu F. Named entity recognition from biomedical text using SVM. In: Proc. of the 5th Int’l Conf. on Bioinformatics and Biomedical Engineering. Wuhan: IEEE, 2011. 1–4.
|
| [19] |
Zhou GD, Su J. Named entity recognition using an HMM-based chunk tagger. In: Proc. of the 40th Annual Meeting of the Association for Computational Linguistics. Philadelphia: ACL, 2002. 473–480.
|
| [20] |
Zhao SJ. Named entity recognition in biomedical texts using an HMM model. In: Proc. of the 2004 Int’l Joint Workshop on Natural Language Processing in Biomedicine and its Applications. Geneva: COLING, 2004. 87–90.
|
| [21] |
Zhang J, Shen D, Zhou GD, Su J, Tan CL. Enhancing HMM-based biomedical named entity recognition by studying special phenomena. Journal of Biomedical Informatics, 2004, 37(6): 411-422.
[doi:10.1016/j.jbi.2004.08.005] |
| [22] |
McCallum A, Li W. Early results for named entity recognition with conditional random fields, feature induction and Web-enhanced lexicons. In: Proc. of the 7th Conf. on Natural Language Learning at HLT-NAACL 2003. Edmonton: ACL, 2003. 188–191.
|
| [23] |
Settles B. Biomedical named entity recognition using conditional random fields and rich feature sets. In: Proc. of the 2004 Int’l Joint Workshop on Natural Language Processing in Biomedicine and its Applications. Geneva: COLING, 2004. 107–110.
|
| [24] |
Collobert R, Weston J, Bottou L, Karlen M, Kavukcuoglu K, Kuksa P. Natural language processing (almost) from scratch. Journal of Machine Learning Research, 2011, 12: 2493-2537.
|
| [25] |
Peters ME, Neumann M, Iyyer M, Gardner M, Clark C, Lee K, Zettlemoyer L. Deep contextualized word representations. In: Proc. of the 2018 Conf. of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies, Vol. 1 (Long Papers). New Orleans: ACL, 2018. 2227–2237.
|
| [26] |
Hakala K, Pyysalo S. Biomedical named entity recognition with multilingual BERT. In: Proc. of the 5th Workshop on BioNLP Open Shared Tasks. Hong Kong: ACL, 2019. 56–61.
|
| [27] |
Shen D, Zhang J, Zhou GD, Su J, Tan CL. Effective adaptation of hidden markov model-based named entity recognizer for biomedical domain. In: Proc. of the 2003 ACL Workshop on Natural Language Processing in Biomedicine. Sapporo: ACL, 2003. 49–56.
|
| [28] |
Zhou GD, Zhang J, Su J, Shen D, Tan C. Recognizing names in biomedical texts: A machine learning approach. Bioinformatics, 2004, 20(7): 1178-1190.
[doi:10.1093/bioinformatics/bth060] |
| [29] |
Zhou GD. Recognizing names in biomedical texts using mutual information independence model and SVM plus sigmoid. Int’l Journal of Medical Informatics, 2006, 75(6): 456-467.
[doi:10.1016/j.ijmedinf.2005.06.012] |
| [30] |
Ju MZ, Miwa M, Ananiadou S. A neural layered model for nested named entity recognition. In: Proc. of the 2018 Conf. of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies, Vol. 1 (Long Papers). New Orleans: ACL, 2018. 1446–1459.
|
| [31] |
Wang J, Shou LD, Chen K, Chen G. Pyramid: A layered model for nested named entity recognition. In: Proc. of the 58th Annual Meeting of the Association for Computational Linguistics. ACL, 2020. 5918–5928.
|
| [32] |
Zheng CM, Cai Y, Xu JY, Leung HF, Xu GD. A boundary-aware neural model for nested named entity recognition. In: Proc. of the 2019 Conf. on Empirical Methods in Natural Language Processing and the 9th Int’l Joint Conf. on Natural Language Processing. Hong Kong: ACL, 2019. 357–366.
|
| [33] |
Su JL, Murtadha A, Pan SF, Hou J, Sun J, Huang WW, Wen B, Liu YF. Global pointer: Novel efficient span-based approach for named entity recognition. arXiv:2208.03054, 2022.
|
| [34] |
Zhang NY, Jia QH, Yin KP, Dong L, Gao F, Hua NW. Conceptualized representation learning for Chinese biomedical text mining. arXiv:2008.10813, 2020.
|
| [6] |
周永惠. 关于现代汉语语素. 西南民族学院学报·哲学社会科学版, 2001, 22(7): 202–205.
|
 2024, Vol. 35
2024, Vol. 35


