2. 陕西师范大学 生命科学学院, 陕西 西安 710062;
3. 武警工程大学 信息工程学院, 陕西 西安 710086
2. College of life Sciences, Shaanxi Normal University, Xi'an 710062, China;
3. College of Information Engineering, Engineering University of PAP, Xi'an 710086, China
生物测序技术的迅速发展实现了大规模基因表达数据的自动获取, 为癌症等疾病的发病机理和诊断研究提供了新途径[1-3].然而, 基因表达数据具有高维小样本特点, 包含了大量与疾病无关的基因(冗余基因)[4-6].因此, 选取具有高分类信息的基因子集是分析基因表达数据的首要任务[5, 7].特征选择可以筛选出与分类任务高度相关的基因, 提高分类准确率[8].
特征选择是从原始特征集合中选取具有强分类信息且尽可能相互独立的特征构成特征子集, 以尽可能地保留原始系统的分类信息且包含尽可能少的特征, 从而达到去除冗余特征、提高分类准确率的目的.根据与分类器的关系, 特征选择算法分为Filter、Wrapper和Embedded方法[9-11].根据是否使用类标信息, 特征选择算法又分为有监督特征选择方法和无监督特征选择方法.
1 相关工作介绍有监督特征选择方法通过计算特征与类标的相关性进行特征选择, 如Relief算法[12]、mRMR(minimal redundancy-maximal relevance)算法[13]、CFS(correlation-based feature selection for machine learning)算法[14]等.然而, 样本类标往往很难获得, 因此无监督特征选择算法引起了研究者的关注.
Dash等人[15]提出基于熵排序的无监督特征选择算法, 利用信息熵度量特征重要性程度, 从而选择最优特征子集; 徐峻岭等人[16]提出基于互信息的无监督特征选择算法, 利用互信息定义特征的相关度与冗余度, 综合考虑特征的相关度与冗余度来评价特征重要性; 张莉等人[17]提出基于K-均值聚类的无监督特征选择算法, 利用特征对聚类结果的影响以及特征之间的相关性作为特征选择的判别标准; He等人[18]针对无监督特征选择算法多是Wrapper方法, 提出独立于任何学习算法的Filter特征选择方法——Laplacian score无监督特征选择算法, 利用同类样本距离更近原理, 对每个特征计算其拉普拉斯分数以反映其局部保持能力, Laplacian score越小的特征其局部保持能力越强, 重要度越高, 越具有代表性, 需要说明的是, 该算法也可以通过监督的方式执行; Cai等人[19]提出多类簇无监督特征选择算法MCFS(multi-cluster feature selection), 使用谱聚类技术, 然后求解带有L1正则项的最小二乘问题, 并定义特征的MCFS score, 选择MCFS score位于最前面的若干个特征, 使选择的特征既能保留更多数据类簇结构, 又能覆盖所有可能类簇的特征; 王连喜等人[20]提出了一种基于聚类集成的特征选择算法, 利用聚类算法将冗余特征聚成一类簇, 然后从各类簇挑选代表性特征构成最优特征子集; Zhao等人[21]基于谱图理论提出SPEC算法, 以特征值的分布与目标的概念是否一致作为评价准则进行特征选择; 我们团队提出基于密度峰值的无监督特征选择算法, 分别定义了特征密度与特征距离, 以二者之积度量特征的重要性[8, 22]; He等人[23]提出基于决策图的无监督特征选择算法DGFS(decision graph based feature selection), 定义特征的局部密度、判别距离和决策图得分, 利用局部密度度量特征代表性, 利用判别距离度量特征之间的冗余性与相似性, 以决策图得分作为评价标准进行特征选择, 决策图得分较高的若干特征构成特征子集; 我们团队提出基于基因密度峰值发现的结肠癌患者诊断基因标志物识别算法[24], 定义基因局部密度和距离, 以密度峰值点基因作为结肠癌患者的识别基因; 鲁棒的无监督特征选择方法RUFS(robust unsupervised feature selection)[25]不同于传统无监督特征选择方法, 通过局部学习正则化的鲁棒非负矩阵分解, 学习样本的伪类簇标签, 在标签学习过程中, 通过鲁棒加入l2, 1范数最小化, 同时完成特征选择.RUFS算法在标签学习和特征学习过程中引入了l2, 1范数, 能够有效地处理异常点和噪音, 并能有效去除冗余和噪音特征, 兼具鲁棒非负矩阵分解、局部学习和鲁棒特征学习的优势.同时, RUFS算法基于有限内存投影, 采用迭代算法解决了算法伸缩性问题; 非负判别特征选择算法NDFS (nonnegative discriminant feature selection)[26]采用谱聚类学习样本类标, 在学习样本类标过程中完成特征选择.类簇标签和特征选择矩阵的联合学习使NDFS算法能够选择最具鉴别性的特征.算法中为了学到更准确的类别标签, 对类指示器添加了非负约束, 为了减少冗余甚至噪声特征, 在目标函数中加入l2, 1范数最小化约束, 保证特征选择矩阵的行稀疏性.算法利用判别信息和特征关联来选择更好的特征子集, 并设计了一种简单、有效的迭代算法来优化目标函数.因为谱聚类的强大优势, 基于谱聚类思想的特征选择方法得到越来越多学者的关注[17, 18, 20, 21, 27, 28].
本文借助谱聚类算法能够发现任意形状类簇, 收敛于全局最优解的性能, 提出基于谱聚类的无监督特征选择思想FSSC(feature selection by spectral clustering).首先对特征进行谱聚类, 使相似(具有强冗余性)的特征聚在同一类簇, 定义特征区分度和独立性, 以特征区分度与独立性之积量化其重要性, 选择各类簇最重要的特征代表该类簇特征, 各类簇的代表特征构成特征子集.利用SC_SD[29](spectral clustering based on standard deviation)、SC_MD[29](spectral clustering based on mean distance)、self-tuning[30]算法进行特征谱聚类, 得到3种不同谱特征选择算法:FSSC-SD(FSSC based on SC_SD)、FSSC-MD(FSSC based on SC_MD)和FSSC-ST(FSSC based on self-tuning).与其他无监督特征选择算法相比, 所提出的算法同时考虑了特征区分度和独立性, 能够选择到代表性强的特征子集.10个癌症基因数据集的实验测试结果表明, FSSC-SD、FSSC-MD和FSSC-ST算法均能选择到具有丰富分类信息的关键基因, 为癌症发病机理、早期诊断、治疗等提供支撑与基础.
2 FSSC无监督特征选择特征选择旨在选择具有高分类信息且相互之间低冗余的特征构成特征子集.本文利用谱聚类算法能够发现任意形状类簇, 收敛于全局最优解的优势, 提出谱聚类特征选择思想FSSC, 以期选择既具有强区分能力, 又彼此之间相互独立的特征构成特征子集.FSSC通过对所有特征进行谱聚类, 将相似(冗余)特征聚到同一类簇, 定义特征标准差为特征区分度, 定义特征与簇内其他区分度更好特征的Pearson相关系数和的倒数为特征独立性, 定义特征重要性为其区分度与独立性之积, 选择各类簇最重要的特征代表该类簇, 所有代表特征构成特征子集.其思想框架如图 1所示.
|
Fig. 1 The frame of proposed FSSC algorithm 图 1 提出的FSSC算法框架 |
2.1 特征谱聚类
聚类根据某种相似性原则将数据对象划分为不同类簇, 簇内对象相似性较高, 但与其他簇对象相似性较低.传统的K-means等聚类算法适合发现球状簇, 无法发现非凸状的簇[31, 32].谱聚类算法以谱图理论为基础, 将样本聚类问题转化为以样本为顶点、样本间相似性为顶点连接边权重的带权无向图的划分问题.谱聚类算法能够发现任意形状的簇, 且收敛于全局最优解[33].因此, 对特征进行谱聚类, 有助于揭示特征之间的内在联系, 发现真正的特征簇.
对特征进行谱聚类, 即以特征为顶点、特征间相似性为顶点连接边权重, 将特征聚类问题转换为特征图划分问题, 分别采用SC_SD[29](spectral clustering based on standard deviation)、SC_MD[29](spectral clustering based on mean distance)、self-tuning[30]算法对特征进行谱聚类, 得到FSSC-SD(FSSC based on SC_SD)、FSSC-MD (FSSC based on SC_MD)和FSSC-ST(FSSC based on self-tuning)这3种谱特征选择算法.其中, self-tuning算法[30]是一种自适应的谱聚类算法, 其对传统谱聚类算法计算亲和矩阵的全局尺度参数σ不能准确体现数据集真实分布信息的缺陷进行了改进, 提出了样本i的局部尺度参数σi, 定义σi为样本i到其第p个近邻的欧氏距离, 采用样本i, j的局部尺度参数σi, σj计算其亲和系数Ai, j.SC_SD和SC_MD谱聚类算法是对self-tuning谱聚类算法的改进[29], 针对self-tuning谱聚类算法的局部尺度参数会受离群点影响的问题, 提出的两种完全自适应的谱聚类算法.SC_SD依据样本i标准差
给定训练数据集
定义1 (特征区分度(feature discernibility)).一个区分能力强的特征对不同类样本的取值往往差异很大, 因此具有较大方差(或标准差), 本文用特征标准差度量特征的类别区分能力.故定义特征fi的区分度disi为其标准差stdi, 如式(1)所示.
| $ di{s_{\rm{i}}}{\rm{ = st}}{{\rm{d}}_i} = \sqrt {\frac{1}{{n - 1}}\sum\limits_{j = 1}^n {{{\left( {{f_{ji}} - \frac{1}{n}\sum\limits_{j = 1}^n {{f_{ji}}} } \right)}^2}} } , {\rm{ i = }}1, 2, ..., d;{\rm{ }}j = 1, 2, ..., n $ | (1) |
fji表示样本j在第i个特征的取值, stdi表示第i个特征的标准差(standard deviation).
定义2 (特征独立性(feature independence)). Pearson相关系数可以度量两变量之间的相关性, 两变量的Pearson相关系数绝对值越小, 则其越不相关.特征选择的目的是选择区分能力强, 且彼此不相关的特征构成特征子集, 剔除不相关和冗余特征.以特征子集中的特征来表达样本, 不仅可以保持和提高系统分类能力, 且能使原系统得到简化.因此, 本文以Pearson相关系数度量特征独立性, 定义特征与同类簇区分能力更强特征的Pearson相关系数绝对值和的倒数为特征独立性.对区分度最大的特征, 定义其独立性为与本簇最不相关特征的Pearson相关性绝对值的倒数.特征独立性定义见式(2), 其中, Mj是特征fi所在的特征类簇.特征fi, fk间的Pearson相关系数定义见式(3).
| $ in{d_i} = \left\{ \begin{array}{l} \frac{1}{{\mathop {\min }\limits_{k \in {M_j}} \left| {{r_{{f_i}, {f_k}}}} \right|}}, \quad {\rm{ }}di{s_i} = \mathop {\max }\limits_{j = 1, ..., d} \{ di{s_j}\} \\ \frac{1}{{\sum\limits_{k:di{s_k} > di{s_i}, k \in {M_j}} {\left| {{r_{{f_i}, {f_k}}}} \right|} }}, {\rm{ otherwise}} \end{array} \right. $ | (2) |
| $ {r_{{f_i}, {f_k}}} = \frac{{\sum\limits_{j = 1}^n {({f_{ji}} - \overline {{f_i}} )({f_{jk}} - \overline {{f_k}} )} }}{{\sqrt {\sum\limits_{j = 1}^n {{{({f_{ji}} - \overline {{f_i}} )}^2}{{({f_{jk}} - \overline {{f_k}} )}^2}} } }}, \quad i, k = 1, 2, ..., d;\quad j = 1, 2, ..., n $ | (3) |
式(2)所示特征独立性定义保障了:若第i特征与区分能力比它强的特征越不相关, 则其独立性越强.区分度最大特征的独立性定义保障了区分能力最强的特征的独立性最强, 这样保障了数据集中区分能力最强的特征一定会被选择到特征子集.
定义3 (特征重要度(feature importance)).特征fi的重要度scorei定义为特征区分度与特征独立性之积, 见式(4), scorei越大, 特征fi越重要.
| $ scor{e_i}{\rm{ = }}di{s_i} \times in{d_i} $ | (4) |
输入:训练数据集
输出:特征子集S.
BEGIN
a) 初始化被选特征子集S=Ø, 全部特征集合为F;
b) 对全部特征分别采用SC_SD、SC_MD、self-tuning算法进行谱聚类, 得到k个特征簇;
c) 利用公式(4)计算各特征的score值, 从各特征簇选取score值最大的特征加入特征子集S;
d) 输出特征子集S.
END
2.4 算法时间复杂度分析本文算法的时间消耗主要在步骤b)的特征谱聚类和步骤c)的基于特征重要度的特征选择.假设训练数据集包含n个样本, 每个样本的维数为d.步骤b)的特征谱聚类的时间复杂度为O(d2).在步骤c)的基于特征重要度的特征选择过程中, 计算特征辨识度的时间复杂度是O(n2d), 计算特征独立性的时间复杂度是O(nd2), 计算特征score值并对其降序排序的时间复杂度是O(dlogd), 由于n < < d, 故步骤c)的时间复杂度为O(d2).因此, 本文基于谱聚类算法的无监督特征选择算法的时间复杂度为O(d2).
3 实验结果与分析实验采用10个常用基因数据集对算法进行测试, 实验使用数据集可从Broad Institute Genome Data Analysis Center(http://portals.broadinstitute.org/cgi-bin/cancer/datasets.cgi)和Gene Expression Model Selector (http://www.gems-system.org/)获取.数据集详细描述见表 1.
| Table 1 The descriptions of datasets used in experiments 表 1 实验数据集描述 |
3.1 实验设计
为了验证所提出的3种谱聚类特征选择算法FSSC-SD、FSSC-MD和FSSC-ST的性能, 实验比较了这3种算法与基于决策图的无监督特征选择算法DGFS(decision graph-based feature selection)[23]、多类簇无监督特征选择算法MCFS(multi-cluster feature selection)[19]、Laplacian分值特征选择算法(Laplacian score for feature selection)[18]、鲁棒的无监督特征选择方法RUFS(robust unsupervised feature selection)[25]以及非负判别特征选择算法NDFS(nonnegative discriminant feature selection)[26]在表 1数据集的实验结果.其中, 在FSSC-ST算法中, 高斯核函数参数设置为经验值7, 对比算法DGFS采用欧式距离计算特征间距离并升序排序, 截断距离dc设置为特征总数2%位置处的距离值; 对比算法Laplacian、RUFS和NDFS采用余弦相似性度量特征相似性, 且近邻数K均设置为5, NDFS算法的正则化参数设置为0.1.
实验采用10折交叉验证方法划分训练集与测试集, 缺失数据采用类内均值填充, 为避免特征间不同量纲对实验结果的影响, 采用最大最小化方法标准化数据, 采用SVM和KNN(K=1)两种分类器, SVM分类器采用林智仁等人开发的SVM工具箱Libsvm[34].其中, 核函数采用线性核函数, 惩罚因子C取20, 其余参数均取默认值.以5次10折交叉验证实验结果的平均值比较各算法的性能, 评价准则采用分类正确率ACC、AUC(或MAUC)、F2[35]、F-measure、Sensitivity和Specificity.其中, F2是针对不平衡数据的评价方法, 可以避免ACC不适合不平衡数据与F-measure主要强调分类器对正类样本识别能力的缺陷[35].MAUC是AUC对多类问题的推广.实验代码使用MATLAB R2017b实现; 实验环境为Win10 64bit操作系统, 8GB内存, Intel(R) Core(TM) i5-6600 CPU @3.30GHz 3.31GHz.
3.2 实验结果比较本节比较提出的FSSC-SD、FSSC-MD、FSSC-ST算法与无监督特征选择算法DGFS、MCFS、Laplacian、RUFS及NDFS在表 1所示数据集选择的基因子集的性能, 比较各算法选择的基因子集对应分类器的各指标值.
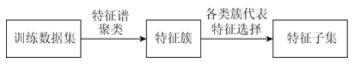
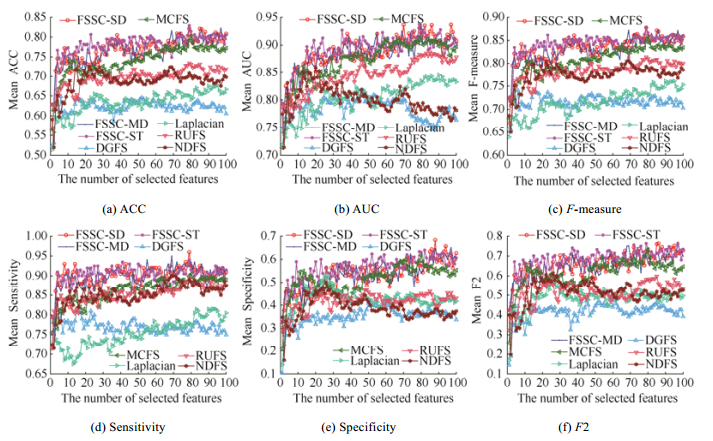
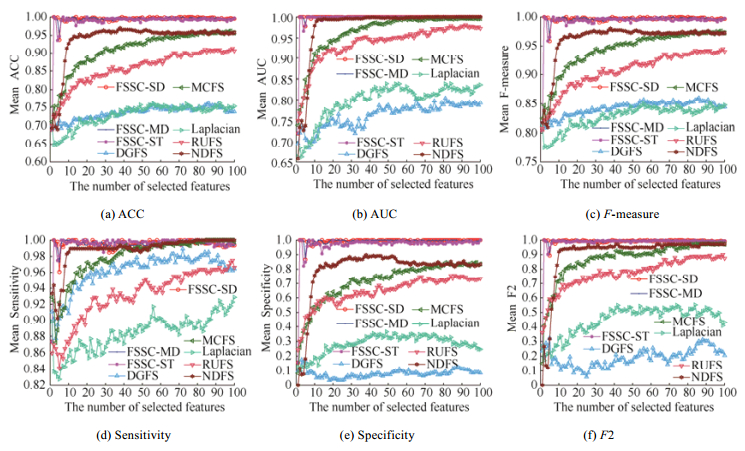
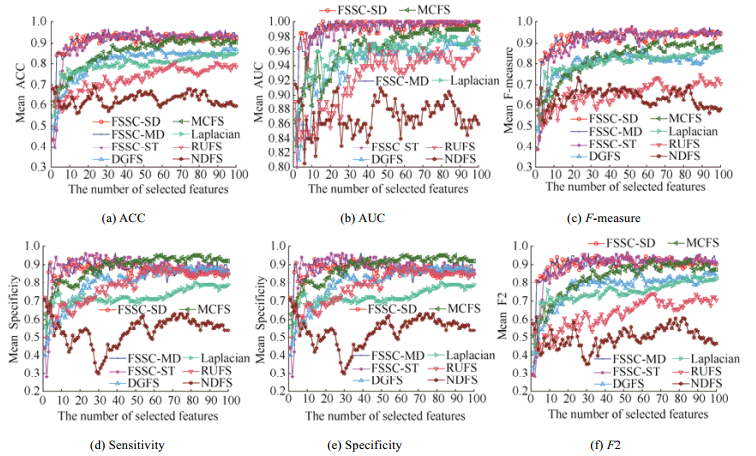
3.2.1 平均实验结果以Colon、Carcinoma、ALL1和DLBCL-Tumor数据集为例, 对比各算法采用KNN的实验结果.图 2~图 5分别是各算法在Colon、ALL1、Carcinoma和DLBCL-Tumor数据集对应不同特征子集的实验结果.
|
Fig. 2 The average indexes of KNN classifier of each algorithm for different feature subsets on Colon dataset 图 2 各算法在Colon数据集对应不同特征子集的KNN分类器平均指标值 |
|
Fig. 3 The average indexes of KNN classifier of each algorithm for different feature subsets on ALL1 dataset 图 3 各算法在ALL1数据集上对应不同特征子集的KNN分类器平均指标值 |
|
Fig. 4 The average indexes of KNN classifier of each algorithm for different feature subsets on Carcinoma dataset 图 4 各算法在Carcinoma数据集上对应不同特征子集的KNN分类器平均指标值 |

|
Fig. 5 The average indexes of KNN classifier of each algorithm for different feature subsets on DLBCL-Tumor dataset 图 5 各算法在DLBCL-Tumor数据集上对应不同特征子集的KNN分类器平均指标值 |
图 2所示实验结果显示, 本文算法FSSC-SD、FSSC-ST和FSSC-MD选择的特征子集的KNN分类器的各指标值绝对地优于对比算法.MCFS、RUFS算法次之, 接着是NDFS算法, DGFS算法与Laplacian算法选择的特征子集的KNN分类器的性能最差.
图 3所示实验结果显示, 本文所提出的FSSC-SD、FSSC-MD、FSSC-ST算法选择的特征子集的分类性能最优, 接着是NDFS算法, MCFS和RUFS算法选择的特征子集的分类性能居中, DGFS算法与Laplacian算法选择的特征子集的分类能力最差.
图 4所示实验结果显示, 本文提出的FSSC-ST、FSSC-SD和FSSC-MD算法选择的基因子集的分类性能优于DGFS、Laplacian、RUFS与NDFS算法选择的基因子集的分类性能.DGFS、MCFS、Laplacian算法选择基因子集的分类性能居中, RUFS和NDFS算法选择的基因子集的各项指标值最低.当选择的特征数较多时, MCFS算法选择的基因子集的KNN分类器的Sensitivity和Specifity指标上略有超过FSSC-ST、FSSC-SD和FSSC-MD算法.因此, Carcinoma数据集的实验结果揭示, 本文所提算法FSSC-SD、FSSC-MD和FSSC-ST均能选择出区分能力好且包含特征数少的特征子集.
图 5所示实验结果显示, 本文提出的FSSC-ST、FSSC-SD和FSSC-MD算法选择的基因子集对应分类器的ACC、AUC、F-measure、Specificity和F2指标非常好, 优于对比算法DGFS、MCFS、Laplacian、RUFS与NDFS.在基因子集规模大于50时, 本文提出的FSSC-ST、FSSC-SD和FSSC-MD算法选择的基因子集的Sensitivity指标优于其他对比算法.因此, 本文提出的FSSC-SD、FSSC-MD和FSSC-ST算法均能选择到区分能力较好的特征子集.
综合图 2~图 5的实验结果来看, 本文提出的FSSC-SD、FSSC-MD、FSSC-ST算法均能选择出类别区分能力很好的特征子集, 优于其他对比算法.
3.2.2 各算法最优值比较为了验证本文算法的整体性能, 比较各算法在10个癌症基因数据5次10折交叉验证选择的特征子集对应分类器的各指标平均结果的最优值, 采用各算法在10个数据集实验结果的win/draw/loss来评价其性能.表 2、表 3分别展示了各算法在10个数据集上所选特征子集对应KNN、SVM分类器的最优平均分类准确率ACC、AUC、F-measure、Sensitivity、Specificity和F2值的win/draw/loss比较.表 4给出了各算法在表 1的10个数据集选择的基因子集的KNN和SVM分类器各指标值的最优值平均的win/draw/loss结果比较.表中加粗和下划线表示本文提出的FSSC-SD、FSSC-MD和FSSC-ST算法优于其他对比算法的结果.表中加粗加红和下划线表示本文提出的FSSC-SD、FSSC-MD和FSSC-ST谱特征选择算法之间比较, win大于loss的结果.
| Table 2 The highest average index comparison of KNN of selected gene subsets on 10 datasets (win/draw/loss) 表 2 各算法在10个数据集所选基因子集的KNN分类器的最高平均分类性能指标比较(win/draw/loss) |
| Table 3 The highest average index comparison of SVM of selected gene subsets on 10 datasets (win/draw/loss) 表 3 各算法在10个数据集所选基因子集的SVM分类器的最高平均分类性能指标比较(win/draw/loss) |
| Table 4 The mean highest average index of KNN and SVM for the selected gene subsets on 10 datasets (win/draw/loss) 表 4 各算法在10个数据集所选基因子集的SVM与KNN分类器的最高平均分类指标的均值比较(win/draw/loss) |
表 2所示实验结果揭示, 本文提出的FSSC-SD、FSSC-MD和FSSC-ST谱特征选择算法选择的基因子集的KNN分类器的分类性能绝对地优于对比算法DGFS、MCFS、Laplacian、RUFS和NDFS选择的基因子集的分类能力.所提出的FSSC-SD、FSSC-MD和FSSC-ST算法相比, FSSC-SD算法选择的基因子集的分类性能最优, 在ACC、AUC、Sensitivity和Specificity这4个指标上优于FSSC-ST算法, 在ACC、F-measure、Sensitivity和Specificity这4个指标上优于FSSC-MD算法.所提出的FSSC-MD与FSSC-ST算法选择的基因子集的分类能力相当.在F2指标上, 提出的3种谱特征选择算法FSSC-SD、FSSC-MD和FSSC-ST选择的基因子集的分类能力相当, FSSC-MD略优于FSSC-SD和FSSC-ST算法.
表 3关于各算法选择的基因子集的SVM分类器的各项最优平均值比较显示:本文提出的FSSC-ST、FSSC-MD和FSSC-ST算法绝对地优于对比算法DGFS、MCFS、Laplacian、RUFS和NDFS.所提出的FSSC-SD、FSSC-MD和FSSC-ST这3种特征选择算法选择的基因子集对应SVM分类器的平均AUC最高值比较显示, FSSC-SD选择的基因子集的性能最好, 其次是FSSC-MD算法, FSSC-ST位居第三.所提出的3种谱特征选择算法选择的基因子集对应SVM分类器的ACC比较显示, FSSC-ST选择的基因子集的分类性能最好, 然后依次是FSSC-MD和FSSC-SD.F-measure指标比较显示, FSSC-MD算法的性能略优于FSSC-SD和FSSC-ST算法. Sensitivity比较显示, 提出的3种谱特征选择算法FSSC_SD、FSSC-MD与FSSC-ST选择的基因子集的分类性能基本相当, FSSC-SD略优于FSSC-MD和FSSC-ST, FSSC-MD略优于FSSC-ST.Specificity最高均值比较显示, 提出的FSSC-ST与FSSC-MD算法略优于提出的FSSC-SD算法, FSSC-ST与FSSC-MD性能相当.各算法的基因子集对应SVM分类器的F2值显示, FSSC-MD算法最优, 然后依次是FSSC-SD算法和FSSC-ST算法.
由表 3所示各算法选择的基因子集对应SVM分类器的实验结果分析得出:本文提出的3种无监督特征选择算法FSSC-SD、FSSC-MD和FSSC-ST绝对地优于对比算法DGFS、MCFS、Laplacian、RUFS和NDFS.在所提出的3种算法之间, 多数情况下的性能指标值比较基本持平, 没有一种算法绝对地优于其他两种算法.
表 4给出关于提出的FSSC-SD、FSSC-MD和FSSC-ST谱特征选择算法在表 1给出的10个基因数据集选择的基因子集对应KNN和SVM分类器相应最优指标的均值比较显示:所提出的3种谱特征选择算法所选基因子集的分类性能绝对地优于对比算法DGFS、MCFS、Laplacian、RUFS和NDFS.所提出的算法FSSC-SD、FSSC-MD与FSSC-ST相比, FSSC-SD算法选择的基因子集的分类能力在多数情况下优于所提出的FSSC-MD和FSSC-ST算法, FSSC-MD在AUC和F2指标绝对地优于FSSC-ST算法, FSSC-ST只在ACC和F2指标略优于FSSC-SD或FSSC-MD.
因此, 综合表 2~表 4的实验结果可以得出:所提出的FSSC-SD、FSSC-MD和FSSC-ST谱特征选择算法绝对地优于对比算法DGFS、MCFS、Laplacian、RUFS和NDFS.提出的3种谱特征选择算法彼此相比, FSSC-SD选择的特征子集的分类能力最强, 其次是FSSC-MD和FSSC-ST算法.
3.2.3 特征子集规模比较前两小节展示了所提出的3种无监督特征选择算法选择的基因子集的分类性能比较, 本小节将比较各算法选择的特征子集的规模, 即选择的基因数.图 6展示了各算法选择的基因子集对应KNN和SVM分类器的最优指标值对应基因子集包含的平均基因数.图中颜色越偏于ColorBar底部颜色, 表示基因数越少(特征子集规模越小), 反之, 颜色越偏于ColorBar顶部的颜色, 特征子集包含的基因数就越多.
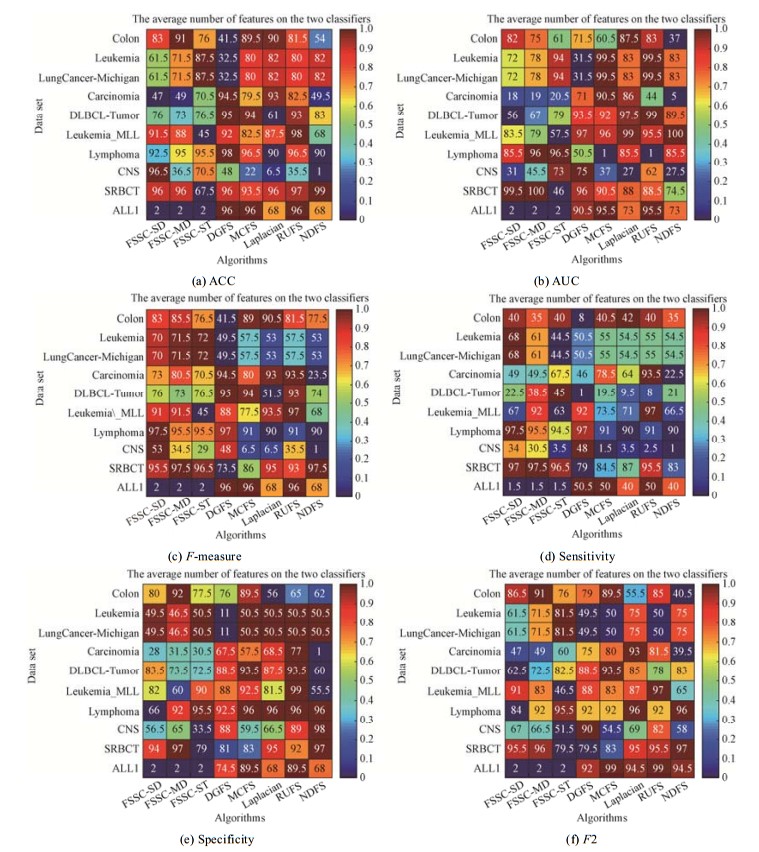
|
Fig. 6 The mean feature number of selected gene subsets with the best average index of SVM and KNN by each algorithm for each dataset 图 6 各算法对各数据集选择的基因子集对应SVM和KNN分类器的最优指标值对应特征子集的平均特征数 |
图 6所示特征子集规模比较显示, 本文提出的FSSC-SD、FSSC-MD和FSSC-ST算法无论采用哪种评价指标, 均能发现在ALL1数据集的最具分类能力且规模最小的基因子集.除了F-measure指标, 在ACC、AUC、F2、Sensitivity和Specificity指标上, 所提出的FSSC-SD、FSSC-MD和FSSC-ST算法在各数据集均能选择到规模相对较小且具有很好分类能力的基因子集.分析其原因是, F-measure指标过分强调了对于正类的识别能力, 而忽略了选择的基因子集对于负类的识别能力.
综合对图 6所示各算法选择的特征子集规模的分析可知, 所提出的3种谱特征选择算法FSSC-SD、FSSC- MD和FSSC-ST能够选择到分类性能好且规模不大的特征子集.
4 各算法统计重要度分析为了检验本文提出的FSSC-SD、FSSC-MD和FSSC-ST算法与对比算法DGFS[23]、MCFS[19]、Laplacian[18]、RUFS[25]和NDFS[26]是否具有统计显著性, 本节采用Friedman检验方法来检验各特征选择算法间的差异[36, 37], 在用Friedman检测到算法间的显著性差异后, 采用多重比较检验(multiple comparison test)作为事后检验, 以发现各对特征选择算法之间的显著性差异.我们依据各算法在10个癌症基因数据5次10折交叉验证选择的特征子集对应KNN分类器的ACC、AUC、F-measure、Sensitivity、Specificity和F2各指标平均结果的最优值, 在α=0.05时, 进行Friedman检测.
6种指标下的Friedman检测显示各特征选择算法之间存在显著差异.基于KNN分类器的预测准确率ACC的χ2=60.9034, df=7, p=9.9624e-11;AUC的χ2=59.4230, df=7, p=1.9679e-10;F-measure的χ2=58.7260, df=7, p=2.7102e-10; Sensitivity的χ2=44.7006, df=7, p=1.5633e-07;Specificity的χ2=23.7982, df=7, p=0.0012;F2的χ2=62.1587, df=7, p=5.5873e-11. p < 0.05对所有评价指标均成立, 因此得出结论:各算法间存在显著差异.
表 5~表 10展示了可信水平为0.95时, 每一对特征选择算法进行多重比较检验的结果.各表上三角表示各对算法间的平均等级差, 下三角表示各算法对之间的统计重要性, 以*表示相应算法之间统计重要性显著.
| Table 5 Paired rank comparison of 8 feature selection algorithms in ACC of KNN predictive model 表 5 8种特征选择算法依据特征子集对应KNN预测模型的ACC等级比较 |
| Table 6 Paired rank comparison of 8 feature selection algorithms in AUC of KNN predictive model 表 6 8种特征选择算法依据对应特征子集的KNN预测模型的AUC等级比较 |
| Table 7 Paired rank comparison of 8 feature selection algorithms in F-measure of KNN predictive model 表 7 8种特征选择算法依据对应特征子集的KNN预测模型的F-measure等级比较 |
| Table 8 Paired rank comparison of 8 feature selection algorithms in Sensitivity of KNN predictive model 表 8 8种特征选择算法依据对应特征子集的KNN预测模型的Sensitivity等级比较 |
| Table 9 Paired rank comparison of 8 feature selection algorithms in Specificity of KNN predictive model 表 9 8种特征选择算法依据对应特征子集的KNN预测模型的Specificity等级比较 |
| Table 10 Paired rank comparison of 8 feature selection algorithms in F2 of KNN predictive model 表 10 8种特征选择算法依据对应特征子集的KNN预测模型的F2等级比较 |
表 5~表 10所示的统计重要性检测结果显示:本文提出的FSSC-ST算法与所有对比特征选择算法DGFS、MCFS、Laplacian、RUFS和NDFS之间均存在显著性差异.对于所提出的FSSC-SD和FSSC-MD算法, 当使用所选基因子集对应KNN分类器的ACC、AUC、F-measure、Sensitivity和F2指标时, 与所有对比特征选择算法DGFS、MCFS、Laplacian、RUFS和NDFS之间均存在显著不同; 当使用Specificity指标时, 与MCFS算法没有显著区别, 但与其他对比算法DGFS、Laplacian、RUFS和NDFS均存在显著性不同.算法MCFS与算法Laplacian、RUFS和NDFS在所有指标下均存在统计显著性不同.对比算法NDFS和RUFS在除了Sensitivity之外的其他指标上均存在显著性不同.
表 10所示统计分析结果显示:当使用F2指标时, 本文提出的3种谱特征选择算法FSSC-SD、FSSC-MD和FSSC-ST不仅与所有对比算法DGFS、MCFS、Laplacian、RUFS和NDFS存在显著性不同, 所提出的FSSC-SD和FSSC-MD算法之间也存在统计显著性差异.另外, Laplacian和RUFS、NDFS算法之间也存在显著性不同.
由以上统计重要性分析可见, 所提出的谱特征选择算法FSSC-SD、FSSC-MD和FSSC-ST是非常有效的基因选择算法, 能够选择到分类性能显著不同于对比算法的基因子集.
5 结论针对癌症基因数据的特征选择问题, 提出基于谱聚类的无监督特征选择算法FSSC-SD、FSSC-MD和FSSC- ST对所有特征进行谱聚类, 将相似性较高(强冗余性)的特征聚成一类, 从各类簇选择代表特征构成特征子集.提出特征区分度、特征独立性、特征重要度概念, 定义特征区分度为其标准差, 定义特征独立性为其与簇内其他区分度更高特征的Pearson相关系数和的倒数, 对区分度最大特征, 定义其独立性为其与所在特征簇最不相关特征的Pearson相关系数绝对值的倒数, 定义特征重要性为其区分度与独立性之积.10个基因数据集的实验测试结果及各算法的统计显著性检测结果表明, 所提出的无监督谱特征选择算法FSSC-SD、FSSC-MD和FSSC-ST均能选择到不仅具有强分类能力, 且包含基因数较少的特征子集, 其中, FSSC-SD算法选择的特征子集的分类能力最优.所提出的谱特征选择算法FSSC-SD、FSSC-MD和FSSC-ST与对比算法DGFS、MCFS、Laplacian、RUFS、NDFS之间存在显著性差异.
| [1] |
Derisi JL, Iyer VR, Brown PO. Exploring the metabolic and genetic control of gene expression on a genomic scale. Science, 1997, 278(5338): 680-686.
http://d.old.wanfangdata.com.cn/NSTLQK/10.1126-science.278.5338.680/ |
| [2] |
Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, Coller H, Loh ML, Downing JR. Molecular classification of cancer:Class discovery and class prediction by gene expression monitoring. Science, 1999, 286(5439): 531-537.
http://cn.bing.com/academic/profile?id=c0d52c8c3b13bd6328654196652ae883&encoded=0&v=paper_preview&mkt=zh-cn |
| [3] |
Khan J, Wei JS, Ringner M, Saal LH, Ladanyi M, Westermann F, Berthold F, Schwab M, Antonescu CR, Peterson C. Classification and diagnostic prediction of cancers using gene expression profiling and artificial neural networks. Nature Medicine, 2001, 7(6): 673.
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=f8409acf442f1f6f80f5c64e5bec786e |
| [4] |
Li YX, Li JG, Ruan XG. Study of informative gene selection for tissue classification based on tumor gene expression profiles. Chinese Journal of Computers, 2006, 29(2): 324-330(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/jsjxb200602018 |
| [5] |
Guyon I, Elisseeff A. An introduction to variable and feature selection. Journal of Machine Learning Research, 2003, 3: 1157-1182.
http://d.old.wanfangdata.com.cn/NSTLQK/10.1162-153244303322753616/ |
| [6] |
Ding C, Peng HC. Minimum redundancy feature selection from microarray gene expression data. Journal of Bioinformatics and Computational Biology, 2005, 3(2): 185-205.
http://d.old.wanfangdata.com.cn/NSTLQK/NSTL_QKJJ022822192/ |
| [7] |
Xie JY, Wang MZ, Hu QF. The differentially expressed gene selection algorithms for unbalanced gene datasets by maximize the area under ROC. Journal of Shaanxi Normal University (Natural Science Edition), 2017, 45(1): 13-22(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/sxsfdxxb201701003 |
| [8] |
Xie JY, Wang MZ, Zhou Y, Li, JY. Coordinating discernibility and independence scores of variables in a 2D space for efficient and accurate feature selection. In: Huang DS, Han K, Hussain A, eds. Proc. of the Int'l Conf. on Intelligent Computing 2016, Part III, Intelligent Computing Methodologies. LNAI 9773, Springer Int'l Publishing Switzerland, 2016. 116-127.
|
| [9] |
Blum AL, Langley P. Selection of relevant features and examples in machine learning. Artificial Intelligence, 1997, 97(1-2): 245-271.
http://d.old.wanfangdata.com.cn/NSTLQK/10.1016-S0004-3702(97)00063-5/ |
| [10] |
Kohavi R, John GH. Wrappers for feature subset selection. Artificial Intelligence, 1997, 97(1-2): 273-324.
http://d.old.wanfangdata.com.cn/OAPaper/oai_pubmedcentral.nih.gov_311150 |
| [11] |
Lal TN, Chapelle O, Weston J, Elisseeff A. Embedded methods. In: Guyon I, Nikravesh M, Gunn S, Zadeh LA, eds. Proc. of the Feature Extraction, Foundation and Applications, Studies in Fuzziness and Soft Computing, 207. Berlin, Heidelberg: Springer-Verlag, 2006. 137-165.
|
| [12] |
Kira K, Rendell LA. The feature selection problem: Traditional methods and a new algorithm. In: Kerber R, ed. Proc. of the 10th National Conf. on Artificial Intelligence. AAAI Press, 1992. 129-134.
|
| [13] |
Peng HC, Long FH, Ding C. Feature selection based on mutual information: Criteria of max-dependency, max-relevance, and min-redundancy. IEEE Trans. on Pattern Analysis & Machine Intelligence, 2005, (8): 1226-1238.
|
| [14] |
Hall MA. Correlation-based feature selection for machine learning[Ph.D. Thesis]. Hamilton: University of Waikato, 1999.
|
| [15] |
Dash M, Liu H, Yao J. Dimensionality reduction of unsupervised data. In: Storms P, ed. Proc. of the 9th IEEE Int'l Conf. on Tools with Artificial Intelligence. IEEE, 1997. 532-539.
|
| [16] |
Xu JL, Zhou YM, Chen L, Xu BW. An unsupervised feature selection approach based on mutual information. Journal of Computer Research and Development, 2012, 49(2): 372-382(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/jsjyjyfz201202017 |
| [17] |
Zhang L, Sun G, Guo J. Unsupervised feature selection method based on K-means clustering. Application Research of Computers, 2005, 22(3): 22-24(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/jsjyyyj200503006 |
| [18] |
He XF, Cai DY, Niyogi P. Laplacian score for feature selection. In: Weiss Y, Sch lkopf B, Platt J, eds. Advances in Neural Information Processing Systems (NIPS18). Cambridge: MIT Press, 2006. 507-514.
|
| [19] |
Cai D, Zhang CY, He XF. Unsupervised feature selection for multi-cluster data. In: Wallace BC, Small K, Brodley CE, Trikalinos TA, eds. Proc. of the 16th ACM SIGKDD Int'l Conf. on Knowledge Discovery and Data Mining. Washington: ACM, 2010. 333-342.
|
| [20] |
Wang LX, Jiang SY. Novel feature selection method based on feature clustering. Application Research of Computers, 2015, 32(5): 1305-1308(in Chinese with English abstract).
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=jsjyyyj201505006 |
| [21] |
Zheng Z, Liu H. Spectral feature selection for supervised and unsupervised learning. In: Ghahramani Z, ed. Proc. of the 24th Int'l Conf. on Machine Learning (ICML 2007). New York: ACM, 2007. 1151-1157.
|
| [22] |
Xie JY, Qu YN, Wang MZ. Unsupervised feature selection algorithms based on density peaks. Journal of Nanjing University (Natural Sciences)., 2016, 52(4): 735-745(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/njdxxb201604018 |
| [23] |
He JR, Bi YZ, Ding LX, Li ZK, Wang SW. Unsupervised feature selection based on decision graph. Neural Computing and Applications, 2017, 28(10): 3047-3059.
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=abfb72650bc41f796290fc94cc340870 |
| [24] |
Xie JY, Fan W. Gene markers identification algorithm for detecting colon cancer patients. Pattern Recognition and Artificial Intelligence, 2017, 52(4): 1019-1029(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/mssbyrgzn201711008 |
| [25] |
Qian MJ, Zhai CX. Robust unsupervised feature selection. In: Rossi F, ed. Proc. of the 23rd Int'l Joint Conf. on Artificial Intelligence (IJCAI 2013). AAAI Press, 2013. 1621-1627.
|
| [26] |
Li ZC, Yang Y, Liu J, Zhou XF, Lu HQ. Unsupervised feature selection using nonnegative spectral analysis. In: Hoffmann J, Selman B, eds. Proc. of the 26th AAAI Conf. on Artificial Intelligence (AAAI-12). Toronto: AAAI Press, 2012. 1026-1032.
|
| [27] |
Hu MJ, Zheng LP, Tang L, Yang H, Fu W. Feature selection algorithm based on joint spectral clustering and neighborhood mutual information. Pattern Recognition and Artificial Intelligence, 2017, 30(12): 1121-1129(in Chinese with English abstract). http://www.cnki.com.cn/Article/CJFDTotal-MSSB201712009.htm
|
| [28] |
Jiang SY, Zheng Q, Zhang QS. Clustering-based feature selection. Acta Electronica Sinica, 2008, 36(s1): 157-160(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/dianzixb2008z1028 |
| [29] |
Xie JY, Ding LJ. The true self-adaptive spectral clustering algorithms. Acta Electronica Sinica, 2019, 47(5): 1000-1008(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/dianzixb201905004 |
| [30] |
Zelnik-Manor L, Perona P. Self-tuning spectral clustering. In: Saul LK, Weiss Y, Bottou L, eds. Advances in Neural Information Processing Systems (NIPS17). Cambridge MA: MIT Press, 2005. 1601-1608.
|
| [31] |
Wang L, Bo LF, Jiao LC. Density-sensitive semi-supervised spectral clustering. Ruan Jian Xue Bao/Journal of Software, 2007, 18(10): 2412-2422(in Chinese with English abstract).
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=20071005&journal_id=jos [doi:10.1360/jos182412] |
| [32] |
Xie JY, Zhou Y. A new criterion for clustering algorithm. Journal of Shaanxi Normal University (Natural Science Edition), 2015, 43(6): 1-8(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/sxsfdxxb201506002 |
| [33] |
Luxburg UV. A tutorial on spectral clustering. Statistics and Computing, 2007, 17(4): 395-416.
http://d.old.wanfangdata.com.cn/NSTLQK/10.1007-s11222-007-9033-z/ |
| [34] |
Chang CC, Lin CJ. LIBSVM:A library for support vector machines. ACM Trans. on Intelligent Systems and Technology (TIST), 2011, 2(3): 27.
http://d.old.wanfangdata.com.cn/Periodical/jdq201315008 |
| [35] |
Xie JY, Wang MZ, Zhou Y, Gao HC, Xu SQ. Differentially expressed gene selection algorithms for unbalanced gene datasets. Chinese Journal of Computers, 2019, 42(6): 1232-1251(in Chinese with English abstract).
http://d.old.wanfangdata.com.cn/Periodical/jsjxb201906006 |
| [36] |
Borg A, Lavesson N, Boeva V. Comparison of clustering approaches for gene expression data. In: Jaeger M, Nielsen TD, Viappiani P, eds. Proc. of the 12th Scandinavian Conf. on Artificial Intelligence. Aalborg: IOS Press, 2013. 55-64.
|
| [37] |
Xie JY, Gao HC, Xie WX, Liu XH, Grant PW. Robust clustering by detecting density peaks and assigning points based on fuzzy weighted K-nearest neighbors. Information Sciences, 2016, 354: 19-40.
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=9cc8de09be4dbbac0918ed9121a329d0 |
| [4] |
李颖新, 李建更, 阮晓钢. 肿瘤基因表达谱分类特征基因选取问题及分析方法研究. 计算机学报, 2006, 29(2): 324-330.
http://d.old.wanfangdata.com.cn/Periodical/jsjxb200602018 |
| [7] |
谢娟英, 王明钊, 胡秋锋. 最大化ROC曲线下面积的不平衡基因数据集差异表达基因选择算法. 陕西师范大学学报(自然科学版), 2017, 45(1): 13-22.
http://d.old.wanfangdata.com.cn/Periodical/sxsfdxxb201701003 |
| [16] |
徐峻岭, 周毓明, 陈林, 徐宝文. 基于互信息的无监督特征选择. 计算机研究与发展, 2012, 49(2): 372-382.
http://d.old.wanfangdata.com.cn/Periodical/jsjyjyfz201202017 |
| [17] |
张莉, 孙钢, 郭军. 基于K-均值聚类的无监督的特征选择方法. 计算机应用研究, 2005, 22(3): 22-24.
http://d.old.wanfangdata.com.cn/Periodical/jsjyyyj200503006 |
| [20] |
王连喜, 蒋盛益. 一种基于特征聚类的特征选择方法. 计算机应用研究, 2015, 32(5): 1305-1308.
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=jsjyyyj201505006 |
| [22] |
谢娟英, 屈亚楠, 王明钊. 基于密度峰值的无监督特征选择算法. 南京大学学报(自然科学), 2016, 52(4): 735-745.
http://d.old.wanfangdata.com.cn/Periodical/njdxxb201604018 |
| [24] |
谢娟英, 樊雯. 结肠癌患者诊断的基因标志物识别算法. 模式识别与人工智能, 2017, 52(4): 1019-1029.
http://d.old.wanfangdata.com.cn/Periodical/mssbyrgzn201711008 |
| [27] |
胡敏杰, 郑荔平, 唐莉, 杨红, 郑荔平, 傅为.联合谱聚类与邻域互信息的特征选择算法.模式识别与人工智能, 2017, 30(12): 1121-1129. http://www.cnki.com.cn/Article/CJFDTotal-MSSB201712009.htm
|
| [28] |
蒋盛益, 郑琪, 张倩生. 基于聚类的特征选择方法. 电子学报, 2008, 36(s1): 157-160.
http://d.old.wanfangdata.com.cn/Periodical/dianzixb2008z1028 |
| [29] |
谢娟英, 丁丽娟. 完全自适应的谱聚类算法. 电子学报, 2019, 47(5): 1000-1008.
http://d.old.wanfangdata.com.cn/Periodical/dianzixb201905004 |
| [31] |
王玲, 薄列峰, 焦李成. 密度敏感的半监督谱聚类. 软件学报, 2007, 18(10): 2412-2422.
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=20071005&journal_id=jos [doi:10.1360/jos182412] |
| [32] |
谢娟英, 周颖. 一种新聚类评价指标. 陕西师范大学学报(自然科学版), 2015, 43(6): 1-8.
http://d.old.wanfangdata.com.cn/Periodical/sxsfdxxb201506002 |
| [35] |
谢娟英, 王明钊, 周颖, 高红超, 许升全. 非平衡基因数据的差异表达基因选择算法研究. 计算机学报, 2019, 42(6): 1232-1251.
http://d.old.wanfangdata.com.cn/Periodical/jsjxb201906006 |
 2020, Vol. 31
2020, Vol. 31


