在生物体内, 基因通过调控相互作用实现它们的生物学功能, 并完成复杂的生命活动.基因之间的调控关系可分为两类:激励与抑制.当一个基因的表达增强致使另一个基因的表达增强时, 称前者对后者存在激励关系; 反之, 当一个基因的表达增强致使另一个基因的表达减弱时, 称为抑制关系.将这种调控关系以图的形式呈现, 就是基因调控网.在基因调控网中, 将每个基因视作一个节点, 具有调控关系的两节点间存在有向边, 由调控基因指向被调控基因.有向边的符号代表了调控关系的类型.将基因调控网在某个时刻的采样称作基因调控网在该时刻的快照.将若干个在时间上具有先后关系, 能够反映基因调控网在一段时间内的动态演化过程的快照集合称作动态基因调控网.动态基因调控网的网络演化, 就是有向边随时间变化而形成、消亡或转变方向的过程.对动态基因调控网网络演化的研究有许多重要意义, 例如, 可以对未来的基因调控关系进行预测, 从而预测并探明癌症等疾病的发病机制; 为疾病的诊断和治疗提供依据; 并在基因靶向药物的开发和测试领域进行仿真实验.当前, 对动态基因调控网的研究包括两个方面:其一是研究如何根据某时刻的基因表达数据推断该时刻的基因调控网快照[1−4]; 其二是研究在已知基因调控网的部分拓扑结构信息的情况下, 如何准确预测基因调控网未知部分或未来时刻的连边[5, 6].对于前者, 近年来已经有许多较为成熟的工具和方法出现, 如TRACE[7, 8]、GENIE3[3]等, 借助这些工具和方法, 可以准确地将输入的基因表达数据映射为基因调控网.但是在获得了动态基因调控网后, 还无法应用于实际工作.只有进一步研究其网络演化机制, 才能准确地预测基因调控网未来的连边, 从而应用于医学和药学研究等领域.
当前, 对基因调控网网络演化的研究仍有一些不足:(1)大部分的研究对象是静态无符号网络[9, 10], 但基因调控网的演化模式并非一成不变.研究带符号的动态基因调控网的网络演化有更为重要的意义; (2)学术界对基因调控网的演化规律和机制虽有一些猜想[11−14], 但目前尚未有公认的、合理的解释, 人们对于基因调控网的网络演化的认知仍然存在一些不足.针对上述缺陷, 考虑到基因调控网与社会网络在拓扑结构特征上有一定的相似性[15], 本文试图借鉴较为成熟的社会网络研究技术对动态基因调控网网络演化展开研究, 以揭示动态基因调控网网络演化的秘密.
在社会网络研究中, 有许多关于网络演化和链路预测的方法被提出.传统的链路预测方法主要分为3类:基于相似性的链路预测、基于最大似然估计的链路预测与概率模型方法.
● 基于相似性的链路预测方法衡量两个节点之间的相似性, 并据此估算两节点之间产生连边的可能性.基于节点相似性的链路预测算法包括共同邻居算法(CN)[16]、AA算法[17]、RA[18]等.类似地, 还有基于路径的相似性算法, 如LP[19]、Katz[20]等, 它们相对于之前的算法考虑了二阶乃至更高阶的间接共同邻居.有最新的研究[21]考虑到节点的差异性, 将上述多种相似性指标综合地应用于链路预测, 在实验中取得了更好的表现.
● 第2类是基于最大似然估计的链路预测方法.通过似然估计值和马尔可夫-蒙特卡洛算法, 可以得到两节点之间产生连边的概率, 最大似然估计方法在面对有明显层次结构的复杂网络时有较好的效果.
● 概率模型方法的基本思想是建立一个具有多参数的概率模型, 通过调节参数, 使模型能够再现该网络的真实连边关系.基于这类思想的经典算法有马尔可夫网络模型(RMN)、朴素贝叶斯[22]等.
以上的传统链路预测方法都是根据网络的某些局部或全局的某些拓扑结构特征来进行预测.如果某种网络的某项特征比较突出, 则可能有较好的预测效果.
基因调控网与社会网络具有某些相似的拓扑结构特征, 如它们都呈现出了无标度网络和小世界网络的特性.这些相似的特征表明将社会网络研究方法应用于基因调控网在一定程度上是可行的.但上述的传统方法的研究对象局限于静态网络, 无法将其直接应用于本文所研究的动态基因调控网.一些较新的方法弥补了这个缺陷, 如Li等人提出的基于深度学习的动态社会网络链路预测模型ctRBM[23], 它考虑了节点自身的历史连接情况和邻居节点对其连边产生的影响; Zhu等人使用基于隐空间的时序链路预测方法[24]将所有节点映射到一个高维空间中, 并认为距离较近的节点更有可能产生边.有研究[25, 26]表明, 链路预测可以反映网络演化机制, 两者在分析网络演化上具有内在的一致性.这些方法将基于相似性的链路预测方法扩展到了动态社会网络上, 在网络演化分析上取得了良好的效果.
综合考虑了现有方法的各种优点和不足, 本文最终将目光放在一种被称为“模体”的网络子结构上.模体是一种特殊的网络子图, 在复杂网络研究中引起了广泛的关注.有研究[27, 28]表明, 在基因调控网中, 模体所占的比例远高于其他类型的子图, 且模体与基因功能、网络演化都有着密切的关系.从模体的角度开展研究, 既能适应基因调控网的动态性, 也能帮助我们寻找其与社会网络演化的相似特征.另一些社会网络的研究者通过迁移学习进行链路预测[29, 30].考虑到动态社会网络与基因调控网本质上的不同, 动态基因调控网的网络演化研究应当具有较强的针对性, 迁移学习为这种特异性的网络演化模型的构建提供了借鉴.本文同时将迁移学习的思路应用于带符号基因调控网的符号判别中, 将动态基因调控网网络演化的研究扩展到有向带符号网络领域.
基于以上认识, 本文提出一种两阶段动态基因调控网网络演化分析方法(dynamic gene regulatory network evolution analyzing method, 简称DGNE):第1阶段将深入研究模体的转换规律, 以模体演化的视角预测动态基因调控网未来时刻的快照; 第2阶段在一阶段的基础上采用基于隐空间特征的符号判别方法对快照中有向边的符号进行判别.最后得到带符号基因调控网在未来时刻的网络快照, 为本文提出的DGNE方法的正确性和有效性提供校验.本文的工作和贡献可总结如下.
(1) 提出一种基于模体转换概率的动态基因调控网连边预测算法(link prediction algorithm based on motif transfer probability, 简称MT), 弥补了以往研究中只考虑静态网络未考虑动态网络的缺陷.该算法将基因调控网网络演化的研究由静态网络扩展到动态网络范围, 能够更加准确地把握网络演化模式, 提高网络连边预测的准确性.
(2) 在考虑动态基因调控网有向边符号信息的情况下, 提出一种基于隐空间特征的符号判别算法, 对有向边进行符号判别.该算法弥补了以往研究只考虑无符号网络未考虑带符号网络的缺陷.将基因调控网网络演化的研究扩展到带符号网络领域, 使网络演化模型更贴近现实, 有利于研究成果在生物医学和药学中的应用.
(3) 本文首次从模体演化的视角考察了基因调控网的演化.模体作为重要的功能和结构单位, 其演化对于网络整体演化有着不可忽视的作用.对模体演化意义的挖掘, 为本方法提供了良好的可解释性.从模体的角度研究基因调控网, 为今后生物信息学和生物医学的研究提供了一种新的观点.
本文第1节描述动态基因调控网的网络演化问题, 包括相关概念的定义、背景知识的简要介绍, 以及对所研究问题的建模.第2节介绍MT算法和基于隐空间特征的符号判别算法.第3节在大量数据集上进行实验, 包括算法内相关模型的选取和参数检验, 以及对本文提出的方法进行有效性验证和健壮性测试等.
1 问题描述本节对动态基因调控网及网络演化的相关概念进行描述, 包含了对基因调控网、模体、隐空间的定义和介绍.然后对动态基因调控网网络演化问题进行形式化描述.
1.1 相关定义定义1(基因调控网). 基因调控网是由基因表达数据经过推断生成的、用来描述基因间调控关系的带符号的有向图.其生成过程如图 1所示.
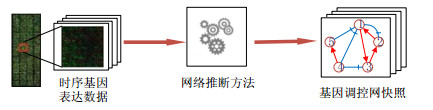
|
Fig. 1 Diagram of inferring gene regulatory network from temporal gene expression data 图 1 从时序基因表达数据推断得到基因调控网方法示意图 |
基因表达数据是一个l×m的矩阵, 表示l个基因在m个不同时刻上的表达强度高低.通过该矩阵, 可以计算基因间表达强度变化的相关性:若为正相关, 即一个基因的表达强度的提高导致另一个基因表达强度提高, 称这种调控关系为激励关系; 反之为抑制关系.将这种基因调控关系映射为复杂网络中节点和边的关系, 就是基因调控网的基本形式.
定义2(模体).模体是网络中的一种特殊的子图结构, 它在网络所有子图中占较大比例.在基因调控网中, 某些模体已被确定具有生物学意义[28].由于模体种类和结构复杂多样, 本文无法完全覆盖, 因此, 本文研究的模体仅限于由3个节点构成的子图.在有向网络中, 3个节点按照两两之间的连边状况, 总共存在64种可能的连边情况.为方便描述, 将这64种模体分别编号并枚举, 如图 2所示.在网络中, 任意3个节点都可以映射成一个模体.

|
Fig. 2 Diagram of enumeration of motifs of gene regulatory network 图 2 基因调控网模体枚举示意图 |
定义3(隐空间). 隐空间是一个人为设定的高维空间.将网络中的一条有向边映射到这个空间后, 得到一个用来表示其拓扑结构的特征向量, 称为隐空间特征.将源网络中的有向边映射到隐空间后, 就可以通过机器学习方法进行目标网络的符号判别.其映射和符号判别过程如图 3所示.

|
|
1.2 基于模体的基因调控网问题建模
本文采用复杂网络的方法来描述基因调控网, 基因调控的复杂网络定义如下.
一个有向图G(V, E), 其中, V是所有节点的集合, E⊆V×V是所有边的集合.节点的数量|V|被称为图的规模.在有向图中, 一条边是以二元组(u, v)的形式定义的(其中, (u, v)∈E), 由节点u指向v.本文通过如下形式将基因调控网映射到一个复杂网络上:将每一个基因当作点集中的一个点, 两点之间的有向边表示两基因之间存在调控关系, 从调控基因指向被调控基因.本文使用邻接矩阵对基因调控网进行描述, 当|V|=M时, Π是该基因调控网的邻接矩阵, Π∈{−1, 0, +1}M×M.对矩阵中的元素Π(u, v)做以下规定:Π(u, v)=0当且仅当基因u不存在对基因v的调控关系, Π(u, v)=1当且仅当基因u对基因v存在激励的调控关系, Π(u, v)=−1当且仅当基因u对基因v存在抑制的调控关系.这样, 基因调控网就以复杂网络的形式被表示出来了.
动态基因调控网是由若干个以一定时间按序排列的基因调控网快照组成的.将各快照中的所有节点3个一组进行排列组合, 即得到该快照中的所有模体.网络中的每一个模体都有且仅有一个代表其类型的编号.网络演化的过程可以看作是模体转换的过程.从微观来看, 一个模体在下一时刻要么保持原有类型不变, 要么转换成另一种类型的模体.本文通过描述统计64类模体的相互转换及概率, 从而描述网络的演化过程.我们可以对比从一种模体到另一种模体的转换过程中, 哪些有向边产生、消失或方向改变, 从而预测基因调控网中对应改模体的子图在下一时刻的结构.上述过程如图 4所示.
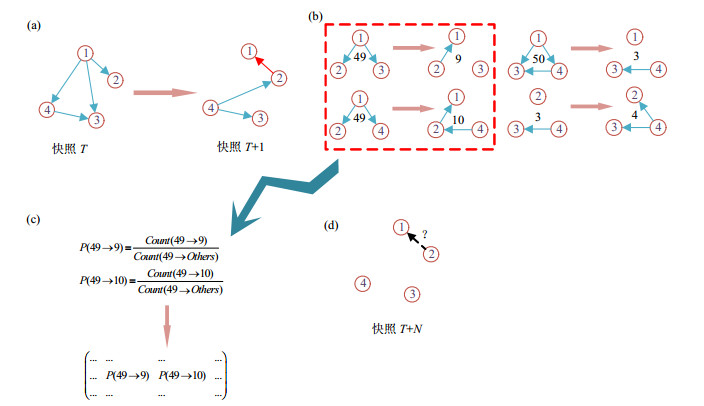
|
(a) 某子图从快照T到快照T+1 的演化,其中,节点 1 和节点 2 之间的有向边发生了方向反转. (b) 从快照T到快照T+1 共涉及到了 4 个模体的转换,其中,与节点 1 和节点 2 之间连边变化相关的 两个模体转换被标记出来,用作下一步的模体转换概率统计. (c) 统计这两个模体的转换概率.Count(i→j)表示快照T中第i种三元组转换成第j种三元组的计数. (d) 利用模体转换概率预测未来快照的连边情况(如在快照T+N中,节点 1 和节点 2 间是否存在连边). Fig. 4 Diagram of modelling of gene regulation network based on motifs 图 4 基于模体的基因调控网建模示意图 |
综上所述, 本文所研究的问题可以描述为:对一个动态基因调控网络G=(G1, G2, …, GT)的两两相邻快照间对应节点组成的模体的变化趋势进行分析, 结合隐空间特征中蕴含的有向边符号信息, 最终揭示基因调控网络的结构随时间变化的演化规律, 得到未来时刻的基因调控网快照.
2 DGNE方法模体是基因调控网的功能单位.虽然当前基因调控网模体的结构与其生物学功能尚未一一对应, 但至少可以知道, 在生物体内中, 生命活动与基因调控网的模体演化是存在相互作用关系的.众所周知, 生物的成长过程具有一定的共性和规律, 因此可以推断基因调控网模体演化也是具有共性和规律的, 进而可以得出整个网络的演化模式.本节将描述这种以模体演化为中心的DGNE方法.
2.1 DGNE方法框架本文提出的DGNE方法以模体演化为中心.如图 5所示, 对于一个输入的动态基因调控网, 首先将其映射为模体, 对相邻快照间的模体变化进行统计分析, 同时结合其他网络拓扑结构特征, 最终得到基因调控网未来时刻的网络结构.DGNE方法由两种算法构成, 分别是基于模体转换概率的动态基因调控网连边预测算法MT(link prediction algorithm based on motif transfer probability)和基于隐空间特征向量的符号判别算法(将在第2.2节和第2.3节详细描述):MT算法将动态基因调控网进行模体演化分析, 得出未来时刻基因调控网的快照; 基于隐空间特征的符号判别算法进一步为未来时刻的基因调控网的快照的边进行符号判别.
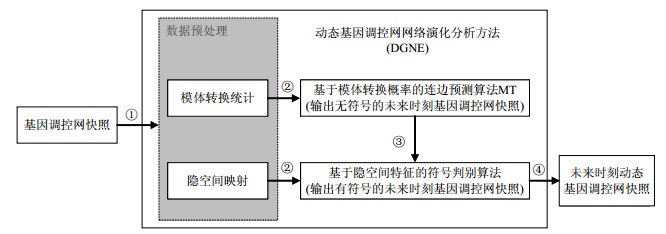
|
Fig. 5 Framework of DGNE 图 5 DGNE框架 |
2.2 MT算法
由于受到不同生命周期的发展特征、生长环境以及各种内外部因素的影响, 模体之间相互转换的概率随时间变化是非线性关系, 不同的模体在基因调控不同阶段转换到其他模体的概率是不同的, 不能简单地采用线性模型来估计未来时刻的模体转换概率.本节提出一种基于模体转换概率和张量分解的算法, 即MT算法进行时间序列预测.
本文将相邻两个快照间模体转换概率用一个64×64的矩阵来表示, 称为模体转换概率矩阵, 记作TCM.矩阵中元素的值TCMt(i, j)=P(trit[i]→trit[j])表示从t时刻到t+1时刻编号为i的模体转换为编号为j的模体的概率, 其中, trit[i]表示t时刻编号为i的模体.构建一个模体转移概率张量(TCT)来表示模体转换的时间序列, 若一个动态基因调控网中有T个快照, 则TCT=(TCM1, TCM2, …, TCMT−1), 张量中的元素TCT(i, j, T)=TCMT(i, j).通过MATLAB的tensor toolbox[31]工具箱中的cp_nmu函数对TCT张量进行非负CP分解, 得到参数λ和3个因式矩阵A, B, C, 其中, ABT表达了不同类型模体间的转换关系; 而C则包含了该关系在时间维度上的信息, 称为时间因式矩阵.使用恰当的模型对时间因式矩阵C的前T−1行进行时间序列预测(见第3.2.1节), 得到矩阵C的第T行元素, 从而预测得到T时刻~T+1时刻的一种可能的模体转换概率矩阵, 称为模体转换似然矩阵(TCLM), 如公式(1)所示.
| $TCLM(i,j) = \sum\limits_{r = 1}^R {{\lambda _r}A(i,r) \cdot B(j,r) \cdot C(T,r)} $ | (1) |
将TCLM按行归一化, 便得到了T到T+1时刻的模体转换概率矩阵TCMT.获得了TCMT, 便可以对T+1时刻的链路状况进行预测.连边预测的目的就是给未来时刻的基因调控网快照的每一个节点对(u, v)赋予一个分数score(u, v), 该分数越高, 意味着该节点对之间存在边的可能性越大.特别地, 由于本文所研究的基因调控网是有向网络, 所以对一个节点对的两种可能的边的方向——即score(u, v)和score(v, u)分别赋分.从上述描述可知, 所有包含了u和v两节点的模体的转换都可以为下一时刻该节点对之间存在边的可能性产生影响, 但显而易见的是, 不同模体的影响力不是一样的.越有影响力的模体, 在连边预测中所占的比重越大.本文将模体的“影响力”定义为两个方面:一是历史快照内该模体中的连边形成频率, 二是历史快照内该模体形成闭合的频率.总的来说, 该模体越稠密, 则说明其内部节点关系越紧密, 在链路预测中发挥的作用相对于其他稀疏模体更重要.除此之外, 对节点对之间形成连边的概率还有一条假设, 即某条历史连边产生的时刻距离待预测时刻越近, 预测结果中这条边仍然存在的可能性越高.
由以上描述, 本文对模体在链路预测中的影响力定义如公式(2)所示.
| ${W_i} = \sum\limits_{t = 1}^{T - 1} {\theta _1^{T - t}g(i,t)} + \sum\limits_{t = 1}^{T - 1} {\theta _2^{T - t}f(i,t)} + 1$ | (2) |
Wi是表示第i个模体的影响力的分值; g(i, t)表示t时刻第i个模体中各节点连边个数; f(i, t)表示t时刻第i个模体是否闭合, 即3个节点两两之间是否至少存在一条有向边, 闭合时为1, 不闭合为0;θ1, θ2是控制不同时期的历史快照对Wi贡献的系数, 距当前时刻越近, 贡献越大.
由此, 根据T到T+1时刻的模体转换概率矩阵TCMT和模体的影响力W, 可以为每一对节点对赋予一个得分, 表示T时刻每个边存在的概率高低, 如公式(3)所示.
| $score(u,v) = \sum\limits_{m = 1}^{|tri(u,v)|} {{W_m} \cdot T(m)} $ | (3) |
其中, |tri(u, v)|表示T−1时刻包含节点对u, v的模体的总数, T(m)表示包含了边(u, v)的第m个模体从T时刻到T+1时刻的转移概率.具体算法如算法1所示.
算法1. MT.
输入:时刻1到时刻2, 时刻2到时刻3, …, 时刻T−2到时刻T-1的模体转换概率矩阵TCM1, TCM2, …, TCMT−1, 张量分解参数K.
输出:时刻T+1的基因调控网快照.
1.构造张量TCT=(TCM1, TCM2, …, TCMT−1)对张量TCT进行CP分解, 获得系数λ和大小为(T−2)×K的矩阵A, B, C.
2.根据公式(3)计算模体转换似然概率矩阵TCLM.
3.根据公式(4)计算模体重要性得分.
4.据公式(5)得到分数矩阵score.
5.取score矩阵中分数最高的前L个元素对应的节点对作为预测的连边(L表示每个快照中包含的边的平均数量), 得到时刻T+1的连边邻接矩阵.
2.3 基于隐空间特征的动态基因调控网符号判别算法在第2.2节研究的基础上, 将动态基因调控网分为源网络和目标网络, 其中, 前T−1时刻的所有快照集合为源网络, 作为该算法的训练集, 记作Gs=(G1, G2, …, GT−1); 由MT算法得到T时刻的快照为目标网络, 记作Gt=(GT).在已经获得关于未来时刻基因调控网已知存在的边的情况后, 对已知存在的边的符号仍所知极少, 因此本文借鉴了迁移学习的思路, 寻找源网络和目标网络拓扑结构上共有的特征空间[30], 并通过机器学习的方法进行符号判别.对于动态基因调控网来说, 各快照都是同一网络在不同演化阶段的不同形态, 因此各快照之间必定存在一些内在的联系.本节通过寻找这种共有的特征空间, 以提取其显式特征和隐空间特征, 并构造一个高效的分类器, 在目标网络上对其边的符号进行判别.
与其他机器学习方法不同的是, 在基因调控网编的符号判别中, 没有任何“先验”的特征向量可以来对训练集中一条边的符号来进行描述.因此, 需要自己来根据源网络和目标网络的拓扑结构进行特征空间的构造.本文构造的特征分为两类:(1)显式特征, 用以表达实例中显而易见的属性; (2)隐空间特征, 不能直接由网络拓扑结构看出, 但也表达了源网络和目标网络之间所共有的一些模式.
2.3.1 显式特征对一个有向边(u, v), 本文为其定义的显式特征包括节点的度数、中介中心性、模体个数以及共同邻居等.这里需要注意的是, 在为每一个样本定义这些特征的时候, 完全不考虑这条边的符号, 因为在目标网络中, 对于绝大部分边的符号都是未知的.各个特征的描述具体如下.
(1) 节点的度数.对一个有向边(u, v), 通过degout(u)和degin(v)来分别指代节点u的出度和节点v的入度.节点的度数代表着它与图中其他节点连接的紧密性.
(2) 中介中心性.对一个有向边(u, v), 采用两端点的中介中心性fbc(u)和fbc(v)作为它的两个特征.中介中心性代表着一个点在图中作为中心节点的地位.
(3) 模体个数.对一个有向边(u, v), 考虑将包含了(u, v)的模体个数作为其特征.设该模体的第3个节点为w.若该模体中存在有向边(u, w), 则称此边为前向边(F), 若存在有向边(w, u)则称此边为后向边(B), 或者w和u之间没有边存在, 则记为N.w和另一个节点v的关系同理.这样, 对于一条边(u, v)有8个特征, 分别为fFF, fFB, fBF, fBB, fFN, fNF, fBN, fNB.
(4) 共同邻居.对一个有向边(u, v), 如果存在另一个节点w, 使得w与u和v之间均有边相连, 则w为u, v的共同邻居.fcn(u, v)指有向边(u, v)的两端点的共同邻居的个数.
以上的显式特征是非常直观的, 但基因调控网的网络演化规律十分复杂, 仅使用以上特征无法很好地对基因调控网的符号进行准确的判别.为了更好地利用源网络中已知符号的边所蕴含的信息, 本文还构造了隐空间特征以捕捉蕴藏在拓扑结构之下的源网络和目标网络之间共有的模式.
2.3.2 隐空间特征提取通过MT算法, 可以得到源网络Gs以及目标网络Gt的邻接矩阵As和At.使用非负矩阵三因子分解将这两组邻接矩阵在同一特征空间中进行因式矩阵分解, 得到源网络和目标网络中边的隐空间特征.
非负矩阵三因子分解是非负矩阵分解的衍生, 它的非负性和对稀疏矩阵的控制, 可以有效地刻画数据中潜藏的局部属性, 并进行细粒度的特征提取.传统的非负矩阵分解用于将一个矩阵分解为两个非负矩阵的乘积, 其可以描述如下:给定矩阵
| $\left. \begin{gathered} \min J = {A_s} - ||{U_s}{\mathit{\Sigma} _k}V_s^T||_F^2 + {A_t} - ||{U_t}{\mathit{\Sigma} _k}V_t^T||_F^2 + \alpha ||{\mathit{\Sigma} _k}||_F^2 \\ {\rm{s}}{\rm{.t}}{\rm{. }}\sum\limits_{j = 1}^k {{U_{s( \cdot j)}}} = 1,\sum\limits_{j = 1}^k {{V_{s( \cdot j)}}} = 1,\sum\limits_{j = 1}^k {{U_{t( \cdot j)}}} = 1,\sum\limits_{j = 1}^k {{V_{t( \cdot j)}}} = 1 \\ {U_s},{V_s},{U_t},{V_t} \in R_ + ^{M \times k} \\ \end{gathered} \right\}$ | (4) |
||·||F为弗罗贝尼乌斯范数, M为基因调控网规模.公式(4)的目标是寻找合适的矩阵分解, 使
本文使用一种迭代更新的算法来求解上式.首先将上式改写成公式(5), 便于用代码描述的形式.
| $\mathcal{J} = tr(A_s^T{A_s} - 2A_s^T{U_s}{\mathit{\Sigma} _k}V_s^T + {V_s}\mathit{\Sigma} _k^TU_s^T{U_s}{\mathit{\Sigma} _k}V_s^T) + tr(A_t^T{A_t} - 2A_t^T{U_t}{\mathit{\Sigma} _k}V_t^T + {V_t}\mathit{\Sigma} _k^TU_t^T{U_t}{\mathit{\Sigma} _k}V_t^T) + \alpha tr(\mathit{\Sigma} _k^T{\mathit{\Sigma} _k})$ | (5) |
其中, tr(·)是指矩阵的迹.以Us为例介绍求解上式的方法.由于约束条件中包含Us≥0, 可以使用拉格朗日乘子法解决此问题.本文引入拉格朗日乘子
基于上式和文献[33]的方法, 本文按公式(6)所示规则迭代更新Us.
| ${U_{s(i,j)}} \leftarrow {U_{s(i,j)}}\sqrt {\frac{{{{({A_s}{V_s}\mathit{\Sigma} _k^T)}_{(i,j)}}}}{{{{({U_s}{\mathit{\Sigma} _k}V_s^T{V_s}\mathit{\Sigma} _k^T)}_{(i,j)}}}}} $ | (6) |
同理可得Vs, Ut, Vt和Σk的迭代规则如公式(7)~公式(10)所示,
| ${V_{s(i,j)}} \leftarrow {V_{s(i,j)}}\sqrt {\frac{{{{(A_s^T{U_s}{\mathit{\Sigma} _k})}_{(i,j)}}}}{{{{({V_s}\mathit{\Sigma} _k^TU_s^T{U_s}{\mathit{\Sigma} _k})}_{(i,j)}}}}} $ | (7) |
| ${U_{t(i,j)}} \leftarrow {U_{t(i,j)}}\sqrt {\frac{{{{({A_t}{V_t}\mathit{\Sigma} _k^T)}_{(i,j)}}}}{{{{({U_t}{\mathit{\Sigma} _k}V_t^T{V_t}\mathit{\Sigma} _k^T)}_{(i,j)}}}}} $ | (8) |
| ${V_{t(i,j)}} \leftarrow {V_{t(i,j)}}\sqrt {\frac{{{{(A_t^T{U_t}{\mathit{\Sigma} _k})}_{(i,j)}}}}{{{{({V_t}\mathit{\Sigma} _k^TU_t^T{U_t}{\mathit{\Sigma} _k})}_{(i,j)}}}}} $ | (9) |
| ${\mathit{\Sigma} _{k(i,j)}} \leftarrow {\mathit{\Sigma} _{k(i,j)}}\sqrt {\frac{{{{(U_s^T{A_s}{V_s} + U_t^T{A_t}{V_t})}_{(i,j)}}}}{{{{(U_s^T{U_s}{\mathit{\Sigma} _k}V_s^T{V_s} + U_t^T{U_t}{\mathit{\Sigma} _k}V_t^T{V_t} + \alpha {\mathit{\Sigma} _k})}_{(i,j)}}}}} $ | (10) |
通过以上算法, 可以在若干次迭代后, 获得使
以上基于隐空间特征的动态基因调控网符号判别算法伪码如算法2所示.
算法2.基于隐空间特征的动态基因调控网符号判别算法.
输入:邻接矩阵As和At、显式特征矩阵(记作Ks和Kt)、参数k.
输出:带符号的T时刻基因调控网邻接矩阵.
1.初始化隐空间特征矩阵Us, Vs, Ut, Vt和Σk.
2. WHILE根据公式(8)极值J收敛.
3.根据公式(9)更新Us.
4.根据公式(10)更新Vs.
5.根据公式(11)更新Ut.
6.根据公式(12)更新Vt.
7.根据公式(13)更新Σk.
END WHILE
8.将Us, Vs, Ks按行拼接作为特征矩阵, 使用LibSVM工具[34]对As上的边的符号进行学习, 得到分类模型.
9.将模型应用到At上, 得到符号判别的结果.
3 实验为了验证本文提出的DGNE方法在基因调控网的演化预测分析上的有效性, 本节设计了一系列实验对本文提出的DGNE方法进行分析和描述.本节首先设计了模型的选取和参数检验实验, 在仿真数据集上测试, 并获得了能使本文提出的DGNE方法取得最优效果的算法参数的取值; 然后对DGNE的时间复杂度进行了分析; 最后, 本节设计了针对DGNE方法有效性的验证实验, 在真实数据集上和对比算法进行比较, 验证了该方法在动态基因调控网网络演化分析中的有效性.
3.1 数据集描述本文使用如下数据集对算法进行实验验证.
(1) 基因调控网仿真数据3组(以下分别记作SynA、SynB、SynC).本文使用GeneNetWeaver 3.1工具[35]自带的Ecoli数据集, 分别生成包括300个、600个和900个基因, 以及20个快照的动态基因表达数据, 每个快照的时间间隔为1 000s.然后使用TRaCE程序[7, 8], 将各快照的基因表达数据作为输入, 得到各个快照的基因调控网的邻接矩阵.该数据为通过仿真软件得到仿真实验数据, 在软件中, 我们可以方便地对数据中的噪声和产生的环境进行控制, 不同的规模可以用来验证算法的普适性.
(2) 果蝇基因调控网数据(以下记作Dro)是一组无符号动态基因调控网数据集, 由Song等人[36]在文献中直接以邻接矩阵的形式提供.该数据集包括果蝇在其不同的生命周期中基因调控网拓扑结构.Dro直观且真实地展示了低等生物在正常生理周期中的基因调控网的自然演化情况, 在科研中非常具有现实意义.
(3) 小鼠烟雾暴露基因调控网数据(以下记作Rat).原始基因表达数据来自Stevenson等人的研究[37].为研究长期处于吸烟环境对机体的影响, 原作者将小鼠完全暴露于吸烟环境中, 并每隔一段时间采集小鼠的基因表达数据.该数据集包括跨度238天、共12次采样的基因表达数据, 采集时间间隔不固定, 为2天~70天.在对原始数据进行log2归一化后, 本文使用DeltaNet程序[38]将原始数据中的12个时间点的基因表达数据分别作为输入, 得到对应的12个基因调控网网络快照.该数据集展示了较高等的哺乳动物在人为干预的非自然状态下的基因调控网的网络演化过程, 与Dro数据集形成对照.
上述实验数据集的网络拓扑信息见表 1.其中, Dro数据集是无符号基因调控网, 网络数据中不包含激励边和抑制边信息.与其他类型的复杂网络相比, 基因调控网非常稀疏.对比几个基因调控网的网络拓扑结构, 可以发现它们具有一定的共性.例如, 节点和边的数量大约在1:1.5~1:3左右、相对其他类型的复杂网络非常稀疏.此外, 由于基因调控网中每个节点对应着一个实际存在的基因, 因此这类网络规模较为有限, 局限在几百或几千个节点之内.从带符号基因调控网激励边和抑制边分布上看, 两种类型的有向边各占一半, 这有利于保证在符号判别过程中学习样本数量的平衡性.
| Table 1 Network topology information of the datasets 表 1 各数据集网络拓扑结构信息表 |
3.2 模型的选取及参数检验 3.2.1 时间因式矩阵预测模型和参数选取
在MT算法里, 要获得准确的从T−1时刻到T时刻模体转换似然矩阵, 就要使用合适的模型和参数对时间因式矩阵C进行时序分析.由T−1个模体转换概率矩阵组成的张量TCT, 其因式分解后得到的时间因式矩阵C∈R(T−1)×k, 其每行分别存储着TCT在时间维度上的隐含信息.时序分析的本质就是选取合适的分布模型, 根据不同快照距离待预测网络时间的远近, 为每一行其分配合适的权重, 得到C(T).根据常识, 距离待预测网络越近的快照, 其对于链路预测的作用就越大, 应该被赋予更高的权重.其网络节点的度分布都大致遵循指数分布或类似分布.公式(11)和公式(12)分别为在两种分布模型下C(T)的计算方式:
| $C(T,r) = \sum\limits_{t = 1}^{T - 1} {{{(T - t)}^a}C(t,r)} $ | (11) |
| $C(T,r) = \sum\limits_{t = 1}^{T - 1} {{a^{T - t}}C(t,r)} $ | (12) |
其中, a是未知参数.本文在synA, synC数据集上分别对两种模型和其参数进行检验, 寻求可以取得最佳效果的模型和参数, 其结果如图 6所示.

|
Fig. 6 ROC curves under different models and parameters 图 6 各模型及参数下的ROC曲线 |
当ROC曲线越向左上角凸出时, 表明在该参数的取值下连边预测算法具有更好的预测效果.从图 6的实验结果可以得到如下结论.
(1) 当采用幂律分布时, v取值为−4或−5时效果最佳; 当采用指数分布模型时, v取值为0.2时可以取得最好的结果.而且无论是哪种取值, 其ROC曲线大致相仿, 考虑到两种分布模型计算复杂度相似, 所以可任选其中一种作为后续实验中该算法的参数.
(2) 在SynA数据集中无论取那种参数和模型, ROC曲线的形状差别非常小.SynA数据集中网络规模只有300个节点, 这说明MT算法在小规模网络上参数不敏感, 在一定程度上不适用于这类网络.
MT算法参数检验为探究MT算法中张量分解的维度K对算法的表现是否具有影响, 本文在SynA、SynB、SynC数据集将不同值赋予K并运行算法, 得到T时刻的网络连边矩阵, 并根据AUC和准确率来进行评价.结果如图 7所示.
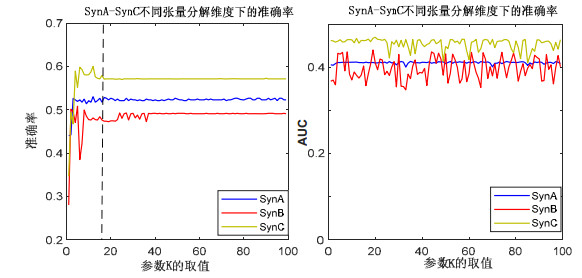
|
Fig. 7 Influence of the dimension K of the tensor decomposition on algorithm MT 图 7 张量分解维度K对MT算法表现的影响 |
从图 7的实验结果可以得到如下结论.
(1) 在synA、synB、synC数据集上, MT算法的准确率一开始随着张量分解维度的增加而增加, 但随着张量分解的维度到达16之后, 准确率便不再升高, 而是开始小幅波动直至最终收敛.因此, 实验选定张量分解维度为16为宜, 过低则算法表现较差, 过高则增加无意义的计算开销.
(2) MT算法的AUC值随张量分解的维度增加无明显变化趋势, 只是一直围绕着固定值做小于±0.1的波动.因此可得出, 张量分解的维度仅对算法的准确率有影响, 而对衡量算法表现的另一指标AUC无明显影响.
3.2.2 基于隐空间特征的动态基因调控网符号判别算法本文提出的DGNE方法中的基于隐空间特征的符号判别算法是以隐空间特征和显示特征共同作为符号判别的特征向量, 对基因调控网中边的符号进行学习.为探究在特征向量中两类特征的比例对算法效果的影响, 即隐空间特征向量的维度对算法的影响, 需要对此算法中非负矩阵三因子的分解维度k进行参数检验, 以确定最佳取值.如图 8展现了在不同维度分解下符号判别的准确率的变化.

|
Fig. 8 Influence of the dimension k of the non-negative tri-matrix factorization 图 8 非负矩阵三因子分解维度k对算法表现的影响 |
从图 8中的实验结果可以得到以下结论.
(1) 随着非负矩阵三因子分解维度k的增加, 开始时算法的准确率会上升, 在k为10左右的时候达到最高值; 但之后随着k的增加, 准确率会小幅下降; 当k大于20后趋于收敛.
(2) 算法准确率随k增加达到最大值的速度在synA、synB、synC中依次递减.这可能与数据集规模有关, 随着基因调控网数据集规模的增大, 在该算法中应选用的k值也要逐渐增加, 以取得更好的效果.鉴于此, 表 2给出了数据集规模与参数k的建议取值.
| Table 2 Recommended values of parameter k in symbol discrimination algorithm based onlatent space character under different scales of gene regulatory network 表 2 基因调控网规模与基于隐空间特征的符号判别算法中参数k的建议取值 |
3.3 时间复杂度分析
本文提出的算法可以分为3个部分:一是相邻模体间转换概率统计, 构建模体转移概率张量; 二是以张量分解为基础, 对下一时刻的基因调控网快照进行连边预测; 三是进行显式特征和隐空间特征的提取, 以每条有向边的符号为标签进行学习和分类.其中, 第3部分是可以和第1、第2部分很大程度上并行完成的.将一个快照中所有模体遍历的时间复杂度是O(n3), n是基因调控网的规模, 即节点数.那么, 若一个动态基因调控网包含T个快照, 则总的时间复杂度是O(Tn3).由于获得的张量大小是64×64×(T−1), 由第3.1节数据集描述可知, 现有的动态基因调控网快照数都不大, 当T是一个不大于100的数时, 其张量分解和时间因式矩阵预测的时间复杂度都可以视作常量O(K)(无论是采用幂律分布模型还是指数分布模型), K是张量分解的维度.进行连边预测时, 需要对每一个节点对进行打分, 时间复杂度是O(n2).特征提取是针对每一条边进行的提取, 所以第3部分的时间复杂度是O(k|E|), 其中, k是非负矩阵三因子分解的分解维度, |E|是涉及到的的边的总数.通常来说, 基因调控网是及其稀疏的, 即|E|≈ξn, ξ是一个1.5~3左右的数.总的来说, DGNE方法的瓶颈在于第1部分, 算法的最大时间复杂度为O(n3).
3.4 DGNE方法有效性验证本节对DGNE方法的相关表现与目前已有的基础和最新方法进行对比分析, 评价其在动态基因网网络演化分析方面的表现.鉴于目前已有的相关方法无法对网络符号进行判别, 因此本节首先验证在不进行符号判别的情况下算法的表现(此时该方法退化为MT算法), 再验证加入了符号判别算法之后DGNE方法的表现.
本节使用若干对比性算法, 包括4种基准算法与2种最新算法与本方法进行对比, 它们是:
(1) 共同邻居(CN)[16]:该方法假设在网络中, 两个节点之间如果有更多的共同邻居, 则它们在下一个时刻的快照中更倾向于连边.
(2) Adamic-Adar(AA)[17]:是共同邻居算法的改良版.该方法假设两个节点如果都与一个度比较小的节点相连, 那么这两个节点在下一时刻的快照中连边的概率更大.即, 度小的共同邻居节点的贡献要大于度大的共同邻居节点.
(3) Katz[20]:是一种基于网络全局拓扑结构的链路预测算法.该方法遍历两节点间的所有路径, 并假设若这些路径中距离短的数量越多, 那么这两个节点在下一时刻快照中连边的概率更大.
(4) Preferential Attachment(PA)[39]:是一种基于网络局部拓扑结构的链路预测算法.在该方法中, 两节点在下一时刻快照中相连的概率正比于两节点各自的度的乘积.
(5) ctBRM[23]:是一个基于受限玻尔兹曼机的深度学习方法, 面对噪声有较好的稳定性.
(6) BCGD[24]:是一个基于隐空间的动态社会网络链路预测算法.该方法假设所有的节点都存在于某个不可观测的隐空间中, 在该空间中, 距离较近的节点对更容易形成连边.
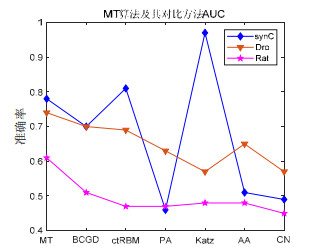
3.4.1 不进行符号判别的情况下DGNE方法的有效性验证在不进行符号判别的情况下, 本文的DGNE方法退化为MT算法.由于3个仿真数据集仅载数据规模有所差别, 故选取其中最具有代表性的SynC数据集与Dro和Rat分别对DGNE的有效性进行验证.实验结果AUC值见表 3, 各算法准确率如图 9所示.当AUC大于0.5时, 其分数越高, 表示算法在此数据集上拥有越高的预测准确性; 当AUC值小于等于0.5时, 表示该算法在该数据集上的预测准确性约等于随机效果, 或不如随机过程.
| Table 3 AUC of MT and its contrastive algorithms 表 3 MT算法及其对比算法AUC |
|
Fig. 9 Precision of MT and its contrastive algorithms 图 9 MT算法及其对比算法的准确率 |
(1) 总体来说, MT算法效果优于其他算法.这说明MT算法能够正确地预测动态基因调控网的连边状况, 即, 其在一定程度上对动态基因调控网网络演化机制具有正确的把握.其中, Katz算法在synC数据集上具有非常良好的表现, 但在真实的数据集中其表现并不如在仿真数据集中那么优秀.这是由于本文使用的仿真工具的数据生成模型过于强调了基因调控网的小世界特性, 而小世界特性与Katz算法的特点相性很好, 导致结果出现异常.在以后的研究中, 应适当调整仿真工具的模型和参数.
(2) 大部分基准算法的AUC指标表明, 它们的预测效果与随机过程几乎无异.该结果说明, 这些算法不适用于对动态基因调控网进行演化分析.动态基因调控网网络演化具有其独特的特性, 从结果上来看, 只有本文提出的DGNE方法以及BCGD在一定程度上可以把握其演化模式.
(3) 本文提出的MT算法与大部分最新算法在真实数据集上(Dro, Rat)的表现不如仿真数据集上好, 这是由于在真实的生物体内, 基因调控网的演化还受到众多外部因素的干扰, 如疾病、环境、食物和药物等.小鼠作为哺乳动物其基因调控网受到的各类外部影响更甚于果蝇, 因此, 单纯地用模体演化解释这种情况下的动态基因调控网网络演化是不足的, 但即使如此, 它也比其他现有算法在AUC和准确率指标上有更好的表现.
3.4.2 进行符号判别的情况下DGNE方法的有效性验证在考虑了符号判别的情况下, 为验证算法在仿真与真实基因调控网上的性能表现, 本文在synC、Dro和Rat数据集上进行各算法的比较.在考虑连边符号的状况下, 不仅要考虑连边的有无, 还要考虑连边的符号正负, 因此不再适用于AUC这一评价指标.图 10是各算法的准确率的对比, 其中, 为了验证本文提出的符号判别算法中显式特征和隐空间特征各自对算法表现的贡献, 我们用DGNE-E指代在符号判别算法中只使用显式特征, DGNE-L指代在符号判别算法中只使用隐空间特征.需要强调的是, 在本实验中, 只有本文提出的DGNE方法是与带符号的测试集进行对比, 其他算法在进行准确率计算时, 均忽略了测试集的符号信息(上述对比算法不提供符号判别功能).

|
Fig. 10 Precision of DGNE and its contrastive algorithms 图 10 DGNE及其对比算法的准确率 |
从图 10的实验结果中可以得到以下结论.
(1) 与图 9对比可以看出, 在引入了符号判别之后, 本文提出的DGNE方法的在各个数据集上的准确率略低于其他对比算法.这是因为在该实验中, 只有DGNE方法进行了符号判别, 而其他算法并没有(也并不能做到符号判别).相对于其他方法“一阶段”地对无符号网络的连边预测, 本文创新地提出“两阶段”的适用于带符号网络的DNGE方法对动态基因王进行连边预测, 虽然为算法的整体性能引入了额外的噪声与误差, 但这种框架与网络模型更加贴近真实的动态基因调控网, 从而能够在实际的生物医学的应用中发挥更大的作用.
(2) 显式特征与隐空间特征的结合, 能够使符号判别取得更好的效果, 从而提升算法的整体性能.相对于与仅仅使用显式特征进行符号判别, 本文提出的将两类特征结合的DGNE方法能够提升5%~10%的准确率; 隐空间特征对符号判别效果的贡献大于显式特征, 相对于仅使用隐空间特征进行符号判别, DGNE方法也有约3%~5%的提升.这说明在使用机器学习方法对带符号网络的符号进行判别的时候, 特征的构造的选取十分重要.
4 总结和展望本文提出的DGNE方法对动态基因调控网网络演化的研究分成两个步骤来进行:首先, 通过MT算法对网络中模体的转换进行了研究, 并通过实验证实了本研究的有效性; 其次, 通过基于隐空间特征的符号判别算法对将来时刻基因调控网快照的连边符号进行了判别, 将研究领域扩展到带符号网络.对动态基因调控网网络演化的研究, 可以为生物学实验节约成本, 并为基因组学、药物研发的相关科研人员提供可靠的参考, 促进生物医学事业的进步.本文的研究成果如下.
(1) 研究了基因调控网的演化机制, 提出了MT算法.将以往对基因调控网的研究扩展到了动态的领域, 能够更好地捕捉到基因调控网的演化模式.实验结果表明, 基于模体演化概率的演化模型能够较好地解释基因调控网的演化机制, 在连边预测上有较好的准确性.
(2) 在连边预测的基础上, 提出了基于隐空间特征的符号判别算法, 填补了以往相关研究中忽略了基因调控网的特有的网络符号缺陷, 将对基因调控网的研究带入了有符号网络的领域.本文将以上两种算法共同结合成了DGNE方法, 弥补了以往相关算法不能进行符号判别的缺陷, 在动态基因调控网的网络演化领域具有开创性意义.
与其他类型的复杂网络相比, 对基因调控网网络演化的研究尚处于起步阶段, 在未来, 可以从以下几方面改进和进一步探索.
(1) 模体转换分析中, 只关注由3节点构成的模体, 而忽视了其他类型的模体(如4节点模体和扇形模体)转换对网络演化的影响.这些更复杂的模体在基因调控网中同样起到很重要的作用, 它们拓扑结构的转换对基因调控网网络演化的作用需要进一步的研究.
(2) 本文提出的DGNE方法由于加入了符号判别功能, 虽然在模型理论性和真实性上得到了提升, 但也不可避免地引入了一定误差.在今后的研究中, 我们将致力减少这类误差.此外, 在基于隐空间特征的符号判别算法中, 本文使用支持向量机作为符号判别的分类模型.是否存在效果更好的非线性分类器有待研究.
| [1] |
Omranian N, Eloundou-Mbebi JM, Mueller-Roeber B, Nikoloski Z. Gene regulatory network inference using fused Lasso on multiple data sets. Scientific Reports, 2016, 6: Article No.20533.
|
| [2] |
Ruyssinck J, Demeester P, Dhaene T, Saeys Y. Netter: Re-ranking gene network inference predictions using structural network properties. BMC Bioinformatics, 2016, 17(1): Article No.76.
|
| [3] |
Irrthum A, Wehenkel L, Geurts P, et al. Inferring regulatory networks from expression data using tree-based methods. PloS One, 2010, 5(9): Article No.e12776.
|
| [4] |
Turki T, Wang JT, Rajikhan I. Inferring gene regulatory networks by combining supervised and unsupervised methods. In:Proc. of the 15th IEEE Int'l Conf. on Machine Learning and Applications (ICMLA). IEEE, 2016, 140-145.
http://ieeexplore.ieee.org/document/7838135 |
| [5] |
Yang J, Yang T, Wu D, Lin L, Yang F, Zhao J. The integration of weighted human gene association networks based on link prediction. BMC Systems Biology, 2017, 11(1): Article No.12.
|
| [6] |
Clauset A, Moore C, Newman ME. Hierarchical structure and the prediction of missing links in networks. Nature, 2008, 453: 98-101.
[doi:10.1038/nature06830] |
| [7] |
Ud-Dean SM, Heise S, Klamt S, Gunawan R. TRaCE+: Ensemble inference of gene regulatory networks from transcriptional expression profiles of gene knock-out experiments. BMC Bioinformatics, 2016, 17(1): Article No.252.
|
| [8] |
Ud-Dean SM, Gunawan R. Ensemble inference and inferability of gene regulatory networks. PLoS One, 2014, 9(8): Article No.e103812.
|
| [9] |
Leskovec J, Huttenlocher D, Kleinberg J. Predicting positive and negative links in online social networks. In:Proc. of the 19th Int'l Conf. on World Wide Web. ACM, 2010, 641-650.
http://dl.acm.org/citation.cfm?id=1772756 |
| [10] |
Barzel B, Barabási AL. Network link prediction by global silencing of indirect correlations. Nature Biotechnology, 2013, 31(8): 720-725.
[doi:10.1038/nbt.2601] |
| [11] |
Carroll SB. Evo-Devo and an expanding evolutionary synthesis:A genetic theory of morphological evolution. Cell, 2008, 134(1): 25-36.
[doi:10.1016/j.cell.2008.06.030] |
| [12] |
Davidson EH. The Regulatory Genome: Gene Regulatory Networks in Development and Evolution. Elsevier, 2010.
|
| [13] |
Monteiro A, Podlaha O. Wings, horns, and butterfly eyespots: How do complex traits evolve? PLoS Biology, 2009, 7(2): Article No.e1000037.
|
| [14] |
Peter IS, Davidson EH. Evolution of gene regulatory networks controlling body plan development. Cell, 2011, 144(6): 970-985.
[doi:10.1016/j.cell.2011.02.017] |
| [15] |
Getoor L, Diehl CP. Link mining:A survey. ACM Sigkdd Explorations Newsletter, 2005, 7(2): 3-12.
[doi:10.1145/1117454.1117456] |
| [16] |
Liben-Nowell D, Kleinberg J. The link-prediction problem for social networks. Journal of the Association for Information Science and Technology, 2007, 58(7): 1019-1031.
http://dl.acm.org/citation.cfm?id=1241540.1241551 |
| [17] |
Adamic LA, Adar E. Friends and neighbors on the Web. Social Networks, 2003, 25(3): 211-230.
[doi:10.1016/S0378-8733(03)00009-1] |
| [18] |
Zhou T, Lü L, Zhang YC. Predicting missing links via local information. The European Physical Journal B, 2009, 71: 623-630.
[doi:10.1140/epjb/e2009-00335-8] |
| [19] |
Lü L, Jin CH, Zhou T. Similarity index based on local paths for link prediction of complex networks. Physical Review E, 2009, 80(4): Article No.046122.
|
| [20] |
Katz L. A new status index derived from sociometric analysis. Psychometrika, 1953, 18(1): 39-43.
[doi:10.1007/BF02289026] |
| [21] |
Hu WB, Wang H, Yan LP, Qiu ZY, Nie C, Du B. Event detection method for social networks based on node evolution fluctuations. Ruan Jian Xue Bao/Journal of Software, 2017, 28(10): 2693-2703(in Chinese with English abstract).
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=5153&journal_id=jos [doi:10.13328/j.cnki.jos.005153] |
| [22] |
Caruana R, Niculescu-Mizil A. An empirical comparison of supervised learning algorithms. In:Proc. of the 23rd Int'l Conf. on Machine Learning. ACM, 2006, 161-168.
http://dl.acm.org/citation.cfm?id=1143865 |
| [23] |
Li XY, Du N, Li H, Li K, Gao J, Zhang AD. A deep learning approach to link prediction in dynamic networks. In:Proc. of the 2014 SIAM Int'l Conf. on Data Mining. SIAM, 2014, 289-297.
http://www.onacademic.com/detail/journal_1000037439498010_2dd7.html |
| [24] |
Zhu L, Guo D, Yin J, Ver Steeg G, Galstyan A. Scalable temporal latent space inference for link prediction in dynamic social networks. IEEE Trans. on Knowledge and Data Engineering, 2016, 28(10): 2765-2777.
[doi:10.1109/TKDE.2016.2591009] |
| [25] |
Hu WB, Wang H, Yan LP, Qiu ZY, Xiao L, Du B. Hybrid quantum swarm intelligence indexing for event detection in social networks. Ruan Jian Xue Bao/Journal of Software, 2016, 27(11): 2747-2762(in Chinese with English abstract).
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=4910&journal_id=jos [doi:10.13328/j.cnki.jos.004910] |
| [26] |
Hu WB, Peng C, Liang HL, Du B. Event detection method based on link prediction for social network evolution. Ruan Jian Xue Bao/Journal of Software, 2015, 26(9): 2339-2355(in Chinese with English abstract).
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=4703&journal_id=jos [doi:10.13328/j.cnki.jos.004703] |
| [27] |
Milo R, Shen-Orr S, Itzkovitz S, Kashtan N, Chklovskii D, Alon U. Network motifs:Simple building blocks of complex networks. Science, 2002, 298(5594): 824-827.
[doi:10.1126/science.298.5594.824] |
| [28] |
Alon U. Network motifs:Theory and experimental approaches. Nature Reviews Genetics, 2007, 8(6): 450-461.
[doi:10.1038/nrg2102] |
| [29] |
Qi GJ, Aggarwal CC, Huang TS. Breaking the barrier to transferring link information across networks. IEEE Trans. on Knowledge and Data Engineering, 2015, 27(7): 1741-1753.
[doi:10.1109/TKDE.2014.2313871] |
| [30] |
Ye J, Cheng H, Zhu Z, Chen M. Predicting positive and negative links in signed social networks by transfer learning. In:Proc. of the 22nd Int'l Conf. on World Wide Web. ACM, 2013, 1477-1488.
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=CC0214512913 |
| [31] |
Bader BW, Kolda TG. Algorithm 862:MATLAB tensor classes for fast algorithm prototyping. ACM Trans. on Mathematical Software, 2006, 32(4): 635-653.
[doi:10.1145/1186785.1186794] |
| [32] |
Ding C, Li T, Peng W, Park H. Orthogonal nonnegative matrix t-factorizations for clustering. In:Proc. of the 12th ACM SIGKDD Int'l Conf. on Knowledge Discovery and Data Mining. ACM, 2006, 126-135.
http://dl.acm.org/citation.cfm?id=1150420 |
| [33] |
Lee DD, Seung HS. Algorithms for non-negative matrix factorization. In:Proc. of the Advances in Neural Information Processing Systems., 2001, 556-562.
http://dl.acm.org/citation.cfm?id=3008829 |
| [34] |
Chang CC, Lin CJ. LIBSVM: A library for support vector machines. ACM Trans. on Intelligent Systems and Technology (TIST), 2011, 2(3): Article No.27.
|
| [35] |
Schaffter T, Marbach D, Floreano D. GeneNetWeaver:In silico benchmark generation and performance profiling of network inference methods. Bioinformatics, 2011, 27(16): 2263-2270.
[doi:10.1093/bioinformatics/btr373] |
| [36] |
Song L, Kolar M, Xing EP. KELLER:Estimating time-varying interactions between genes. Bioinformatics, 2009, 25(12): i128-i136.
[doi:10.1093/bioinformatics/btp192] |
| [37] |
Stevenson CS, Docx C, Webster R, Battram C, Hynx D, Giddings J, Cooper PR, Chakravarty P, Rahman I, Marwick JA, et al. Comprehensive gene expression profiling of rat lung reveals distinct acute and chronic responses to cigarette smoke inhalation. American Journal of Physiology-Lung Cellular and Molecular Physiology, 2007, 293(5): L1183-L1193.
[doi:10.1152/ajplung.00105.2007] |
| [38] |
Noh H, Gunawan R. Inferring gene targets of drugs and chemical compounds from gene expression profiles. Bioinformatics, 2016, 32(14): 2120-2127.
[doi:10.1093/bioinformatics/btw148] |
| [39] |
Barabási AL, Albert R. Emergence of scaling in random networks. Science, 1999, 286(5439): 509-512.
[doi:10.1126/science.286.5439.509] |
| [21] |
胡文斌, 王欢, 严丽平, 邱振宇, 聂聪, 杜博. 面向节点演化波动的社会网络事件检测方法. 软件学报, 2017, 28(10): 2693-2703.
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=5153&journal_id=jos [doi:10.13328/j.cnki.jos.005153] |
| [25] |
胡文斌, 王欢, 严丽平, 邱振宇, 肖雷, 杜博. 混合指标量子群智能社会网络事件检测方法. 软件学报, 2016, 27(11): 2747-2762.
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=4910&journal_id=jos [doi:10.13328/j.cnki.jos.004910] |
| [26] |
胡文斌, 彭超, 梁欢乐, 杜博. 基于链路预测的社会网络事件检测方法. 软件学报, 2015, 26(9): 2339-2355.
http://www.jos.org.cn/jos/ch/reader/view_abstract.aspx?flag=1&file_no=4703&journal_id=jos [doi:10.13328/j.cnki.jos.004703] |
 2020, Vol. 31
2020, Vol. 31


