2. 贵州省公共大数据重点实验室(贵州大学), 贵州 贵阳 550025
2. Guizhou Provincial Key Laboratory of Public Big Data(Guizhou University), Guiyang 550025, China
基因数据是富含人类重要信息的生物大数据[1], 并且是人类脱氧核糖核酸(deoxyribonucleic acid, 简称DNA)序列的总称.DNA是生物遗传信息的携带者, 与生物的繁殖、遗传及变异密切相关.DNA序列包含30亿由4种核苷酸(腺嘌呤A、鸟嘌呤G、胸腺嘧啶T、胞嘧啶C)组成的碱基对, 人类有99.9%共同的DNA序列, 其中, 大约有5 000万单核苷酸多态性(single nucleotide polymorphism, 简称SNP).SNP是最常见的DNA变异, SNP是指个体DNA序列同一位置单个核苷酸变异所引起的多态性.SNP变异由单个碱基的转换(C↔T, 在其互补链上则为C↔A)或颠换(C↔A, G↔T, C↔G, A↔T)所引起, 一般所说的SNP变异由碱基转换所致.通常对于每个SNP位点具有两个不同核苷酸(称为等位基因), 一个是高频率的主要等位基因, 一个是低频率的次要等位基因.等位基因是同源染色体上的相同位点控制同一性状的不同形式的基因.位点是染色体上一个基因或标记的位置.SNP的连锁不平衡(linkage disequilibrium, 简称LD)是一种普遍存在的生物现象, 指的是基因序列中任意两个邻近SNP之间的等位基因在多代遗传中的非随机组合现象.
随着高通量基因测序技术的发展, 测序成本大幅度降低, 产生了海量高维的基因数据.基因数据广泛用于科学研究、面向消费者服务和法律与司法鉴定等[2].例如, 在全基因组关联研究(genome-wide association studies, 简称GWAS)中可以识别与SNP相关的疾病[3].但是, SNP携带个体健康的隐私敏感信息, 并且可以唯一标识人类个体, 基因数据使用不当会导致敏感信息泄露[4], 例如, 载脂蛋白E(apolipoprotein E)基因的两个SNP(rs7412和rs429358)会增加患老年痴呆症(Alzheimer’s disease)的风险.并且在连锁不平衡下, 可以从SNP相关的敏感信息推断出其他SNP相关的敏感信息.因此, 本文基于SNP连锁不平衡相关系数, 提出基因隐私保护模型:矩阵差分隐私(matrix differential privacy, 简称MDP).该模型既可以保护基因数据和SNP连锁不平衡的隐私, 同时确保基因数据具有一定的效用.
由于SNP可以唯一标识人类个体, 并且关联表型和血缘关系等隐私敏感信息.如果没有适当地对基因数据进行隐私保护, 将会阻碍科学研究的进步和发展, 并给人类社会带来巨大影响.例如, 在基因组序列中只需30~80个独立的SNP位点就可以唯一重识别个体[5], 进而导致其关联的隐私敏感信息泄露.从GWAS中揭示个体的疾病状态[6]可能会导致工作和保险中的基因歧视[7].考虑到具有血缘关系个体之间的基因数据非常相似, 可以从GWAS中推断个体的亲戚及其相关的表型敏感信息[4].因此需要联合法律法规和隐私保护技术来实现基因数据的隐私保护.目前国内尚未有专门的基因隐私保护法律法规, 美国于1996年颁布了HIPAA(health insurance portability and accountability act)禁止基因歧视.
除了专门的基因隐私保护法律法规外, 还需要隐私保护技术来实现基因数据的隐私保护.由于基因与人类的敏感信息密切相关, 基因-疾病关联分析中目前主要有3类基因隐私保护方法, 包括密码学[8-11]、安全计算[12, 13]和差分隐私[14-18].
为了从分布式基因数据中分析罕见疾病, Chen等人[8]提出隐私保护分布式协作框架PRINCESS, 并使用AES-GCM(advanced encryption standard in galois counter mode)加密所有基因数据, PRINCESS为了保护健康信息的隐私对加密数据执行安全的分布式计算.在使用AES-GCM加密基因数据时, 由于密钥分发通信代价高而使加解密受限, 并且不可信用户解密后通过分析基因数据导致患者隐私泄露.因此, 为了防止不可信用户解密后分析基因数据导致的隐私泄露, 使用同态加密直接对密文进行计算.Ayday[9]使用Paillier密码系统和Honey加密方法保护基因数据的隐私.为了发现罕见变异与疾病易感性的关系, 基于惩罚似然的确切逻辑回归(exact logistic regression)减少偏差的方法, Wang等人[10]在同态加密的确切逻辑回归的基础上提出HEALER框架, 便于在GWAS中安全地实现小抽样的罕见疾病变异分析.为了实现查询和结果的隐私保护, Shimizu等人[11]基于加法同态加密的不经意传输(oblivious transfer)隐藏序列查询和感兴趣的基因区域.由于同态加密基于有限域数学理论, 计算效率非常低, 并且在不可信用户解密后同样面临隐私泄露的问题.
在人类基因序列之间, 安全计算编辑距离(edit distance)在医学的个人基因数据和公共健康领域呈现出许多有趣的应用.Wang等人[12]结合基因编辑距离近似算法和隐私集合差大小协议设计隐私编辑距离协议, 并基于此, 设计全基因组安全相似患者查询系统GenSets.最近的工作表明, 个体的微生物DNA序列与人类个体标识相符合, 并且可以关联基因数据集中敏感属性的实际身份.目前, DNA隐私保护分析工具不满足微生物测序研究的要求.为了解决微生物测序的隐私问题, Wagner等人[13]使用安全计算实现宏基因组分析.基因数据的安全计算中计算效率低, 而且通信代价高.
从基因数据选择到GWAS统计值的隐私保护, 差分隐私[14]已经广泛应用于基因数据.例如, 在DNA数据选择过程中, Zhao等人[15]利用连锁不平衡对高维单体型降维到单体型块, 并通过对单体型块的次要等位基因计数加噪音产生差分隐私实验数据集, 不但保护了患者的隐私, 而且保证了DNA数据的效用.在隐私保护数据选择中仅仅通过对次要等位基因计数加噪音来实现差分隐私.由于隐私攻击对参与GWAS患者的隐私具有潜在的威胁, Cai等人[16]提出了差分隐私技术是一个有希望的研究方向, 差分隐私通过注入随机噪音到基因型频率、基因型-疾病关联性和基因型-基因型关联性统计值.但并未考虑SNP的连锁不平衡性质.假设GWAS的基因数据是不相关的, Tramèr等人[17]考虑更多合理的背景知识作为先验分布, 提出有界先验差分隐私用于GWAS中每个SNP列联表的χ2-统计值达到效用与隐私的平衡.不过, 同样没有考虑基因数据中SNP的连锁不平衡性质.然而, 在挖掘最重要的SNP的所有差分隐私方法中都具有准确度或计算效率的缺点, 为此, Simmons和Berger[18]使用等位基因检测统计值的输入扰动和自适应边界的方法来克服准确性问题.总的来说, 在GWAS中的差分隐私保护研究仅仅考虑添加噪音到统计值, 而没有考虑SNP的连锁不平衡性质, 并且没有对原始基因数据进行隐私保护.
但是, 基于GWAS中的统计值和SNP的连锁不平衡, 可以推断出患者的隐私信息.因为SNP连锁不平衡是同一染色体上相互邻近的等位基因可能同时遗传到后代.那么从一个SNP位点的敏感信息可以推断出其他SNP位点相关的敏感信息.例如, 在SNP连锁不平衡下, 观察到的SNP越多, 基因隐私保护强度越低[4].现有的工作主要有两方面的局限性:(1)没有从基因数据而仅仅是从GWAS中的统计值上实现患者的差分隐私; (2)没有考虑SNP连锁不平衡下的基因数据隐私保护.另外, 由于基因型数据只包含数值0、1和2, 如果对基因数据直接使用差分隐私机制将导致基因数据效用灾难, 详见第4.4节基因数据的效用分析.
为了解决此问题, 本文提出基因数据和SNP连锁不平衡的矩阵差分隐私保护模型.首先将单核苷酸多态性二倍体基因数据进行矩阵存储, 然后在连锁不平衡下基于严格的差分隐私定义实现二倍体基因数据以及SNP连锁不平衡的不可区分性, 最后运用模余运算进行二倍体基因数据的置换.矩阵差分隐私保护模型不仅满足差分隐私, 而且确保一定的基因数据效用.同时, 矩阵差分隐私保护模型可以扩展到基因数据的其他应用领域.本文主要贡献如下.
(1) 结合SNP二倍体基因数据的矩阵存储、SNP连锁不平衡下严格的差分隐私定义和模余运算, 提出矩阵差分隐私保护模型作为基因隐私保护的新方法.
(2) 基于拉普拉斯机制和高斯机制, 在SNP连锁不平衡相关系数下, 设计矩阵差分隐私保护模型的算法, 实现基因数据与SNP连锁不平衡的隐私保护.
(3) 矩阵差分隐私保护模型确保基因数据效用在区间[R0, 1]中, 其中, R0表示当隐私预算最小时矩阵差分隐私下噪音矩阵中模3余0元素数量的百分比值.
本文第1节介绍基因背景知识以及矩阵计算、模余运算和差分隐私的预备知识.第2节提出矩阵差分隐私保护模型.第3节对矩阵差分隐私进行理论分析.第4节分别对矩阵差分隐私的隐私保护和基因数据效用进行实验分析.第5节对全文进行总结.
1 预备知识首先介绍基因的背景知识.然后介绍矩阵计算、模余运算和差分隐私的预备知识.
1.1 基因组尽管人类的DNA大部分是相同的, 但是产生的变异大约有5 000万, 其中SNP是人类最常见的DNA变异.由于每个SNP位点的两个核苷酸分别从父亲和母亲的基因中遗传而来, 因此可能是高频率的主要等位基因, 也可能是低频率的次要等位基因.每个SNPgi具有次要等位基因的频率为
在人类基因组序列中, 每个序列可以表示为有序的SNP序列g1, g2, …, gm序列, 其中, 每个gi∈{0, 1, 2}.假设gi与gj相互连锁不平衡, (B, b)和(D, d)分别是gi和gj的等位基因.假设(p1, 1–p1)和(p2, 1–p2)分别是(B, b)和(D, d)的等位基因概率.这里, 等位基因频率即是等位基因的概率.如果gi和gj相互独立, 那么个体在gi和gj的主要等位基因是B和D的概率为p1p2.然而, 由于gi和gj的关联性, 因此连锁不平衡系数为LD=P(BD)–P(B)P(D), 其中, 在连锁不平衡下, P(BD)等于在SNP位点i和j的等位基因B和D共同出现在群体中的频率, 并使用
对于两个n×m矩阵S=(sij)n×m和T=(tij)n×m, 其中, 1≤i≤n, 1≤j≤m.S和T之间的加运算定义为(cij)n×m= (sij)n×m+(tij)n×m, 其中, cij=sij+tij.另外, round(S)表示运用四舍五入规则将矩阵S中的元素取整的近似运算.
1.3 模余运算给定整数s、t、q和r, 余数r=s–qt表示为r ≡s mod t(0 < r < t), 该运算称为模余运算.如果任意整数si(1≤i≤k)除以t的余数都是r, 那么集合R=(s1, s2, …, sk)构成一个等价类.因此, 从集合R中选择一个整数si满足等式r=si–qit的概率是1/k.
1.4 差分隐私根据两个相同的概率分布是不可区分的, 对于个体数据的集合, 差分隐私[14]确保一个攻击者的能力是相同的, 独立于任何个体是否在数据集中.因此, 在同样大小的数据集之间, 邻近数据集仅只有一个不同.也就是说, 两个邻近数据集X1和X2的汉明距离(Hamming distance)为d(X1, X2)=1.其中, 差分隐私定义如下.
定义 1(差分隐私. 给定(ε≥0, 如果有任意两个邻近数据集X1和X2, 对于拥有全背景知识的攻击者, 随机机制M的任意输出S⊆Range(M)使得
其中, 1–δ∈[0, 1]是M满足(ε, δ)-差分隐私的概率, 并且, 如果δ=0, 那么M是ε-差分隐私.
为了实现差分隐私机制, 需要计算查询函数f的敏感度, 查询函数f:X→Rk的敏感度是
| $\Delta f = {\max _{d({X_1}, {X_2}) = 1}}{\left\| {f({X_1}) - f({X_2})} \right\|_1}$ | (1) |
另外, 差分隐私具有后处理(post-processing)和并行组合(parallel composition)[19]的性质.
性质 1(后处理). 随机机制M:X→R关于数据集X是(ε, δ)-差分隐私, f:R→R’是一个随机映射, 那么f°M:X→R’是(ε, δ)-差分隐私.
性质 2(并行组合). 随机机制Mi满足(εi, δ)-差分隐私, 数据集Xi是X的子集, 且Xi∩Xj=0(i≠j), 那么Mi的并行组合满足(max{εi}, δ)-差分隐私.
2 基因隐私保护模型首先引入SNP连锁不平衡下对基因数据的攻击模型, 接下来提出基因隐私保护模型:矩阵差分隐私.
2.1 攻击模型因为通过SNP可以识别个体及其相关的敏感信息.假设攻击者已经观察到隐藏的SNP, 并且攻击者是honest-but-curious.攻击者可以通过成对的SNP连锁不平衡获得敏感信息, 例如相邻两个位点i和j的SNPgi和gj, 它们之间存在SNP连锁不平衡, 如果gi与某种疾病易感性相关, 那么gj也与该疾病相关.
2.2 矩阵差分隐私保护模型在SNP连锁不平衡下, 由于基因数据的隐私保护需求, 我们首先给出基因隐私保护模型——矩阵差分隐私, 如图 1所示, 该模型主要包括3部分.第1部分为编码SNP二倍体基因数据并用矩阵存储.第2部分为对已编码的SNP二倍体基因数据进行随机扰动, 同时满足基于SNP连锁不平衡下的差分隐私.第3部分为使用模余运算置换随机扰动的SNP基因数据.其中, 各个部分的主要思想如下.

|
Fig. 1 The genomic privacy preserving framework for SNP linkage disequilibrium 图 1 SNP连锁不平衡下的基因隐私保护模型 |
在第1部分, B表示主要等位基因, b表示次要等位基因, 根据等位基因的频率, 将主要等位基因B编码为0, 次要等位基因b编码为1, 并且B, b∈{A, C, G, T}, 编码基因型BB为0, Bb为1, bb为2.那么对于n个个体, 每个个体有m个SNP, 用矩阵表示为X=(xij)n×m(1≤i≤n, 1≤j≤m), 且xij∈{0, 1, 2}表示第i个个体第j个位点的SNP基因型.
第2部分是对SNP二倍体基因数据进行随机扰动, 并且满足SNP连锁不平衡下的差分隐私.图 2所示为SNP二倍体基因型数据随机扰动的主要思想, 根据SNP连锁不平衡下的差分隐私扰动机制, 将SNP二倍体基因型矩阵元素xij∈{0, 1, 2}分别以概率p1、p2和p3进行随机扰动.这里, p1、p2和p3是SNP连锁不平衡下差分隐私随机噪音对应的概率.
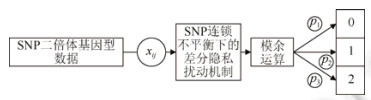
|
Fig. 2 The differential privacy perturbation mechanism for SNP linkage disequilibrium 图 2 SNP连锁不平衡下的差分隐私扰动机制 |
如图 2所示, 第3部分对随机扰动的二倍体基因型数据进行模余运算, 使其具有SNP二倍体基因型数据的语义, 并根据等位基因频率和基因型编码置换为相应的基因型.
2.3 矩阵差分隐私矩阵X=(xij)n×m表示n个个体的SNP, 每个个体的DNA序列有m个SNP, 其中, xij∈{0, 1, 2}表示个体i的SNPgj的随机变量.特别地, Xi=(xi1, xi2, …, xim)表示个体i的SNP序列取值.
| $\Delta f = {\max _{d(({x_{ij}})_{n \times m}^1, ({x_{ij}})_{n \times m}^2) = 1}}{\left\| {f(({x_{ij}})_{n \times m}^1) - f(({x_{ij}})_{n \times m}^2)} \right\|_1}$ | (2) |
因为xij∈{0, 1, 2}, 所以查询函数f的敏感度为Δf=2.
本文用
下面结合矩阵加运算、SNP连锁不平衡下的差分隐私定义和模余运算, 给出矩阵差分隐私的定义.
定义 2(矩阵差分隐私). 给定ε≥0, 任意两个邻近矩阵
| $M = [{({x_{ij}})_{n \times m}} + round({({y_{ij}})_{n \times m}})]{\text{ }}\bmod {\text{ }}3$ | (3) |
是
另外, 由于个体i的SNP序列值表示为向量Xi=(xi1, xi2, …, xim).类似地, 下面我们来定义向量差分隐私.
定义 3(向量差分隐私). 给定ε≥0, 任意两个邻近向量
| $M = [{({x_{ij}})_{1 \times m}} + round({({y_{ij}})_{1 \times m}})]{\text{ }}\bmod {\text{ }}3$ | (4) |
是
因此, 向量差分隐私是矩阵差分隐私的特例.下面给出矩阵差分隐私的通用算法1.其中, 概率分布(Δfc/max
算法 1. 在SNP连锁不平衡下的矩阵差分隐私.
输入:SNP二倍体基因型矩阵(xij)n×m, 且xij∈{0, 1, 2}.初始化ε, δ和Δf;
输出:随机扰动和置换的SNP二倍体基因型矩阵(sij)n×m.
1:计算SNP连锁不平衡的相关系数rij
2:生成噪音矩阵(yij)n×m, 且
| $3: ${({s_{ij}})_{n \times m}} = [{({x_{ij}})_{n \times m}} + round({({y_{ij}})_{n \times m}})]{\text{ }}\bmod {\text{ }}3$$ |
下面从理论上分析矩阵差分隐私的性质.
定理 1. 矩阵差分隐私是
证明:让
| $\Pr [({x_{ij}})_{1 \times m}^1 + {({y_{ij}})_{1 \times m}}] \leqslant {{\text{e}}^{r_i^{\max }\varepsilon }}\Pr [({x_{ij}})_{1 \times m}^2 + {({y_{ij}})_{1 \times m}}] + \delta $ | (5) |
由性质2可知, 不等式
| $\begin{align} &\Pr [({{x}_{ij}})_{n\times m}^{1}+round({{({{y}_{ij}})}_{n\times m}})]{{\text{e}}^{\max {{\{r_{i}^{\max }\}}^{n}}\varepsilon }}\Pr [({{x}_{ij}})_{n\times m}^{2}+round({{({{y}_{ij}})}_{n\times m}})] \\ &+\delta \\ \end{align}$ | (6) |
| $\Pr [(({x_{ij}})_{n \times m}^1 + round({({y_{ij}})_{n \times m}})){\text{ }}\bmod {\text{ }}3] \leqslant {{\text{e}}^{\max {{\{ r_i^{\max }\} }^n}\varepsilon }}\Pr [(({x_{ij}})_{n \times m}^2 +\\ round({({y_{ij}})_{n \times m}})){\text{ }}\bmod {\text{ }}3] + \delta $ | (7) |
所以不等式
为了分析矩阵差分隐私的效用, 因为
定理 2. 矩阵差分隐私的效用在[R0, 1]区间, R0表示隐私预算ε最小时矩阵差分隐私下噪音矩阵中模3余0元素数量的百分比值.
证明:首先考虑3种极端的情况.
(1) 当噪音矩阵Y=(yij)n×m的所有元素满足round(yij) mod 3=0时, round((yij)n×m)的所有元素都模3同余0.因此, 在(xij)n×m与(sij)n×m⊆Range(M)之间的所有SNP二倍体基因型数据相同.因此, 矩阵差分隐私机制的最大效用为1.
(2) 当噪音矩阵Y=(yij)n×m的所有元素满足round(yij) mod 3≡1时, (0+1) mod 3≡1, (1+1) mod 3≡2和(2+1) mod 3≡0.因此, (xij)n×m与(sij)n×m⊆Range(M)之间的所有SNP二倍体基因型取值都不相同, 此时矩阵差分隐私机制的效用是0.
(3) 当噪音矩阵Y=(yij)n×m的所有元素满足round(yij) mod 3≡2时, (0+2) mod 3≡2, (1+2) mod 3≡0和(2+2) mod 3≡1.因此, (xij)n×m与(sij)n×m⊆Range(M)之间的所有SNP二倍体基因型取值也都不相同, 此时矩阵差分隐私机制的效用是0.
上述证明中考虑(2)和(3)两种极端情况, 使矩阵差分隐私下基因数据的效用为0.然而, 由于噪音的随机性, 矩阵差分隐私下基因数据的最小效用是大于0的, 详见第4.4节基因数据的效用分析.下面考虑第4种情况.
(4) 在矩阵差分隐私中, 由于隐私预算ε越小, 邻近基因数据矩阵
综上, 由于噪音的随机性, 矩阵差分隐私机制的效用属于区间[R0, 1].
定理 3. 考虑连锁不平衡、矩阵加运算和模余运算的计算复杂度分别为O(n×m2)、O(n×m)和O(n×m).矩阵差分隐私的计算复杂度如下:(1)当n=m时, 矩阵差分隐私的计算复杂度为O(n3); (2)当n > m时, 矩阵差分隐私的计算复杂度为O(nm2); (3)当n < m时, 矩阵差分隐私的计算复杂度为O(nm2).
证明:在矩阵差分隐私中, 产生随机噪音是有效的, 忽略其计算复杂度, 而计算连锁不平衡、矩阵加运算和模余运算分别需要8n×(m2-m)、n×m和n×m次运算, 考虑3种情况.
(1) 当n=m时, 矩阵差分隐私的计算复杂度为O(n3).
(2) 当n > m时, 矩阵差分隐私的计算复杂度为O(nm2).
(3) 当n < m时, 矩阵差分隐私的计算复杂度为O(nm2).
总之, 矩阵差分隐私满足差分隐私的定义, 同时具有效用属于区间[R0, 1], 其中, R0是矩阵差分隐私下隐私预算最小时噪音矩阵中模3余0元素数量的百分比值, 并且矩阵差分隐私的计算复杂度是多项式时间的.
4 实验分析本文在矩阵差分隐私下选择拉普拉斯分布和高斯分布来进行实验分析.首先进行噪音分析, 然后与拉普拉斯机制和高斯机制比较分析矩阵差分隐私保护模型的隐私和效用.在所有的实验分析中, 考虑SNP二倍体基因型数据的特点, 初始化SNP连锁不平衡的相关系数为rij=1和敏感度Δf=2.另外, 分别初始化隐私预算ε=0.001和概率值δ=0.01.
4.1 数据集国际人类基因组单体型图计划(Int’l Hapmap Project)的数据是公开可用的[21], 本文使用2010年5月发布的阶段III的165个CEU(utah residents with northern and Western European ancestry from the CEPH collection)群体的22号染色体的基因型和频率数据集.在实验分析之前, 基于频率数据集预处理基因型数据集, 将SNP二倍体基因型数据编码为0、1和2.在CEU基因型数据集中, 将丢失的数据’NN’用0代替.本文分别选择500、1 000和1 500个SNP位点进行实验分析.
4.2 噪音分析在矩阵差分隐私中, 尺度参数为
(yij)n×m.在两种机制下, 图 3所示分别计算矩阵round((yij)165×500)、round((yij)165×1000)和round((yij)165×1500)模3余0的噪音数量的百分比值R.可以观察到, 模3余0的噪音数量百分比值随着隐私预算的增加而增加, 而不随噪音数量的大小而发生变化.这个结果为解释隐私和基因数据效用的实验结果奠定了基础.随着隐私预算的增加, 拉普拉斯机制与高斯机制相比, 所有模3余0的噪音数量的百分比值明显更快地增加.当隐私预算ε=7时, 拉普拉斯机制的R值将达到80%, 而高斯机制的R值才达到40%.这是因为, 在相同的隐私预算下, 拉普拉斯分布与高斯分布相比, 基于拉普拉斯机制的矩阵差分隐私产生的噪音矩阵中模3余0的元素更多.
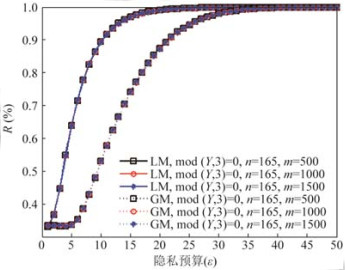
|
Fig. 3 The percentage of noises matrix entries module 3 satisfying the residue to be 0 for matrix differential privacy 图 3 矩阵差分隐私下噪音矩阵模3余0的元素数量的百分比值 |
4.3 隐私分析
为了评估基因隐私保护模型的隐私, 对于拥有全背景知识的攻击者, 本文定义标准化期望估计误差作为隐私度量.因为元素xij在矩阵差分隐私下的随机扰动元素为sij, 因此, 定义基因数据的隐私度量为
| $E = \frac{{\sum\limits_{1 \leqslant i \leqslant n} {\sum\limits_{1 \leqslant j \leqslant m} {P({s_{ij}})} } {{\left\| {{s_{ij}} - {x_{ij}}} \right\|}_1}}}{{mn}}$ | (8) |
通过比较, 我们来分析矩阵差分隐私与拉普拉斯机制、高斯机制的标准化期望估计误差.如图 4和图 5所示, 矩阵差分隐私、拉普拉斯机制和高斯机制的标准化期望估计误差都随隐私预算的增大而减小.主要原因是, 隐私预算越大, 拉普拉斯分布和高斯分布的方差越小, 矩阵差分隐私产生模3余0的噪音越多.因此拉普拉斯机制和高斯机制直接添加噪音到SNP基因型数据会导致效用灾难, 而矩阵差分隐私通过噪音模余运算提高了SNP基因型数据的效用, 见第4.4节矩阵差分隐私的效用分析.由此, 矩阵差分隐私实现了基因数据的隐私保护, 不过, 隐私保护强度显然低于拉普拉斯机制和高斯机制.另外, 由图 4和图 5可知, 随着隐私预算的增加, 高斯机制的标准化期望误差较拉普拉斯机制要大, 为了更好地权衡隐私和效用, 可以选择拉普拉斯机制实现矩阵差分隐私.
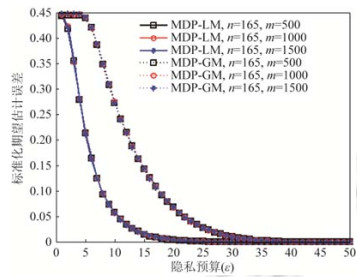
|
Fig. 4 The normalized expected estimation error for matrix differential privacy 图 4 矩阵差分隐私下的标准化期望估计误差 |
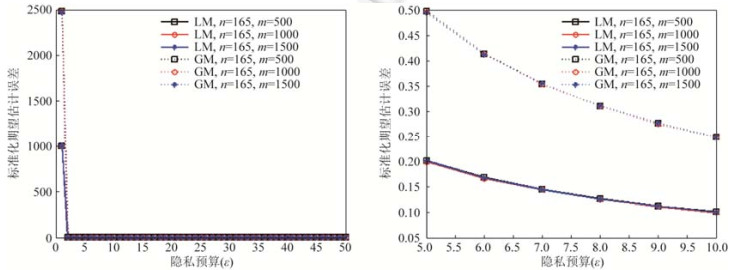
|
Fig. 5 The normalized expected estimation error for Laplace mechanism and Gaussian mechanism 图 5 拉普拉斯机制和高斯机制下的标准化期望估计误差 |
因此, 根据SNP连锁不平衡下差分隐私的不可区分性, 矩阵差分隐私实现了SNP基因型数据和SNP连锁不平衡的隐私保护.
4.4 效用分析尽管矩阵差分隐私可以实现SNP基因型数据的隐私保护, 考虑到SNP基因型数据的分析, 因此还需要分析SNP基因型数据的效用.在矩阵差分隐私中, 对于原始的SNP基因型数据(xij)n×m和扰动后的SNP基因型数据(sij)n×m, 根据
如图 6所示, 随着隐私预算的增加, 矩阵差分隐私保护模型下的基因数据效用递增, 并且增长到100%保持不变.这是因为, 随着隐私预算增大, 拉普拉斯分布和高斯分布的方差变小, 矩阵差分隐私产生模3余0的噪音就更多.当隐私预算较小时, 基于拉普拉斯机制的矩阵差分隐私可以实现更好的基因数据效用, 以此保证较好的计算不可区分性, 进而实现更好的差分隐私保护.例如, 当ε=7时, 基于拉普拉斯机制的基因数据效用可以达到80%, 而基于高斯机制的基因数据效用为40%, 这与图 3中拉普拉斯机制和高斯机制产生噪音矩阵的四舍五入近似值模3余0的噪音数量的百分比值是一致的.而图 7中随着隐私预算的增加, 基因组数据的效用保持0不变.这是因为, 拉普拉斯机制和高斯机制直接添加噪音到基因数据, 破坏了基因数据效用, 导致基因数据效用灾难.由此可知, 矩阵差分隐私比拉普拉斯机制和高斯机制更适合于基因数据的隐私保护.

|
Fig. 6 The genome data utility for matrix differential privacy 图 6 矩阵差分隐私下的基因数据效用 |

|
Fig. 7 The genome data utility for Laplace mechanism and Gaussian mechanism 图 7 拉普拉斯机制和高斯机制下的基因数据效用 |
因此, 矩阵差分隐私相比于拉普拉斯机制和高斯机制更适合于基因数据的隐私保护, 保证了基因数据和SNP连锁不平衡的隐私保护与基因数据效用之间的权衡.在表 1中, 通过比较分析, 总结矩阵差分隐私与拉普拉斯机制、高斯机制的相关性质.其中, 最小效用R0表示矩阵差分隐私在最小隐私预算下所有模3余0的噪音数量的百分比值.
| Table 1 The comparison among matrix differential privacy, Laplace mechansim and Gaussian mechanism 表 1 矩阵差分隐私与拉普拉斯机制、高斯机制的比较 |
5 结论
为了保护SNP连锁不平衡下基因关联的敏感信息, 本文提出了矩阵差分隐私保护模型.该模型满足差分隐私, 同时保证基因数据效用在[R0, 1]区间, 其中, R0是矩阵差分隐私在隐私预算最小时噪音矩阵中模3余0的噪音数量的百分比值, 并且矩阵差分隐私是多项式时间计算有效的.
对于基因数据, 基因隐私保护模型在连锁不平衡下保证隐私是可行的.通过结合矩阵加运算、SNP连锁不平衡下差分隐私的定义和模余运算, 提出了向量差分隐私和矩阵差分隐私, 并且向量差分隐私是矩阵差分隐私的特例.根据矩阵差分隐私的性质, 为了疾病标记发现, 基因隐私保护模型可以用于DNA数据集的差分隐私选择[15]; 在GWAS中, 矩阵差分隐私也可以对基于隐私编辑距离相似患者查询提供隐私保护[12]; 矩阵差分隐私阻止从GWAS统计值中识别特定的个体[16]; 并且, 矩阵差分隐私可以实现隐私保护罕见疾病变异分析[8]; 矩阵差分隐私在基因组串搜索中是有效的隐私保护方法[11].更进一步说, 在矩阵差分隐私下可以实现宏基因组分析[13].因此, 矩阵差分隐私可以推广到基因数据收集、搜索和序列配对等应用的隐私保护中.
在矩阵差分隐私中, 可以通过行划分、列划分或者其他快速矩阵计算方法[22]降低其计算复杂度, 进而提高计算效率.另外, 考虑高阶的SNP连锁不平衡, Samani等人[23]表明了对隐藏SNP的个体基因数据具有更强的推断攻击.Tramèr等人[17]考虑有界先验知识的差分隐私, 并应用于GWAS.通过孟德尔定律、基因变异之间的统计关系和基因与表型之间的统计关系, 在个体的基因组或表型被观察到的情况下, Humbert等人[4]详述了重构攻击推断该个体的亲戚的基因组.相比较考虑攻击者的背景知识, 本文仅考虑了SNP连锁不平衡下基因隐私保护.在下一步的工作中, 研究SNP连锁不平衡下具有先验知识的基因隐私保护模型, 除了考虑成对SNP连锁不平衡外, 还需要考虑高阶的SNP连锁不平衡, 并考虑攻击者更多的先验知识, 包括可利用的基因数据、个体的血缘关系以及重组规则等.
| [1] |
Li Y, Chen L. Big biological data:Challenges and opportunities. Genomics, Proteomics & Bioinformatics, 2014, 12(5): 187-189.
[doi:10.1016/j.gpb.2014.10.001] |
| [2] |
Naveed M, Ayday E, Clayton EW, Fellay J, Gunter CA, Hubaux JP, Malin BA, Wang X. Privacy in the genomic era. ACM Computing Surveys (CSUR), 2015, 48(1): 6:1-44.
[doi:10.1145/2767007] |
| [3] |
Wagner I. Evaluating the strength of genomic privacy metrics. on Privacy and Security (TOPS), 2017, 20(1): 2:1-34.
[doi:10.1145/3020003] |
| [4] |
Humbert M, Ayday E, Hubaux JP, Telenti A. Quantifying interdependent risks in genomic privacy. ACM Trans. on Privacy and Security (TOPS), 2017, 20(1): 3:1-31.
[doi:10.1145/3035538] |
| [5] |
Lin Z, Owen AB, Altman RB. Genomic research and human subject privacy. Science, 2004, 305(5681): 183.
[doi:10.1126/science.1095019] |
| [6] |
Homer N, Szelinger S, Redman M, Duggan D, Tembe W, Muehling J, Pearson JV, Stephan DA, Nelson SF, Craig DW. Resolving individuals contributing trace amounts of DNA to highly complex mixtures using high-density SNP genotyping microarrays. PLoS Genetics, 2008, 4(8): 1-9.
[doi:10.1371/journal.pgen.1000167] |
| [7] |
Gottlieb S. US employer agrees to stop genetic testing. British Medical Journal, 2001, 322(7284): 449.
[doi:10.1136/bmj.322.7284.449/a] |
| [8] |
Chen F, Wang S, Jiang X, Ding S, Lu Y, Kim J, Sahinalp SC, Shimizu C, Burns JC, Wright VJ, Png E, Hibberd ML, Lloyd DD, Yang H, Telenti A, Bloss CS, Fox D, Lauter K, Ohno-Machado L. PRINCESS:Privacy-protecting rare disease international network collaboration via encryption through software guard extensions. Bioinformatics, 2017, 33(6): 871-878.
[doi:10.1093/bioinformatics/btw758] |
| [9] |
Ayday E. Cryptographic solutions for genomic privacy. In: Proc. of the Int'l Conf. on Financial Cryptography and Data Security. Berlin, Heidelberg: Springer-Verlag, 2016. 328-341.[doi:10.1007/978-3-662-53357-422] https://link.springer.com/chapter/10.1007/978-3-662-53357-4_22
|
| [10] |
Wang S, Zhang Y, Dai W, Lauter K, Kim M, Tang Y, Xiong H, Jiang X. HEALER:Homomorphic computation of exact logistic regression for secure rare disease variants analysis in GWAS. Bioinformatics, 2016, 32(2): 211-218.
[doi:10.1093/bioinformatics/btv563] |
| [11] |
Shimizu K, Nuida K, Rätsch G. Efficient privacy-preserving string search and an application in genomics. Bioinformatics, 2016, 32(11): 1652-1661.
[doi:10.1093/bioinformatics/btw050] |
| [12] |
Wang XS, Huang Y, Zhao Y, Tang H, Wang X, Bu D. Efficient genome-wide, privacy-preserving similar patient query based on private edit distance. In: Proc. of the 22nd ACM SIGSAC Conf. on Computer and Communications Security. New York: ACM, 2015. 492-503.[doi:10.1145/2810103.2813725] https://www.researchgate.net/publication/301415304_Efficient_Genome-Wide_Privacy-Preserving_Similar_Patient_Query_based_on_Private_Edit_Distance
|
| [13] |
Wagner J, Paulson JN, Wang X, Bhattacharjee B, Bravo HC. Privacy-preserving microbiome analysis using secure computation. Bioinformatics, 2016, 32(12): 1873-1879.
[doi:10.1093/bioinformatics/btw073] |
| [14] |
Dwork C, Pottenger R. Toward practicing privacy. Journal of the American Medical Informatics Association, 2013, 20(1): 102-108.
[doi:10.1136/amiajnl-2012-001047] |
| [15] |
Zhao Y, Wang X, Jiang X, Ohno-Machado L, Tang H. Choosing blindly but wisely:Differentially private solicitation of DNA datasets for disease marker discovery. Journal of the American Medical Informatics Association, 2015, 22(1): 100-108.
[doi:10.1136/amiajnl-2014-003043] |
| [16] |
Cai R, Hao Z, Winslett M, Xiao X, Yang Y, Zhang Z, Zhou S. Deterministic identification of specific individuals from GWAS results. Bioinformatics, 2015, 31(11): 1701-1707.
[doi:10.1093/bioinformatics/btv018] |
| [17] |
Tramèr F, Huang Z, Ayday E. Differential privacy with bounded priors: Reconciling utility and privacy in genome-wide association studies. In: Proc. of the 22nd ACM SIGSAC Conf. on Computer and Communications Security. New York: ACM, 2015. 1286-1297.[doi:10.1145/2810103.2813610] https://www.deepdyve.com/lp/association-for-computing-machinery/differential-privacy-with-bounded-priors-reconciling-utility-and-Ev50l9H0n0
|
| [18] |
Simmons S, Berger B. Realizing privacy preserving genome-wide association studies. Bioinformatics, 2016, 32(9): 1293-1300.
[doi:10.1093/bioinformatics/btw009] |
| [19] |
McSherry FD. Privacy integrated queries: An extensible platform for privacy-preserving data analysis. In: Proc. of the 2009 ACM SIGMOD Int'l Conf. on Management of Data. New York: ACM, 2009. 19-30.[doi:10.1145/1559845.1559850] https://www.researchgate.net/publication/221213139_Privacy_integrated_queries_an_extensible_platform_for_privacy-preserving_data_analysis
|
| [20] |
Dwork C, Roth A. The algorithmic foundations of differential privacy. Foundations and Trends® in Theoretical Computer Science, 2014, 9(3-4): 211-407.
[doi:10.1561/0400000042] |
| [21] |
NCBI retiring HapMap Resource. https://www.ncbi.nlm.nih.gov/variation/news/NCBI_retiring_HapMap/
|
| [22] |
Golub GH, Van Loan CF. Matrix Computations. Baltimore: The Johns Hopkins University Press, 2012: 1-104.
|
| [23] |
Samani SS, Huang Z, Ayday E, Elliot M, Fellay J, Hubaux JP, Kutalik Z. Quantifying genomic privacy via inference attack with high-order SNV correlations. In: Proc. of the 2015 IEEE Security and Privacy Workshops. IEEE, 2015. 32-40.[doi:10.1109/SPW.2015.21] https://www.researchgate.net/publication/278685494_Quantifying_Genomic_Privacy_via_Inference_Attack_with_High-Order_SNV_Correlations
|
 2019, Vol. 30
2019, Vol. 30


