2. 北京理工大学 计算机学院, 北京 100081;
3. 商丘师范学院 计算机学院, 河南 商丘 476000
2. School of Computer Science and Technology, Beijing Institute of Technology, Beijing 100081, China;
3. School of Computer Science and Technology, Shangqiu Normal University, Shangqiu 476000, China
医学影像(medical imaging)是指为了医疗或医学研究, 对人体或人体某部分, 以非侵入方式取得内部组织影像的技术与处理过程.随着各种科学技术的迅速发展, 医学影像技术也有了飞速的进步[1], 逐渐形成了以X射线(包括CT)、磁共振成像、超声和核医学为代表的多种医学影像方法[2].目前, 应用于医疗的影像技术主要有基于X-ray的、基于超声波的、基于伽马射线的、基于磁共振的、光学摄影(内视镜)和其他成像技术(如萤光血管显影术、显微镜等).医学影像逐渐在疾病诊断中变得不可或缺[3], 特别是在癌症诊断中与活检相结合, 逐步成为精确定论的重要依据[4].但随着科学技术的发展和医学影像应用的推广[5], 有越来越多的医学图像需要医生解读[6].医学图像解读逐渐成为一个挑战性的工作[7], 医生有可能会因为经验不足或疲劳而产生解读错误, 使一些疾病漏诊[8], 导致假阴性出现[8-15], 也可能将非病变解读为病变, 或将良性病变误解读为恶性, 导致假阳性出现.据统计, 医学影像的疾病误诊率可达到10%~30%[14, 16].由此产生的假阴性结果会使病人错过最佳的治疗时机, 而假阳性病例一般要依靠活检等临床手段排除, 这不但增加了病人的费用, 也给他们增添了痛苦和病情恶化风险.在此形势下, 计算机辅助检测(computer aided detection, 简称CADe)与计算机辅助诊断(computer aided diagnosis, 简称CADx)的出现, 最终成为时代的需求[7, 17, 18].
1963年, Lodwick等人发表了把X光片数字化的方法[18, 19], 基于医学图像的CAD开始产生, 在此领域的研究也开始活跃[20, 21].现在, CAD通常被看作医生的诊断决策参考[22, 23]和“第二意见(second look/opinion)”[12, 24]. CAD能够减轻医生的工作负担[25], 但还不能替代医生诊断[11].经过50年的发展, 特别是近些年, 医学图像CAD系统研究成果丰硕, 已经成为医学影像学和放射诊断学领域的热门课题[13], 在各种疾病诊断中被广泛应用[26]. CAD系统对于疾病的早期筛查尤为重要, 早期筛查对疾病的诊断、治疗起着重要的作用.肺癌、乳腺癌、结肠癌和前列腺癌是目前四大患病率和死亡率最高的疾病.肺癌是世界上致病率[27]和致命率最高的癌症, 2015年统计表明, 在中国, 约有4 929 000新增病例和2 814 000人因癌症死亡[28]; 在美国, 每年约有221 200新增病例, 死亡158 040人.肺癌的总体5年存活率为16.8%[29].早期筛查是减少肺癌死亡率最有效的方法[30].肺部筛查检查出的癌症患者85%为临床Ⅰ期, 他们的10年存活率为88%[31].乳腺癌早期筛查项目能够减少30%~70%的致死率[32].大多数结肠癌来自息肉, 而息肉转变成癌症一般要5年~15年的时间, 早期检测和去除10mm以下的息肉能够有效地减少结肠癌的发病率[33, 34].前列腺癌目前在美国男性中是发病率最高的癌症[35], 早期发现和切除病灶是疾病治疗和提高病人生存率最有利的方法[36].目前, 学者对CAD系统的研究针对以上4种癌症的较多.本文以这些医学部位为主线, 结合由不同成像技术所产生的不同特性的医学图像, 从应用CAD系统较多的病类出发, 对医学图像CAD系统近几年的研究进展进行综述.
本文第1节对医学图像CAD系统进行概述.第2节~第5节分别从肺、乳腺、结直肠、前列腺这4部分, 按照不同的成像技术和病类对近几年的CAD系统进行梳理.第6节回顾医学图像CAD系统的性能评估方法.第7节汇总文中提到的CAD系统所用的主要算法和去假阳性或诊断分类时用到的特征.第8节对此研究领域面临的问题进行描述, 并对研究前景进行展望.第9节对本文进行总结.
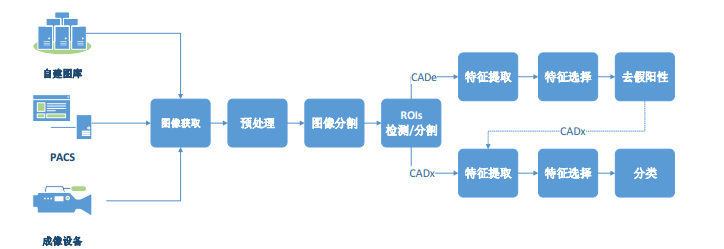
1 医学图像CAD系统概述基于医学图像的CAD系统分为两类:一类是计算机辅助检测(CADe)系统, 在医学图像上检测异常并定位呈现出来; 另一类是计算机辅助诊断(CADx)系统, 在医学图像上检测异常并帮助医生决定异常的类别以及恶性级别.CADe系统与CADx系统的一般处理框架如图 1所示.
|
Fig. 1 Workflow of computer aided detection/diagnosis systems 图 1 CAD系统流程图 |
从图 1可以看出, CAD系统一般包含7个步骤:图像获取、预处理、图像分割、感兴趣区域(ROIs)检测、特征提取、特征选择、去假阳性(CADe)或诊断分类(CADx).CADx系统也可以在假阳性去除步骤后再进行特征提取、特征选择、ROIs良恶性分类(或评估).不同类别的医学图像在CAD的具体环节上略有不同.
● 图像获取是指系统获取医学图像的方式, 一般有3种方式:(1)从自建影像库获取, 这些库一般是用从合作医院得到的医学图像建立的[37]; (2)通过影像产生设备所附系统获取, 如PACS读取数据[38]; (3)直接从成像系统实时得到数据[39].
● 预处理过程是指矫正由于介质衰减、噪声或运动伪影而导致的失真, 对原始图像做归一化处理[40], 通过去噪和增加对比度的方式增强图像的显示质量[41], 以便后续环节的处理.
● 为了减少外围组织或背景对感兴趣区域检测的干扰, 减少计算量, 预处理之后, 有些CAD系统还要进行一步图像分割操作, 把欲研究区域从背景或周围组织中分离出来.图像分割是这一步也是CAD系统的基础, 也是图像处理中最重要的步骤之一[42], 对图像的分析任务非常重要, 大部分图像描述和识别技术高度地依赖分割的结果[43].
● ROI检测是依据密度、形状等特征把所有疑似病变的区域抽取出来.抽取的信息包括了ROI的位置和形状等特征, 把ROI从周围的组织中标记出来, 所以有时也叫ROI分割.
● 特征提取是利用算法计算ROI的各种特征值, 如形状特征、视觉特征和密度特征等.
● 特征维数较多时要对特征做优化选择, 只保留对分类结果作用大的特征, 即特征选择.
● CADe与CADx的区别是:CADe系统在这个环节把检测到的疑似区域标注出来呈现给医生做诊断[44], 为了提高检测准确性, 要进行假阳性去除; CADx系统对得到的ROIs进行良、恶性分类或良恶程度分级, 即实现对疾病的诊断, 也可在CADe的基础上对检测结果做进一步诊断.
2 肺部医学图像CAD系统 2.1 基于胸部X光片(CXR)的肺结节CAD系统 2.1.1 CXR的肺结节CAD系统X光片是最早应用于医疗诊断的影像技术, 现在仍是最常用的检查方式之一.Nagata等人[45]实现了一个在胸部X光片上诊断结节的CAD系统, CAD中引入了主动轮廓方法, 并对多域值算法做了改进.系统分4个阶段.
(1) 首先用主动轮廓模型在胸片图像上分割出肺, 然后检测出肺的顶点、中线、长和宽, 与已有的模板通过参照点进行配准, 归一化.
(2) 用模板匹配方法检测初始的结节候选区域, 把归一化的肺部图像用7×7个64×64像素的矩阵化分开, 对每一个小区域通过阈值法检测ROI.
(3) 对ROIs进行两个阶段分类, 去除假阳性:第1阶段先用图像局部增强对比法分割出候选结节区域, 用密度梯度计算检测出结节; 第2阶段用多尺度模板匹配技术进一步对检测到的结节做假阳性去除.
该算法取得了良好的实验结果, Nagata等人还发现, 如果分类时考虑更多的特征, 系统性能会得到一步提升.
Htike等人[46]展示了一个3层框架的肺结节CAD系统.在预处理层, 他们选用拉普拉斯滤波器对图像进行处理; 在特征抽取层从图像的灰度共生矩阵中提取了对比度、能量和熵等44维特征; 在分类层, 使用旋转森林进行分类, X光图像被随机地分成K个子集, 在每个子集, 用PCA把子集上的特征投射到低维的空间并保持尽可能多的特异性, 这样形成了基于新特性的基分类器, 以达到提高分类准确度的同时保持分类的多样性.这个集成的旋转森林框架的分类准确度达到了令人满意的实验结果.
秦菊等人[47]对商用的CAD系统IQQA®Chest的临床应用效果进行了对比实验.由5名不同年资医师对300例中有100例是肺结节患者的医学影像进行诊断, 3个月后, 再用CAD系统辅助重新诊断.对照两次诊断结果显示:CAD系统对数字化胸片肺结节检出的敏感性和特异性等都有影响, 对特异性有明显的提高趋势.
2.1.2 CXR上的其他CAD系统Harrison等人[48]建立了一个通过阈值化时间减影图像来确定病理变化候选区域的CAD系统.每一个域值技术产生两个不同的候选区域:亮区和暗区.他们对比测试了10个不同的域值技术.与全局域值化图像相比, 通过分别域值化亮和暗的候选区域, 恰当地提高了域值区域的数量, 得到更少的全局候选区域, 但包括更多的实际病理性变化.
由于成像对象的厚度和肺域的重叠, 侧面视图通常在对比度和信噪比方面都表现出较差的图像质量.因此, 找出侧面胸部的肺部轮廓是很困难的.侧位肺部X光片能够提供用于肺气肿识别的重要信息, 例如, 仅在侧位视图中才能看到后胸部空间的扩张, 它也能准确地描述隔膜扁平化, 然而结合侧位信息的计算机诊断研究却很少. Coppini等人[49]利用前后位和侧位的放射图像识别肺气肿.针对前后位和侧位视图下的肺区域分割问题, 他们将肺边界建模为闭合的模糊曲线, 这样, 分割任务被转化成了像素成员的隶属度估计问题, 取决于图像的非线性和空间变化的特征.对此, 他们又引入了多层神经网络——改进的Kohonen网络.由于其有拓扑保留特性, 能够作为一个脊检测器平滑地增强边界的闭合, 有效地解决了CXR上肺轮廓分割的难题.
Cao等人[50]介绍了一个基于小波分析的肺间质病理诊断CAD.在预处理环节, 他们首先用sym5小波变换图像到一个二层分解中, 接着用一个函数量化高频系数阈值, 去掉频率高于域值的高频部分, 结合低频系数重建二维小波信号.在特征抽取环节, 利用小波变换抽取多分辨率和时频域局部特征.Muyoyeta等人[51]开展了一个结核病诊断的CAD系统与Xpert MTB/RIF试剂测试方法的对比实验.对350个中291个有结核病的人群, 用CAD和用Xpert MTB/RIF试剂测试方法测试得到相同的敏感性、特异性、阳性预测值(PPV)和阴性预测值(NPV).CAD的AUC为0.71.实验结果表明, CAD系统可以提高结核病筛查的有效性.
2.2 基于CT图像的肺部CAD系统X光成像是一种光影投影, 只显示1个方向上的物体结构, 体内不同器官会发生重叠, 显示不清楚.CT计算多角度的投影, 将这些信息合成横截面图像.CT图像的分辨率是X线的10倍~20倍, 能够分辨尺度较小的组织病变.CT影像是一系列连续的断层图片, 在此基础上可以对肺部进行3D建模, 比CXR更适于肺部疾病诊断.目前, 肺部疾病的计算机辅助检测/诊断研究大都集中在CT图像上进行.
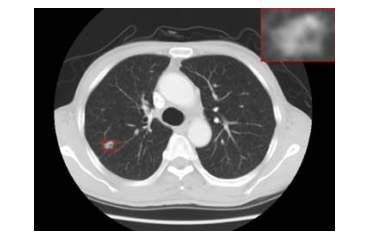
2.2.1 基于CT图像的肺结节CAD系统目前, CT检查是肺癌筛查最有效的手段, 越早发现, 病变就越小, 治愈率也就越高[52].CT图像的高分辨率[53]和高解剖结构对比度能够显示用传统X光影像几乎看不到的结节, 肺结节是“圆形模糊, 有适度的边缘, 直径不大于3cm”[54]的异常(如图 2所示), 小于1cm的则叫微结节.结节大都是肺癌的潜在表现.对以前的CT影像回顾研究表明, 75%的癌症在早期影像检查中已经表现出来.但由于此时结节很小[55, 56], 易被医生漏诊.另外, 由于读片的工作量大、医生经验不足或疲劳等因素也会产生一些解读错误.现有的CAD系统多是致力于弥补医生诊断的这些不足.
|
Fig. 2 Pulmonary nodules 图 2 肺结节 |
下面按第2节所归纳的CAD系统的一般处理框架, 对现有基于肺部CT图像的CAD系统进行介绍.
(1) 预处理
CT图像相对于X光片本身, 其噪声已经很少, 但为了得到更精确的结果, 也用一些算法对图像做预处理, 给CAD后面的操作提供更好的输入.Messay等人[57]采用降采样算法对训练用的CT图像采样, 产生可比较的片间距离, 同时可以提高计算速度, 减少噪声.为了增强细节对比度, 还使用了局部对比增强算法.为了去除加性噪声, Darmanayagam等人[58]使用了Wiener算法.Ashwin等人[59]用有限对比自适应直方图均衡法(CLAHE)去除在图像捕捉中由于眩光、噪声引起的图像对比度不高的情况.此外, 还有增强滤波(enhancement filter)、快速傅里叶变换(fast Fourier transform)、小波变换(wavelet transform)、噪声校正(noise correction)[37]、中值滤波(median filtering)、gabor滤波、直方图均衡(histogram equalization)[60]等算法在预处理时使用.在AL-TARAWNEH等人[61]提出的肺癌检测技术中, 分别使用了Gabor滤波、自动增强和快速傅里叶变换算法对肺部CT图像进行预处理, 实现去噪声、污染和干涉的目的.
(2) 分割
肺部CT图像上还包含背景、肺壁、心脏和肝等其他器官区域, 在图像预处理阶段要确定肺叶范围, 抽取肺实质(也叫肺实质分割).肺实质分割在肺部疾病诊断中是最为关键的步骤之一[62], 这个阶段精确的分割对提高系统的最终性能是非常重要的[63], 对增强肺结节检测的可靠性、准确度、精度和减少计算量起着重要的作用[64]. CT图像上的像素CT值是与组织密度成正比的, 常用阈值法分离体腔和肺实质[57].有学者使用迭代阈值法[58]进行肺实质分割.Firmino等人[65]使用了区域增长法和形态学法.进一步的分割还用到了区域、形状、相邻解剖学引导[57]和机器学习的方法[66].由于胸膜牵拉、近胸膜结节等原因, 有时分割得到的肺叶有缺口, 常用滚球法[67]等形态学方法来修正边缘.
(3) ROI检测
ROI检测的目的是查找肺叶中的肺结节, 如果有, 则确定其位置[37]和区域信息.这个环节的算法要高敏感度, 追求的性能目标是不漏检, 即召回率100%.常用像素亮度、形态学和纹理等特征检测结节[65].检测方法有灰度域值法[68]、多阈值法[43, 57, 59]、区域增长法[58].Krewer等人[69]在3D结节分割时采用了单点集合分割算法, 此外还有圆柱形和球形过滤、基于模式、形态学算子[57]、聚类、连通成份分析、基于规则的算法.Wang等人[70]在2D和3D模式下分别用到了滚球和区域增长法.Shi等人[71]的CADe系统中, 在ROI分析和提取时使用海森矩阵分析和双边拉普拉斯变换.Li等人[72]的CADe系统在感兴趣区抽取环节使用了2D大津法和数学形态学方法.3D模式下, 有时还用到基于三维物体建模和血管提取技术[41]等.
虽然3D CNN是对时空数据进行统计建模很有前景的工具, 但是它们有一个限制是需要详细的3D标签, 与获得2D标签相比, 这是非常昂贵的.现有的CAD方法依赖于获得大量肺结节的详细标签来训练模型, 这也是困难和耗时的.为了减轻这一挑战, Anirudh等人[73]提出了一种解决方案.在该方案中, 专家只需提供点标签, 即结节的中心像素, 以及其最大的预期大小.程序使用非监督方式分割来扩展3D区域得到标签体, 用于训练CNN.最后, 在SPIE-LUNGx数据集上进行实验结果表明, 即使没有准确的3D标签, 使用这些弱标签训练的网络也能在产生合理的高灵敏度的同时保持低假阳性率.
(4) 特征提取
要判断ROIs是不是结节、是良性病变还是恶性结节, CAD系统一般是根据有经验医生标注的样本库训练算法模型, 从形状、纹理、灰度和形态学等角度在候选区域上提取特征值, 利用这些特征值区分不同类别相近的对象.磨玻璃密度影(GGO)结节59%~73%是恶性, 实性结节是恶性的概率是7%~9%, 外形不规则、分叶征和毛刺征的多为恶性, 外形圆形匀滑的多为良性[65].Demir等人[74]在实验中发现, 在CADe系统中使用了外部表面纹理特征后, 系统的分类性能会有明显的提升.病人人口学特征、生活习惯特征, 如年龄、吸烟史等有时也常用到.
医学图像诊断领域尽管有各种优秀的算法出现, 但传统方法建立的特征抽取和模型选择总难鲁棒地泛化.卷积神经网络(CNN)的应用使这一问题得到了很好的解决, CNN基于人工神经网络, 针对不同分类任务, 通过反向传播算法自动强化或削减相应特征的权值, 在卷积层用滤波器自动抽取特征, 实现图像特征提取[75].图像可以直接作为CNN的输入, 从而避免了传统算法中复杂的特征提取设计和数据重建过程.
对于胸部CT肺结节诊断分类的问题, 与传统的主要依赖于结节分割进行区域分析的研究不同, Wajid等人[76]直接对原始结节图像块建模, 而不预先定义结节形态.他们提出了一个分层学习框架:多尺度卷积神经网络, 通过交替堆叠的层提取判别特征来捕获结节的异质特性.为了充分量化结节特征, 该框架利用多尺度图像块来学习一组特定类的特征.同时对每个尺度输入, 通过串联神经元在最后一层获得激活.最后, 在LIDC-IDRI上对该方法进行了评估, 证实了不用图像分割而直接对结节进行诊断分类是可行的.
(5) 特征选择
特征选择是指选择使分类结果最显著的特征子集的过程[77], 目的是减少冗余的特征, 降低特征维数[78], 以提高运算效率, 最大化分类准确性.降维常用到LDA和PCA两种经典的算法:LDA对特征值进行新的投影, 投影后不同性质的数据点的距离更大, 同性质的数据点距离更紧凑; PCA从协方差的角度, 将高维的特征映射到低维空间中表示, 并期望在所投影的维度上, 数据的方差最大.特征选择不做投影或映射, 仅从所有特征中选择部分特征使用.常用的算法有RELIEF法、顺序向前法(SFS)和遗传算法(GA)[79-82]等.
特征的高维不相关、特征子集的异构性以及样本类别分布不均衡, 一直是肺结节检测准确率的阻碍.Cao等人[83]在肺结节检测CAD中, 用一个基于多核的框架来解决这些问题.为了消除特征的高维不相关性, 整合多内核学习和特征选择, 他们提出了两种基于多内核的特征排序技术, 根据从基于内核的特征选择函数(MKFF)导出的内核空间中的预测能力(分数)来选择识别特征, 以减小特征向量的大小和提高分类性能.为了有效地融合和选择异构的特征子集, 他们通过带混合l2, 1核权重约束的多内核学习法, 恰切地结合有关参数结构的辅助信息, 以利用互补异构特征子集, 同时考虑不同多个特征子集的分类贡献.为了解决不平衡结节数据学习的问题, 他们构建了一种最优多内核过采样方法来平衡数据分布.这种方法可以生成准确的实例, 解决在构建内核分类器模型时输入空间中执行过采样的不一致问题.该方案性能的几何均值和AUC都明显超过实验中的其他对比方法.
纹理特征具有很强的表征力和特异性.Krewer等人[69]在现有基础上研究新CADx系统过程中, 在大小、形状等基础上加入纹理特征, 尝试用Relief-F、RFNC、CFS算法进行特征选择, 并分别结合J48、RF、k-NN和SVM分类算法做了实验对比.在特征选择过程中, Laws和小波特征结合得到最好的级别, 也意味着对分类性能贡献最大.最佳的整体性能是CFS特征选择、5-NN分类器和仅用纹理特征的数据集组合.这表明, 分类时采用纹理特征是有前景的.
(6) 去假阳性
检测的结果中, 有一部分得到的ROIs不是结节, 可能是表现相近的有大曲率或分叉的厚血管、呼吸或心脏运动及肺实质组织上的散射形成的斑迹等.上述非结节ROIs也叫假阳性(false positive, 简称FP)ROIs, 需要去除, 为此需要对得到的ROIs提取特征.常用的有密度[57]、形态学[58]、纹理[65]等相关特征, 也有基于统计学的如灰度共生矩阵(GLCM)[58]特征, 根据得到的特征值对ROIs进行真阳性和假阳性分类, 去除假阳性区域.对于CADe系统, 通过这个环节, 将带有位置标注的区域作为输出供医生诊断.CADx系统还要在此基础上对得到的区域进行良恶性诊断, 有的CADx系统没有去除假阳性环节, 在最终分类结果中体现ROI类别.
Camarlinghi等人[84]对蚂蚁探索通道模型(channeler ant model)、立体区域增长平台(region growing volume plateau)和基于体素的神经网络方法(voxel based neural approach)这3个CADe系统分别做了详细的介绍, 并依据每个CADe系统的不同性能指数进行了三者加权融合的分类研究.在去假阳性环节, 分别用前馈神经网络(FFNN)和支持向量机算法.实验结果表明, 相对于单个系统, 不同系统结合的检测性能有明显的提高.他们分析, 这主要是得益于这些系统在识别假阳性方面, 贡献是互补的.
Wang等人[70]的CADe系统中设计了一种基于三阶张量模式的SVM算法, 可以直接对感兴趣体(VOI)进行分类.他们使用SVM, MTANN, SVM(matrix), SVM(based on unfolding 3D matrixes)和SVM(3Dmatrix)这5个CAD方案做对比, SVM(3Dmatrix)达到了98.2%的敏感性, 假阳性率为9.1FPs/切片, 总体上更优.
(7) 分类
在CAD系统中, 常用的分类算法有基于规则的分类器、LDA、模式匹配、神经网络(NN)、马尔科夫随机场、SVM[41, 65, 85-89], 线性判别分类器[57]、k-NN[43]、多层神经网络(ANN)[58, 90]、决策树[91]等.为了提高肺结节分类识别的准确率, 李秋萍等人[92]提出了一种半监督FCM聚类算法, 引入专业医生标记的监督信息来指导聚类过程, 通过衡量两个样本特征向量之间的距离, 获得它们被划分到同一类别中的可能性.最后, 参考隶属度对非标记样本进行聚类, 在一定程度上解决了传统聚类算法聚类效果不理想的问题.
基于逐像素的深度学习方法如CNN, 由于能够避免由对复杂和细微对象的特征计算或分割不准确而导致的计算错误, 显示出了良好的发展前景[87, 93].最近的研究结果表明, 由CNN抽取的特征是可以通用的, 能够用于训练网络的特定领域之外的分类任务.文献[94]使用OverFeat训练作为自然图像中目标检测的网络.对LIDC数据库中865个CT图像上的每个结节所在区域块, 分别从矢状、冠状和轴向面抽取特征, 在网络的倒数第2层得到4 096维的特征, 最后用线性SVM分类.对于各种参数配置, 使用现成的CNN特征, 系统都执行得相当好, 但性能不如常规专用的检测系统.当把两种方法结合以后, 得到的结果明显比单独要好.从实验结果看, 在三维医疗数据辅助检测任务中, CNN特征具有进一步研究的价值.
2.2.2 基于CT影像的肺部其他疾病CAD系统针对其他系统假阳性率太高的情况, Tajbakhsh等人[95]用一种新的血管对齐多平面图表示方法和CNN实现CT肺血管造影图像上的计算机辅助肺栓塞检测系统.系统先进行肺分割, 然后用雪降算法(tobogganing algorithm)抽取肺栓塞候选区域.对每个区域, 用血管对齐多平面图像描述产生一个2通道图像表示.把得到的2通道图块放入一个CNN进行栓塞和非栓塞分类.尽管在2mm和5mm定位误差上稍有不足, 系统的诊断性能明显优于传统的手动选取特征的方法.
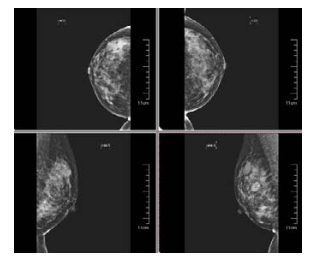
3 乳腺医学图像CAD系统 3.1 基于钼靶影像的乳腺CAD系统乳腺钼靶摄影(mammography)是一类用低剂量X-ray检查乳腺的技术, 具有全面、直观、操作简单、安全和费用低廉等特点, 是目前诊断早期乳腺癌的首选而有效的方法[96].钼靶影像按头尾位(CC)和内外斜位(MLO), 双侧分别产生两张图像(如图 3所示[97]).CAD系统利用计算机图像技术检测或诊断乳腺影像上的异常如肿块、微钙化和结构变形这些病变.
|
Fig. 3 Top left is CranioCaudal (CC) right side, top right is CC left side, bottom left is MedioLateral-Oblique (MLO) right side, bottom right is MLO left side 图 3 左上图为头尾位右侧, 右上图为头尾位左侧, 左下图为内外侧斜位右侧, 右下图为内外侧斜位左侧 |
3.1.1 预处理
乳腺钼靶图像在产生时会有内在的噪声, 在正常的腺体和恶性的组织之间, X射线的衰减差较小[98], 因此, CAD系统在预处理阶段要做的工作有降噪、增强对比度.有时, 为了减少干扰和计算量, 还要去除背景区域和MLO视图下的胸肌区域.降噪常用的算法有中值滤波[99-101]和有限对比适应性直方图均衡化(CLAHE)[99, 100]等. Jain等人[102]提出了一种组合的方法来抑制密度脉冲噪音, 引入带多状态自适应增益的非线性增强算子来增强乳腺图像特征.Bhateja等人[103]使用0型Volterra滤波器和Ⅱ型Volterra滤波器的线性组合来增强病变区域对比度.0型滤波器提供对比增强并抑制不良背景噪声, Ⅱ型滤波器提供边缘增强.
3.1.2 异常检测/分割此环节是找到并标出包含异常病变的ROIs.乳腺异常表现为肿块、微钙化、结构扭曲或双边非对称.
(1) 恶性肿块
恶性肿块的征象通常是边缘模糊、毛刺状、分叶状、边界不规则、密度不均匀、灶状致密影等.检测/分割算法常用的有区域增长法[104]、阈值法[101]等.Jen等人[105]使用灰度值量化的方法检测ROIs.Sharma等人[106]采用k-means聚类的方法检测/分割ROIs.Al-Absi等人[107]在CAD系统中用一个优化的度量计算输入图像上动态窗口和模板的不同, 对应记录矩阵中最小记录的图像窗口被作为疑似区域抽取出来.实验取得了97.73%的诊断准确度.
Dhungel等人[108]引入和评估了一种基于使用卷积神经网络和深度信念网络(DBN)作为其潜在函数的结构化预测模型, 从乳房X线照片分割肿块的新方法.他们将基于CNN和DBN的深度学习表征模型引入到条件随机场(CRF)和结构化支持向量机(SSVM)这两个结构化输出模型中.CRF模型通过使用树重加权置信传播进行推理, 并用截断拟合来学习; SSVM模型使用图形切割推理和切割平面进行训练.他们以实证的方式展示了这种方法产生的结果是DDSM-BCRP和INbreast数据库上迄今为止最好的结果.同时, 他们发现CRF模型在推理和训练时间方面明显快于SSVM, 这体现出CRF模型与深度学习潜在函数相结合的优势.
(2) 微钙化
微钙化是钙在乳腺组织中的沉淀, 通常比周围组织更亮、更小.良性病变的钙化一般是较大的粗糙且边缘较圆和光滑, 恶性钙化表现为许多、聚集、小、大小和形状变化多样、有角、不规则和多向分叉[109].钙化在乳腺X线片上表现为较高的亮度, 所以常用算法有基于小波变换的和阈值的算法.尽管微钙化检测已有先进的CAD系统出现, 但由于售价太高而不能普及, 所以此领域的研究仍是社会需求[110].
Oliver等人构建了一种基于知识的检测微钙化和簇的方法.首先建造一个字典, 字典中的元素是通过一组滤波器得到的包含不同微钙化特征的卷积块.这个字典允许表征已知微钙化的范例, 后面用于表征未知的图像.用有正负样本的微钙化字典训练Gentleboost分类器.最后, 用得到的分类器对未知的乳房X光片检测.3D图像包含更丰富的信息, 对提高医学图像CAD性能有很大的潜力.
Sahiner等人[111]提出一种在3D模式下用阈值迭代的方法检测成簇的微钙化点的方法.该系统由预筛选、聚类和假阳性减少阶段组成.在预筛选阶段, 通过增强调制的3D钙化反应函数增加微钙化样对象的显著性.使用阈值迭代和3D对象生长方法来检测作为微钙化簇的潜在中心的簇种子对象.在簇检测阶段, 使用二次阈值迭代程序识别微钙化候选物, 其应用于具有阳性钙化回馈的信噪比(SNR)增强图像体素.以每个聚类种子对象作为初始聚类中心, 动态聚类算法通过在簇种子对象的三维邻域内, 包含满足聚类标准的微钙化候选点来形成聚类候选簇.在去假阳性阶段, 使用微钙化的数量、大小和信噪比以及簇形状属性来减少FP的数量.
乳房组织的致密性和乳房X线照片图像较差的对比度, 是有效识别微钙化的最大障碍, Malar等人[112]尝试了基于小波的组织纹理分析, 使用极限学习机(ELM)在数字化乳房X线照片中的微钙化检测的方法, 并与使用灰度空间依赖矩阵(GLSDM)和基于Gabor滤波器的技术提取的不同特征向量进行比较.在此次研究中, 共使用从55个乳腺X线照片图像中提取的120个感兴趣区域, 包括正常和微钙化图像.采用小波特征进行训练, 结果表明:与Bayesnet类器、Naivebayes分类器和SVM等人工神经网络相比, ELM具有较好的分类精度(94%), 训练时间显著减少.ELM还避免了诸如局部最小值、不正确的学习率和过度拟合等问题.
通过回顾性分析, 谭婉嫦等人[113]探讨了计算机辅助诊断系统在微钙化检测与特征提取基础上的分类对于导管原位癌(DCIS)的诊断价值.分析采用乳腺X线摄影检查发现微钙化, 并使用经病理学证实的623例患者影像资料(其中, 良性病变378例, DCIS 245例).用受试者操作特征曲线(ROC)分别分析采用计算机方法提取的每个微钙化特征对于这两类病变判别的诊断效能, 以及应用所有微钙化特征集且基于支持向量机(SVM)分类器的CADx的分类诊断效能.实验结果是:CADx对于良性病变和DCIS这两类病变微钙化分类的ROC曲线下面积(Az)为0.853;特异度、准确率、敏感度分别为70.1%, 82.1%, 90.7%, 都高于单个微钙化特征的诊断性能.此结果也说明:采用CADx对于DCIS微钙化能够较好地检测与定位, 对乳腺癌早期病变的识别提供有益的参考.
(3) 结构扭曲
结构扭曲是指乳腺结构变化没有明确的肿瘤发现, 但乳腺组织结构紊乱, 这包括有毛刺从一点辐射出来和病灶收缩或腺体实质的边缘变形.结构扭曲也可以是一个附属的发现, 是第3类最常见的未扪及肿块的乳腺钼靶影像症状的癌症[114].由于它的细微和多样性表现, 在筛查时往往被漏检.如果是创伤引起的瘢痕或软组织损伤, 这种变形是良性的.
尽管是高度恶性, 乳房X线片中的结构扭曲也一直是放射科医师最容易漏检的病变.为了提高检测结构变形的准确率, Yoshikawa等人[115]开发了一种用于自动检测乳房X线照片中的结构变形的新方法.他们使用一个自适应Gabor滤波器来检测乳腺结构扭曲, 通过改变参数的组合来创建3个Gabor滤波器.选择与乳腺X线照片中逐像素的乳腺结构最佳匹配的滤镜.检测乳腺后, 进行结构密集区域的增强和假阳性减少.在实验中, 他们使用50个乳房X线照片验证了该方法的检测性能.真阳性率为82.45%, 每图像假阳性率为1.06.
Singh等人[116]的CADx系统在诊断结构变形的乳腺病变时利用了GLCM纹理、GLRLM纹理、不规则碎片形纹理和谱纹理特征, 分类时采用SVM算法, 系统的整体敏感度和准确度都很高.他们分别对4种纹理特征的系统性能进行了各种组合对比, 发现最好的输出结果是所有特征的结合.
(4) 双边非对称
双边非对称表示左、右乳腺的一侧和另外一侧对应区域对比, 没有明显的肿块, 表现出一个较大的体积或密度不同, 或更突显的导管[117].双边非对称是判断乳腺癌的一个很有价值的指标, 已被证明是乳腺癌的一个重要预测[118].目前, 研究检测双边非对称的文章较少.用到的算法有基于形状、拓扑、亮度分布、纹理等特征的算法[119].
人工免疫方法是模仿自然免疫系统的一种智能方法, 有耐噪声、无师学习、自组织、记忆等进化学习机理.自适应人工免疫网络是人工免疫网络的一种半监督学习框架, 这种自我智能的网络可用来解决复杂的分类问题, Magna等人[120]研究了基于自适应人造免疫系统(A2INET)的计算机辅助方法, 以捕获数字乳腺X光片上乳腺的早期非对称体征, 用于乳腺癌的早期诊断.在该算法中, 类模板被看作抗体, 新样本被看作抗原, 它们的距离表示其亲和力.为了测试算法的效率, 他们考虑了两个公共数据集:从DDSM中检索到32对乳腺X线影像, 包括MLO投影; 以及来自mini-MIAS数据库的30对影像.对于其他更传统的分类器LDA, k-NN和PLS-DA, A2INET产生了最佳的结果.
在二维乳腺投影X片上准确地分割病变区域非常困难, 而且不可靠, Kelder等人[121]对一个双边配准、图像特征提取和朴素贝叶斯线性分类的三环节CAD系统进行了性能评估.评估从mini-MIAS数据库中选用了161个对象, 使用留一法交叉验证, 结果显示出良好的性能.此方法通过检测和分析双边乳腺影像局部特征的非对称性, 有效归避了图像分割环节.
Wang等人[122]对一个检测乳腺肿块的CADe系统进行了结合双边乳腺密度非对称信息之后的性能评估.在600个病例2 400张全域数字乳腺影像片上, 阳性和阴性病例各占一半.两次实验分别是单独和融合双边非对称信息检测疑似肿瘤.研究结果表明:双边非对称信息检测疑似肿瘤比双侧平均密度更有效, 与传统的特征结合, 检测性能得到明显的提升.
3.1.3 特征提取与选择Murakami等人[123]使用商用的“Second Look”CAD系统, 利用形态学、密度、纹理等特征分别对152个乳腺肿瘤病例做了回顾性分析.CAD系统表现出很高的敏感度, 且不受乳腺密度和病变大小的影响.Elfarra等人[77]在CADx系统中提出了一种新的特征抽取方法——长方形中心线灰度分布法(SCLGM).这种方法达到了很高的全局测试特异性和准确度.Taylor等人[124]对商用的CAD系统R2 ImageChecker在乳腺癌筛查项目中的潜在作用进行了评估.评估结果显示:该系统对被忽略的癌变有较强的提示作用, 在医生做决策时有进一步的提示作用.
从乳腺异常ROIs中能够抽取到的特征可达几百维之多, 其中有一些特征与诊断分类无关[125]或影响非常小.如前所述, 特征选择是选择使分类结果最显著的特征子集的过程[76], 目的是减少冗余的特征、降低特征维数[77], 以提高运算效率、最大化乳腺异常分类准确性.常用的降维算法有主成分分析法(PCA)、线性判别分析法(LDA)等.特征选择算法一般有嵌入式、封装式、滤波式及混合型这4类, 在此基础上, 可以用顺序前向或后向法进行特征子集选择[78].Eltoukhy等人[126]设计了用小波变换和曲波变换的算法抽取乳腺X线图像上的特征形成特征集, 并用动态阈值法选取分类效果明显的特征.
Choi等人[127]提出一个新的集成分类框架以提高CAD系统的乳腺图像分类能力, 这个新框架分为两部分: (1)联合使用一个实例的不同特征描述和重采样产生的多样化的数据, 以准确的基分类器作为成员; (2)结合使用一个新的“组合分类选择”机制, 进一步最大化整体分类的性能.然后, 在乳腺X光基准数据集上测试两个分类应用:(1)使用分类器去除假阳性; (2)使用分类器诊断良、恶性肿块.实验结果表明, 此分类器比常用的单神经网络或SVM算法有更显著的分类性能.
深度学习模型直接从数据中自动学习特征, 并在自然场景分类和物体检测等计算机视觉问题上取得了显著的成绩[128].Arevalo等人[129]对基于传统和CNN特征的乳腺X光摄片上肿瘤CAD分类器的性能做了评估, 实验选取了传统的HOG, HGD, HCfeats, 与CNN特征相比, 基于传统特征的分类性能最好为AUC 0.799, 而CNN可达到0.86.同时, 他们在实验中还发现, 较小的CNN模型表现最好, 并得出两个结论:一方面, 说明CNN模型对于乳腺X线图像的分析表征是最好的; 另一方面, 这次自动分析有自身的特殊性, 不需要用大的CNN模型来学习表示.而且, 学习过程中应该使用具有广泛视觉变化性的训练集, 这样才能表现大量病变中呈现的纹理模型和形状特征.
3.1.4 分类在乳腺CAD系统中, SVM依然是使用最多的分类方法[99, 106, 130]; 此外, ANN也是功能强大的分类工具[77, 131]. Choi等人[127]提出了同一实例的不同特征表示相结合和数据重采样的方法生成更加多样化和精确的分类器集合成员, 还采取一种新型“组合选择”机制进一步提高整体分类性能的提高.与SVM与ANN等算法的融合的方案做对比, 此方案有明显的优势.这种集成框架还同时具有弱分类器和强分类器的工作优势.Verma等人[132]实现了在数字乳腺钼钯图像上的微钙化分类CAD系统, 系统中采用了模糊神经技术和特征抽取技术, 并发现在分辨微钙化良恶模式时, 系统与熵、标准方差和像素数是最好的结合.他们分别采用MLP, k-NN和SVM这3种算法并做性能对比, 这种方法更高效和易用.Dheeba等人[133]的论文研究了CADe系统上的一种新的分类算法:粒子群优化小波神经网络.它是一种从乳腺X光图像上抽取LAWS纹理能量的方法, 通过应用一个模式分类器对可疑区域诊断分类.实验结果展示出该算法良好的系统性能.
Cascio等人[134]对CyclopusCAD和SecondLook®两个商用的乳腺CAD系统的诊断性能进行了对比分析.两个系统分别单独对126个数字乳腺影像样例进行操作.在标准阈值时, CyclopusCAD的全局敏感性为76.9%, SecondLook®为66.2%(P=0.04).对于肿块病变, CyclopusCAD的敏感度为76.9%(0.73FP/im), SecondLook®的敏感度为61.5%(0.28FP/im).对于微钙化病变, CyclopusCAD的敏感度为76.2%(0.64FP/im), SecondLook®的敏感度为61.9%(0.19FP/im).
受人工免疫算法的启发, Peng等人[135]研究了一种半监督算法——人工免疫半监督学习算法, 以减少对标注数据的依赖.算法中引入Kent混沌在整个“抗体细胞”特征向量空间中搜索最好的解决方案.他们使用两个从UCI机器学习数据库下载的知名的乳腺癌症基准数据集做研究.在这两个数据集上进行的大量的实验和评估都显示了算法的有效性和效率, 证明了此算法在乳腺癌症自动诊断中非常有潜力.
近几年, 深度学习模型在计算机视觉和机器学习中产生了相当有竞争力的结果[136].Carneiro等人[137]展示了在医学图像分析中使用深卷积神经网络的两个重要发现:其一, 使用计算机视觉数据库(例如imagenet)预先训练的CNN模型在医学图像应用中是有用的, 尽管图像外观有显着的差异; 其二, 不需要预先配准输入图像就可以进行多视角分类, 相反, 可以分别使用在每个视图中训练的CNN产生的高级特征.针对乳房X线照片的分类, 使用头尾位和内外侧斜位视图及其相应的肿瘤和微钙化分割图:首先, 使用基于Imagenet预训练的模型为每个视图和每个分割图训练单独的CNN模型; 然后, 使用从每个分割图和未配准视图中学习到的特征训练一个最终的CNN分类器, 使用乳腺成像报告和数据系统(BI-RADS)得分作为输出, 估计患者发生乳腺癌的风险. Carneiro等人在两个数据集(InBreast和DDSM)中测试了这种方法, 显示它在ROC上产生了超过0.9的面积, 并且对于二分类问题——良性和恶性AUC超过0.9.此方法输出了非常好的分类结果, 也展示了一种解决这一具有挑战性的分类问题的全面、综合的方法.
3.2 基于乳腺超声图像的CAD系统尽管乳腺钼靶影像目前被认为是早期检查乳腺癌最可靠的方式, 能够减少18%~30%的致死率, 但其对比度不高, 肿瘤与周围正常组织的分界不明显, 这会导致10%~30%的癌症漏检[138].由于这个原因, 在最近几十年, 作为乳腺X光影像乳腺病变检查的技术辅助, 引入了超声波检查, 主要对影像学上致密的乳房提供更精确的评估.当这两种影像技术结合使用时, 鉴别囊性结节和实性结节的有效率能够达到约95%, 减少了25%~35%的活检数量[139].
超声乳腺病变CAD系统一般包括4个环节:图像预处理、图像分割、特征提取/选择和分类.散斑干涉和低对比度是超声影像的主要局限.与其他CAD系统相比, 超声乳腺病变CAD系统的预处理主要致力于增强对比度和抑制散斑.散斑降噪技术主要有滤波方法、小波域法和组合方法这3类[140].背向散射回声的密度值也可被用作表征肿瘤的特性.
Moon等人[141]提出一个通过B超散斑的统计学特征区分乳腺恶性良性病变的CAD系统.对每个肿瘤统计感兴趣区的平均散斑像素数作为散数密度, 通过一阶和二阶散斑像素统计得到像素分布值和空间关系, 以此散斑特性作为特征检测肿瘤.
Alam等人[142]开发了一个利用回声反射性、不均匀性、影、面积、纵横比、边缘不规则性和边界清析度等多种特征在超声图像上进行乳腺良性恶性病变的分类诊断系统.为了得到定量的声学特征, 他们使用滑动窗傅里叶分析, 计算了病灶和相邻区域的射频(RF)回波信号的频谱参数图, 通过跟踪的病变边界的几何和分形分析量化形态特征.他们对130例活检定期患者常规超声检查期间获得的数据进行分析, 产生曲线下的受试者操作特征面积为0.947±0.045.
Huang等人[143]在一个CADx中提出了基于模式识别的病变区域分割方法:先使用全局变分模型以减少斑点噪声; 然后, 用一种基于图的鲁棒分割方法把图像分割成一些子区域; 再结合图像纹理抽取、特征选择和分类过程, 以自动地确定与乳腺相关的肿瘤区域; 最后, 使用主动轮廓模型提取出肿瘤区域.系统在实验应用中表现出良好的性能.
Cheng等人[144]对通过避免不准确的图像处理结果而造成的潜在错误的CADx系统进行了全面的研究, 并具体对比了两个基于SADE的乳腺超声病变和肺CT结节诊断CADx系统性能.SDAE架构具备自动特征探索机制和容忍噪声的优势, 因此适于处理从不同成像方式产生的有内在噪声特性的医学图像数据.为了展示基于SDAE的CADx系统相对于传统方案的优越性, 他们实现了两种最新的常规算法以进行比较.实验结果表明, 基于SADE的CADx算法相对于常规的算法, 性能有显著的提升.也表明深度学习技术能够潜在地改变CADx设计的模式, 而无需面对具体的特征设计和选择问题.
3.3 基于CT图像的乳腺癌症CAD系统由于乳腺钼靶摄影对病变的显示存在非显著性和误报问题, 这会导致不必要的阴性穿刺活检.锥束乳腺CT扫描仪(bCT)作为一种新的专用影像方法, 能够生成高质量的层析数据以提高乳腺组织和结构的可视化, 并使病灶显示更显著.量化成像分析能够被用来从bCT图像集中抽取关于感兴趣组织的有用的数字信息[145].
CAD的准确输出依赖于对病灶的有效分割, Kuo等人[146]提出了3D模式下的乳腺肿瘤分割方法.首先, 用3D径向梯度指数分割得到一个粗糙的轮廓; 然后, 用基于3D水平集的主动轮廓算法对边缘进行细化.算法用肿块的中心点作为输入, 设定3个边缘估计停止标准:(1)体积的变化范围; (2)每一次增长迭代后得到的分割区域的平均密度; (3)已分割区域内外的平均密度变化率.
Ray等人[145]通过使用定量成像(QI)分析的方法实现一个CADx系统:系统先执行图像预处理、感兴趣区分割(用迭代分水岭分割法), 然后对分割的对象进行结构性分析(提取8个形态学参数、6个基于GLCM的纹理特征参数), 最后用ANN算法进行分类.并针对有效训练数据量较小的问题引入网络优化和交叉验证的方法加以弥补.上述两个方案准确率都较高, 如果训练更大的数据集, 系统的泛化性能还可以进一步得到提高.
3.4 基于MRI图像的乳腺癌症CAD系统自20世纪90年代以来, 乳腺MRI就开始被用于表征和检测乳腺病变.MRI的敏感度非常高, 可达到78%~ 98%, 但其特异度不足, 只有43%~75%.近些年发展起来的计算机辅助诊断软件的目的主要是为了使MRI分析和报告更加便利, 或试图进一步突出显示检测到的病变[147].
Wang等人[148]开发了一个在3D动态对比增强磁共振影像上对乳腺肿瘤分割, 利用动力学、形态学特征诊断分类的CAD系统.系统用由交叉动力学与AUC下面积相结合得到彩色映射图的方法探测潜在的乳腺病变区域, 用区域增长法分割肿瘤.采用药代动力学或传统曲线分析算法对代表肿瘤的分割的动能曲线特征化, 得到鲁棒的系统.
Pang等人[149]在乳腺磁共振图像上建立了一个全自动的乳腺肿块恶性和良性诊断分类系统.系统包括: (1)乳腺分割, 包含一个确定空气和乳腺边界, 并用曲线拟合确定肺壁的预处理步骤; (2)乳腺肿块分割, 此环节引入了Chan-Vese(CV)水平集方法; (3) SVM结合ReliefF特征选择, 用于把抽取的形状和纹理特征融合, 得到一个分类得分.这个RelifF/SVM/CVLS混合的模型达到了很好的性能.
Gallego-Ortiz等人[150]对于在动态对比增强磁共振乳腺图像上区分肿瘤和非肿瘤的CAD系统, 介绍了如何确定合适的特征和优化分类算法等问题.Agha等人[151]对一个新开发的3T MRI CAD系统的诊断性能进行了评估, 实验对120个从超声影像上看, 表现有争议的或疑似恶性病变的女性病例的3T MRI CAD诊断结果进行评估.评估结果显示:CAD系统能够达到良好的敏感性和特异性, 对避免不必要的侵入式检查过程是有效的.
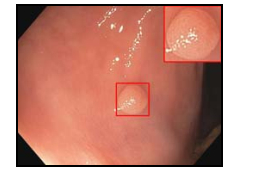
4 结直肠医学图像CAD系统 4.1 基于光学结肠镜图像的CAD系统结直肠癌(CRC)是世界范围内第3类最常见癌, 致死率第四, 每年导致700 000人死亡[152, 153].结直肠癌与结直肠息肉有着重要的关系(如图 4所示[154]).早期检查并移除息肉, 对潜在的结直肠癌患者的寿命延长是至关重要的.光学结肠镜(OC)是息肉检出最普遍的工具, 然而, 由于结直肠的结构错综复杂, 息肉的漏诊率保持在约25%左右.因此, 针对结直肠息肉的CAD系统是必要的.
|
Fig. 4 Colon polyp 图 4 结肠息肉 |
Kominami等人[39]对他们开发的实时检测结直肠息肉的CADx系统进行了应用评估.系统从窄带图像上以5像素和7像素尺度得到不同的128维SIFT特征描述子, 用级联的k-means聚类形成描述不同类型图像的特征袋, 最后, 用线性核SVM分类.通过对41个病人、118个直肠癌切除后的病变检查, 把系统的诊断结果与内窥镜、细胞组织学诊断结果进行了对比, 取得了很高的一致性、敏感性和特异性.
Devi等人[155]提出了一种减少结肠息肉CADx计算时间的方法:在CAD系统中, 首先用大津法分割结肠组织, 用K-Means聚类抽取特征; 最后, 用SVM和ANN利用特征对病变区域分类.系统的学习效率很高, 能够为临床诊断决策提供有关的额外信息.与已有的系统相比, 此系统有更高的学习率和更小的均方误差, 而且计算用时也更少.
4.2 基于CTC图像的CAD系统临床医学光学结直肠镜是目前息肉检测和去除的主要工具, 不过, 由于它有侵入性, 对于50岁以上的人群筛查, 它是被禁止使用的.作为替代, CT结直肠成像(CTC)技术逐渐发展起来, 对息肉的检测展现出较好的性能[156, 157].CTC也叫虚拟结肠镜, 是一种用CT成像在经过清洗和空气扩张过的结肠上检测结肠息肉的技术.大多数CTC CAD系统由在下3步组成:从CTC图像中分割出结肠; 从抽取的结肠中检测息肉ROI; 从息肉候选区域中去除假阳性或分类为良性/恶性病变.
4.2.1 结肠分割对CT图像的准确和自动的分割, 是CT结肠造影中许多临床应用的关键步骤, 在结肠息肉计算机辅助诊断检测中也是如此.结肠分割实际上是指结肠壁的分割, 是CAD方案的基本步骤.结肠壁的理想分割, 对于整个CAD方案的性能表现至关重要.如果结肠部分错过或小肠和一些其他结肠结构被错误地包括在内, 那么后面的息肉检测和分类的质量将受到明显的影响[158].
Yang等人[159]引入了图推理方法以移除结肠之外的部分, 应对结肠分割中的两个挑战——结肠折叠和附着的非结肠结构去除, 达到高质量的分割.首先, 把每一个3D空气填充的对象分解成一组三维区域, 用区域层特征训练一个分类器以区分结肠区和非结肠区域, 去除明显分离的非结肠部分; 然后, 用一个先验拓扑约束进行全局建模, 去除剩下的未附着非结肠部分.对于附着的非结肠部分, 在每一个3D对象内部的区域, 用一个级联的条件随机场建模, 用生成的图形进行剪除.尽管在小区域分类时性能稍有欠佳, 但实验结果证实:从各个区域之间依赖的角度建模, 性能超出了单纯从对象识别层次的差别学习方法.同时, 这种方法也能提高依赖结肠分割的其他应用程序的性能, 如fly-through、CAD和俯卧/仰卧配准等.Lu等人[160]也为这两个问题提供了可行的解决方案:对于第1个问题, 通过最短路径选择算法解决, 整个过程中没有任何额外的操作正确连接结肠的断开段; 对于第2个问题, 环和结肠附着物, 如骨骼和小肠, 他们引入了基于互补的测地线距离图(CGDM)算法剔除.算法的实验结果与医生的手动分割结果达到了90.56%的正确重合率.
4.2.2 息肉检测息肉是从大肠(结肠或直肠)的粘膜突出并伸到肠道(管腔)中的组织的异常生长.一些息肉是扁平的, 其他的有蒂.大部分结肠癌是从息肉发展而来的, 不过这需要5年~15年的恶性转变.结肠息肉的大小是一个与恶性风险相关的生物标志物, 也对其临床处理措施起着指导作用.虚拟结肠镜工作组的专家的一致共识是, 6mm~10mm的阈值是尤其值得关注的[161].
Chen等人[162]发现, 结肠息肉的实际大小比在CTC下观察到的平均要小1.2mm左右.为了更准确地检测结肠壁上的小息肉(5mm~8mm), Wang等人[161]展开了一项结肠结构分解的息肉检测CAD方案研究.为了获得更好的检测结果, 研究中使用了相对的全局和局部方法.复杂的结肠被分离成多个均匀一致的分段, 通过对立体图像的二阶导数分析, 将Haustral折叠和Haustral壁分成两种独立形态的形状.这样, 息肉检测看起来就像是在相对简单的表面上发现某些异常突起, 因此, 立体图像中的小息肉形态学的突出特征将被增强.实验中选取了60个患者病例, 得到了0.99的平均灵敏度.
Song等人[163]展示了一个基于Haralick纹理分析模型的息肉虚拟病理学模型, 探索利用从图像密度分布的高阶微分或振幅得到纹理特征, 如梯度和曲率, 模仿病理学振幅.一阶特征从图像密度分布梯度中抽取, 二阶特征从图像密度分布曲率导出.通过在一个3x3×3的块中增加9个方向, 将2D Haralick特征模型扩展成3D, 同时, 使用3D密度图和密度的相关信息及高阶关系构建了一个新的共生矩阵.最终的实验结果表明, 通过模型计算出的高阶纹理特征包含更多病理诊断所需的判别信息.
张国鹏等人[164]在虚拟结肠镜系统中利用三维纹理特征实现病灶的计算机辅助诊断, 并对几种常见的临床病理类型病灶和正常样本进行显著性分析.分析结果显示:除了增生型息肉和管状腺瘤两组之间区别不明显以外, 其余组两两之间均呈现显著性差异.此探索说明, 在虚拟结肠镜中用三维纹理特征对病灶进行分类, 具有实现计算机辅助诊断的可行性.
在文献[165]中, Sun等人对以前用于检测结肠镜检查视频中的息肉CAD算法做了改进.在以前的工作中, 他们应用了一个特征图像模型, 从各种视角和成像条件的结肠镜视频中提取表示息肉、正常组织、憩室等的特征, 使用条件随机场(CRF)模型进行分类.该模型考虑了结肠镜检查视频中存在的空间和时间邻接关系.在本文中, 使用从CNN、经过训练以识别结肠镜检查视频中相同图像类型的框架提取的特征作为图像特征描述符, 用作CRF分类器的输入.与以前的传统特征图像模型相比, CNN导出的特征显示出对视角和图像品质因素变化的更大的不变性.最后, 使用人和小鼠结肠镜检查数据报告新算法的测试结果, 达到了85%的敏感性和86%的特异性.尽管如此, Sun等人仍指出:此研究中仅把CRF单独地用于息肉检测, 也没有把视频中相邻帧的关系结合到CNN中.
4.2.3 去假阳性/分类CAD方案中一般在候选区域检测之后是有监督的分类[166], 候选检测的任务是通过包括尽可能多的疑似病变区域来实现高检测灵敏度.CAD中通常的分类方法是基于领域知识人工提取许多基于灰度、纹理、几何和其他性质的特征, 在此基础上设计和训练分类器进行分类.
并不是所有提取到的特征都有助于判别病变与非病变, 因此, 在有效分类器的设计中, 选择最具差异性的特征来区分病变与非病变是至关重要的.Xu等人[167]提出了一种基于顺序前向浮动选择(SFFS)和特征选择的方法, 以提高CT结肠镜(CTC)中息肉计算机检测分类器的性能.他们将SFFS与SVM分类器相结合, 选择能够最大化AUC的相关特征.在SFFS程序中提出了两种使用不同停止标准的方法:第1种方法搜索SFFS过程中允许的所有特征组合, 并选择最大化AUC值的子集; 第2种方法在SFFS执行期间对每个步骤进行统计检验, 如果AUC值的增加没有统计学显著性, 则其终止.通过比较发现, 第2种变体有较低的计算成本.最后, 他们用这些选择特征训练的两个SVM分类器, 分别在息肉的假阳性(FP)率为4.1和6.5时产生96%的检测敏感性.
对于诊断结肠息肉病变的恶性/良性的CAD, Hu等人[156]发现:对于支持向量机(SVM)、随机森林(RF)和线性判别分析(LDA), 每个分类器各有优缺点, 在不同的情况下性能变化较大.受此启发, 他们探索新的分类器, 突破单个分类器的局限性.通过对3种分类器进行不同的组合并实验测试, 最终发现, 3种分类器相结合, 分类性能是最好的.在通过计算机辅助诊断疑似病变时, 特征分类扮演着重要的角色.在CAD系统中, 随机森林(RF)是广泛集成的分类学习算法, 由kNN和wkNN分类器得到的位置指数(LI)有很高的判别准确度, Hu等人[168]设计了一种算法, 把LI集成到RF中.对于一个新的待检测样例, 先用位置指数尺度检测其LI得分, 如得分在边缘阈值外, 则明显属于某一类; 如在边缘阈值内, 则进一步用建立的RF模型间断其类别.这样既提高了运算速度, 也得到了更高的系统分类性能.
4.3 其他结直肠医学图像CAD系统自体荧光是指生物组织被一定波长的激发光激发后, 处于激发态的分子在下降到基态过程中, 以光量子的形式释放出所吸收的能量, 即荧光.荧光的产生与生物体特定的分子结构有关, 不同的生物组织由于其分子结构的不同, 其所对应的荧光光谱也不同.这种特异性的自体荧光可作为内镜下确定活检部位的参照指标, 有助于提高早期癌症的检出率.
Aihara等人[169]开发了一个在自体荧光内镜(AFE)检查时, 通过实时颜色区分结肠肿瘤病变和非肿瘤病变的CAD系统.在AFE检查中, 对病变区域色调采样, 绿/红比小于1.01的判断为肿瘤, 大于1.01则相反.研究实验结果表明, 系统可以明显减少对非肿瘤病变的误诊.
5 前列腺医学图像CAD系统前列腺癌(PCa, 如图 5所示[170])是世界范围内最常见的男性癌症之一, 约占男性癌症总量的21%.致死率在所有癌症中排在第三位, 占8%左右.美国癌症学会预测:2016年, 美国新增的前列腺癌症患者约为180 890, 而因此死亡的人数不少于26 000人[35].

|
Fig. 5 Prostatic cancer 图 5 前列腺癌 |
5.1 基于MRI图像的CAD系统
减少前列腺癌致死率的主要方法是早期发现并加强治疗.数字直肠检查(DRE)只能鉴别到后周围区域的肿瘤, 因此不能检测到发病于前部周边、中心区域和过渡区域的许多肿瘤[171], 小的肿瘤也不能被触诊检出.基于血液检查的前列腺特异抗原(PSA)测试伴随着过度诊断和过度治疗的高风险[172].尽管经直肠超声(TRUS)引导下的随机系统的活检易于发现小而危险度低的癌症, TRUS比MP-MRI更方便、成本更低, 但其低灵敏度不适全筛选大量患者群体[173].
磁共振(MR)能够提供功能性的组织信息以及解剖信息.多参数核磁技术的T1加权(T1-weighted)、T2加权(T2-weighted)、弥散加权(diffusion-weighted)、动态对比增强(dynamic contrast-enhanced)是目前用在前列腺诊断的常用影像方法[174].
在基于MRI的CAD系统流程中, 预处理环节的主要目的是减少噪声, 去除伪影和标准化信号密度; 由于后面的操作都集中在前列腺上, 所以需要把前列腺从每一个MRI模式中分割出来; 配准是把所有分割出来的MRI图像匹配重叠到同一个参考帧上, 以消除由于病人移动或不同获取参数引起的不对齐问题; 经过上面的步骤数据正则化以后, 就可以抽取特征并分类这些数据, 得到可能病变的位置(CADe)或这些病变的恶性程度(CADx)[175].
5.1.1 预处理和配准多参数MRI(MP-MRI)不但能够通过T2-w提供图像的形态学信息, 还能利用DW-MR图像和DCE-MR图像估计组织的生理学特性, 所以逐渐成为潜在的前列腺癌替代筛选方法.Giannini等人[172]展示了一个基于MP- MRI分析的前列腺癌CAD系统, 在图像配准阶段进行如下两个步骤.
(1) DWI-T2w配准.首先, 应用刚性配准来校正主要由运动或涡流引起的可能的平移和缩放伪像.对膀胱通过应用于ADC图上的分水岭算法在DW上自动分割, 并且通过k均值聚类在T2-w图像上自动分割, 发现仿射变换耦合在T2-w和DW图像上的膀胱分割掩模的边界点.然后, 执行具有变形场(T)的非刚性配准步骤, 从由该算法自动分段的卷的上边界开始.假设由磁场不均匀性引起的像素偏移特别是在相位编码方向上发生, 并沿着与卷的距离线性减小, T沿着垂直方向被建模为线性衰减场.
(2) DCE-T2w配准.选择基于相互信息(MI)的相似性作为度量, 使用0阶(boxcar)B样条内核计算图像概率密度函数(PDF); 同时, 为确保平滑度, 用三阶B样条核计算运动图像强度PDF, 通过试错逐渐改进迭代计算, 直到满足终止条件为止.配准之后的数据集中的每一个像素都可以表示成由图像密度、量化的生理信息值和表观扩散系数(ADC)值等特征组成的向量表示.接着, 所有这些参数被送到分类器中, 这个分类器最大化对真阳性的检测, 同时使假阳性良性区域最小化.
Liu等人[176]探索了在CADx中使用一个基于T2加权、弥散加权和动态增强MRI图像的有监督的统计学习方法.系统首先对待处理图像做预处理, 对T2WI图像按比例归一化, 对DWI图像逐体素计算出表观扩散系数(ADC)的映射图, 对于DCE图像, 用Tofts双室药代动力学模型生成Ktrans图.然后, 使用存储在DICOM图像头部的坐标信息来配准T2WI、ADC和Ktrans图.每个图像切片被认为是在标题信息中给出的原点和方向的平面.为了找到对应的体素, 最高分辨率序列(T2WI)中的体素被投射到其他成像模式中最接近的切片.最后提取每种模式图像的局部统计学特征, 包括密度、形状和活检靶的纹理信息, 用监督方法训练SVM.实验结果表明, 使用三模MRI特征的算法的CADx性能相对于仅用T2加权特征的传统算法, 表现出明显的提升.
5.1.2 分割前列腺医学图像分割是临床和图像处理工作流程中的重要一步.在临床设置中, 前列腺分割用于诸如放射治疗、前列腺特异性抗原(PSA)密度的计算, 也是前列腺体积和计算诊断上的指标.在图像处理中, 器官的分割通常是强制性的第1步, 这使得后续的算法可以集中在感兴趣的区域上.这也会降低算法复杂性和计算时间.Litjens等人[177]对他们的多图集分割技术进行了评估.多图集算法的过程由两个不同的步骤组成:首先, 使用局部互信息作为度量, 将图集配准到未知的案例, 将获得的变换应用于图集的分割; 然后, 使用选择性和迭代方法进行性能等级估计(SIMPLE)算法来合并图集标签, 将变换的分割融合, 以获得感兴趣的器官的最终二进制分割.通过在前列腺MR图像分割挑战(PROMISE12)的数据上的数据测试, 得到了中值Dice系数为0.83.
活动外观模型(AAM)使用一组解剖标志来定义每个对象的形状, 然而解剖标志可能难以识别, 而且传统的AAM只允许对单个感兴趣的对象进行分割.MLA可以同时分割多个对象, 并利用多个级别而不是解剖标记定义形状.Toth等人[178]提出了流行的(AAM)算法的改进方案, 构建一个分层分割框架, 称为多级集AAM (MLA).作为解剖标志独立的AAM, MLA允许确定多个对象的级别, 并允许它们与图像强度耦合, 使得MLA具有在新图像中同时分割多个感兴趣对象的灵活性.这个构架通过给定的前列腺分割取得领域的特定属性, 来帮助驱动嵌入在前列腺内的其他组织的分割.他们应用MLA从一组40个直肠内T2加权MRI图像中分割前列腺囊、前列腺周围区(PZ)和前列腺中央腺(CG), 产生的平均DSC值分别为0.89, 0.84和0.76.
高效、准确地提取前列腺, 特别是其临床3D MR图像上的子区域, 对图像引导的前列腺干预和前列腺癌的诊断非常有意义.Qiu等人[179]提出了一种新的多区域分割方案, 可以同时定位前列腺及其两个主要子区域的边界:中央腺体和周边区域.该方案利用空间区域一致性的先验知识, 并采用定制的前列腺外观模型来同时分割多个有临床意义的区域.通过凸松弛来解决所产生的具有挑战性的组合优化问题.方案引入了一种新的空间连续流最大化模型, 并展示了其区域一致性约束条件下凸松弛优化问题的二元性.此外, 他们提出的连续最大流模型自然产生了一种新型高效的连续最大流量算法, 在数值方面具有很大的优势, 并可以很容易地在GPU上实现.最后, 他们通过15个T2加权3D前列腺MR图像的实验, 证明此方案的表现是非常有前景的.类似地, Yuan等人[180]提出了耦合水平集/轮廓演化方法, 可以同时定位前列腺区域及其两个子区域, 引入了前面开发的凸松弛技术, 以全局优化方式共同演化两个耦合水平集.与传统的水平集方法相反, 他们证明了两个耦合水平集可以在每个离散的时间帧同时移动到它们的全局最优位置, 同时保持空间的表面间一致性; 他们通过凸松弛解决了在每个离散时间演化过程中产生的复杂的组合优化问题, 并展示了其全局和精确的最优性, 同时引入了新的耦合连续最大流模型, 展示了其对所研究的凸松弛优化问题的二元性区域约束.通过10个T2加权3D前列腺MRI的实验, 表现出其良好的分割性能.
5.1.3 特征选取和分类Andrik等人[181]描述了一个结合T2加权磁共振引导、对周围区域进行前列腺活检的CAD系统.他们把周边区域分成4个子区域, 通过对比子区域灰度直方图与恶性灰度直方图模板乘积和及子区域与正常灰度直方图模板乘积和的总概率矩阵来估计异常程度.Emilie等人[182]对一个基于多参数核磁共振图像确定前列腺癌在外围区域发生可能性测量的CAD系统进行了评估.从灰度、纹理和梯度这3个方面共140个特征中, 通过学习自动选取特征.实验使用t-test特征选择算法, 结合SVM分类.模仿专家区分恶性病变和疑似组织, CADx的性能AUC显示出, 其在周边区域辅助检测癌症方面是有潜力的.
对于MRI图像的计算机辅助诊断, Zhou等人[183]尝试了基于集成SVM的多特征前列腺CAD系统研究.方案如下:(1)提取MRI图像中前列腺ROI的统计学、纹理和不变矩这3类特征; (2)在每类特征空间中选取不同的特征子集, 结合SVM形成子函数, 并通过投票算法对每类特征中的子函数整合; (3)对步骤(2)的结果通过投票再次整合; (4)以前列腺患者的MRI图像作为原始数据, 将此集成的SVM用于辅助诊断.实验结果显示, 该算法能够有效提高前列腺癌的识别准确性.
在计算机辅助诊断领域, 深度学习已经显示了巨大的潜力.但在许多应用中, 大型数据集不可用, 这使得复杂的深度学习神经网络(DNN)的训练困难.Chen等人[184]展示了通过迁移学习, 用一个前列腺X挑战赛提供的有限数据快速重新训练最新的DNN模型.训练数据由330例病灶组成, 其中仅78例临床显著.他们引入了ImageNet预先训练的inceptionV3和Vgg-16模型.针对样本量不足, 使用随机旋转和平移进行数据放大; 对数据不平衡问题, 他们对阳性样例实施采样以增加样本量, 对阴性样例实施降采样以减少数据增加.他们还对预训练的模型在最终的全连接分类层进行修改, 以满足此分类任务的需求.对前列腺X检验数据, 两模型分别获得了0.81和0.83的AUC.他们还发现, 针对不同前列腺区域训练的模型具有不同的灵敏度, 如在合并结果之前, 应用比例因子会提高最终结果的AUC.
5.2 其他医学图像CAD系统成功治疗前列腺癌在很大程度上取决于早期诊断, 而其确诊要通过手动分析活检样本来确定[185].在美国, 每年进行100万次以上的前列腺活组织检查, 其中每一次都产生大约6个~14个组织样本.随后, 在病理学家的显微镜下分析这些样品中癌症的存在和分级.这些活检的约60%~70%是PCa阴性[186], 这意味着病理学家的大部分时间都用于检查良性组织.被鉴定为PCa的区域再进行Gleason评分, 基于样本中变异组织存在的模式反映肿瘤的恶性程度[187].精确的组织分级受到许多因素的阻碍, 包括病理学家的疲劳、应用的变异性和分级标准的解释以及假PCa(良性增生, 高级别前列腺上皮内瘤变)外观的良性组织的存在[188, 189].引入能够自动、准确和可重复地在图像上发现可疑PCa区域的定量“第二读者”来减轻这些困难[190]是必要的.
Doyle等人[191]提出了一个增强的贝叶斯多分辨率(BBMR)系统来识别数字活检片上PCa的区域:系统先将整个幻灯片图像分解成包含多个分辨率级别的图像金字塔; 随后, 在更高分辨率水平下更详细地检查以较低分辨率水平通过贝叶斯分类器识别为癌症的区域, 从而允许对大图像的快速和有效的分析.在每个分辨率级别, 使用AdaBoost集合方法从超过900个一阶统计、二阶共生和Gabor滤波器特征的池中选择10个图像特征.在从58例患者中获得的100张图像的实验中, BBMR方案产生了非常理想的分类结果.
6 医学图像CAD的性能评估 6.1 医学图像数据集性能评估是CAD系统研究的一个重要环节, 使用公共的医学影像样本数据集是各类CAD系统性能有效和客观、公平地评估的基础[192].样本集的建立是非常困难的工程, 大多数研究机构的数据集不对外公开, 目前研究对比常用的医学影像公开样本数据集见表 1.
| Table 1 Open available medical image databases 表 1 公开的医学影像数据集 |
6.2 评估方法
通常用敏感度(sensitivity)、特异度(specificity)[193]、假阳性率(FPR)、准确率(accuracy)、精确度(precision)、受试者操作特征曲线和混淆矩阵等指标来衡量CAD系统的检测/诊断性能.对于医学图像的ROI区域, 可以用阳性(positive)或阴性(negative)来描述其为病变或非病变, 对其判断的正确与否可以用真(true)或假(false)表示, 那么CAD系统输出的检测诊断结果可能为:(1)真阳性(TP)——诊断为阳性, 对象真值也是阳性; (2)真阴性(TN)——诊断为阴性, 对象真值也是阴性; (3)假阳性(FP)——诊断为阳性, 对象真值为阴性; (4)假阴性(FN)——诊断为阴性, 对象真值为阳性.ROI的真实值与CAD判断结果的交叉对应关系见表 2.
| Table 2 Comparison table of performance evaluation metrics 表 2 诊断性能评价指标对照表 |
敏感度(sensitivity)、特异度(specificity)、假阳性率(FPR)、准确率(accuracy)、精确度(precision)的计算公式分别表达为
| $ Sensitivity{\rm{ = }}TPR{\rm{ = }}TP/\left( {TP + FN} \right) \times 100\% $ | (1) |
| $ Specificity = TNR = TN/\left( {TN + FP} \right) \times 100\% $ | (2) |
| $ FPR = 1-TFR = FP/\left( {TP + FN} \right) \times 100\% $ | (3) |
| $ Accuracy = \left( {TP + TN} \right)/\left( {TP + TN + FP + FN} \right) \times 100\% $ | (4) |
| $ Precision = PPV = TP/\left( {TP + FP} \right) \times 100\% $ | (5) |
| $ NPV = TN/\left( {TN + FN} \right) \times 100\% $ | (6) |
敏感度(sensitivity)又称为真阳性率(TPR), 为异常区域中被正确识别为阳性的比率, 是衡量一个系统真阳性识别性能的尺度.敏感的系统能够识别出要找的阳性个体, 同时很少产生假阴性.特异度(specificity), 又称为真阴性率(TNR), 是正常类别中被正确识别为阴性的比率.特异度衡量一个系统能够在多大程度上把阴性个体正确地识别出来或挑出那些不是期望的个体.假阳性率(FPR)是真阴性类别中被识别为阳性的比率.一个好的系统有很高的敏感度和特异度, 同时假阳性率极低.准确率(accuracy)是对象中真阳性和真阴性个体被正确识别的比率.精确度(precision)也称为阳性预测值(PPV), 是被识别为阳性个体中真阳性的比率.阴性预测值(NPV)是被识别为阴性的个体中的真阴性的比率.
受试者操作特征曲线是基于统计学决策理论产生的, 广泛应用于CAD系统评估中[194].ROC曲线把系统的真阳性率(TPR)表示为1-sensitivity的函数, 用ROC曲线下面积(AUC)度量CAD系统的整体性能.AUC越接近1, 表示系统的性能越好, 如图 6所示, 系统CAD1的性能优于CAD2的性能.当AUC为1时, 系统是完美的, 它能将所有的样本都能正确地分类.一般情况下, 当系统的真阳性率值增大时, 相应的假阳性率值也会增大, 所以系统的AUC一般不会达到1.当ROC曲线与如图 6所示的对角线重合时, 表示系统决策成功的机率为50%, 这样的系统是不成功的.
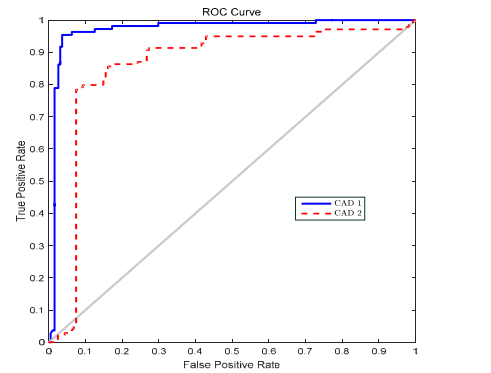
|
Fig. 6 Receiver operating characteristic curve (ROC curve) 图 6 受试者操作特征曲线(ROC curve) |
当一幅图像上有多个异常结果需要定性、定量、定位检测分析时, ROC方法无法完成评价任务.Bunch等人[195]提出了另外一种常用的评估方法:自由响应ROC曲线(FROC).这个曲线描述了敏感度随每张图像(或区域)上的假阳性率变化的函数, 并将不同阈值下的这两个检测量的统计值绘制在同一张图中以评价系统的性能.类似于ROC, 曲线越靠近图的左上角, 表明系统的检测性能越好.FROC曲线有两种表现形式, 如图 7所示(其中, u=1-e-λ, λ为每张图片(或区域)上的假阳性数).
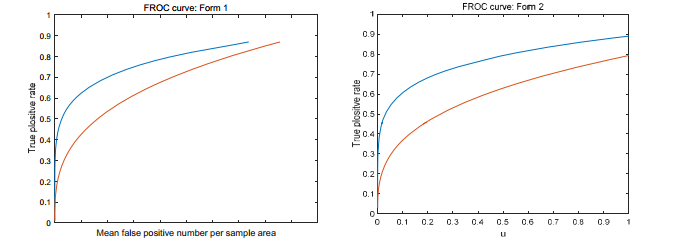
|
Fig. 7 Free-Response receiver operating characteristic curve (FROC curve) 图 7 自由响应ROC曲线(FROC curve) |
混淆矩阵也是评价CAD系统性能的一种常用方法[196].假设对于N类模式的分类任务, 识别数据集D包括T0个样本, 每类模式分别含有T1个数据(i=1, …, N).对于某个CAD系统C, cmij表示第i类模式被CAD系统C判断成第j类模式的数据占第i类模式样本总数的百分比率.
| $ CM(C, D) = \left[{\begin{array}{*{20}{c}} {c{m_{11}}}&{c{m_{12}}}&{...}&{c{m_{1i}}}&{...}&{c{m_{1N}}}\\ {c{m_{21}}}&{c{m_{22}}}&{...}&{c{m_{2i}}}&{...}&{c{m_{2N}}}\\ \vdots & \vdots & \vdots & \vdots & \vdots & \vdots \\ {c{m_{i1}}}&{c{m_{i2}}}&{...}&{c{m_{ii}}}&{...}&{c{m_{iN}}}\\ \vdots & \vdots & \vdots & \vdots & \vdots & \vdots \\ {c{m_{N1}}}&{c{m_{N2}}}&{...}&{c{m_{Ni}}}&{...}&{c{m_{NN}}} \end{array}} \right] $ | (7) |
混淆矩阵中, 元素的行下标对应目标的真实属性, 列下标对应分类器产生的识别属性.对角线元素表示各模式能够被分类器C正确识别的百分率, 而非对角线元素则表示发生错误判断的百分率.在理想情况下, 如果每个样本的预测类别都是正确的, 混淆矩阵就变成一个对角阵.
本文引文中提到的CAD系统性能评价使用评价方法或指标分布如图 8所示.
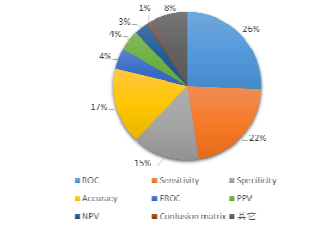
|
Fig. 8 Distribution chart of performance evaluation metrics used in the cited articles 图 8 本文引文所用性能评价指标分布图 |
7 系统所用算法和特征汇总
基于医学图像的计算机辅助检测/诊断技术已经成为国内外研究的热点之一, 相关的理论和方法也得到不断发展和完善, 部分成果在实际医疗诊断中得到应用.优秀的算法和恰到好处的特征选择是卓越CAD系统的基础.表 3汇总了本文引文中2012年以来的一些CAD系统在不同环节所用的主要算法.表 4列举了CAD系统中在假阳性去除或ROIs诊断分类时所用到的特征.
| Table 3 Algorithms used in CAD systems 表 3 CAD系统所用算法 |
| Table 4 Features used in classification or false positive reducement 表 4 去假阳性或分类时所用特征 |
8 面临的问题和研究展望
近些年, 越来越多的CAD系统被提出来, 但由于医学图像本身内容结构复杂、医学征象标准库建立困难等原因, 在医学图像CAD系统研究方面依然面临着挑战.
8.1 存在的问题(1) 有效标注的样本量太小.
在对文献中提到的CAD系统所用算法作统计的同时, 我们也对各CAD系统训练所用的训练图像样本进行统计, 发现样本图像数量超过5 000的仅3个, 仅使用几十个或100个左右样本量的不算少, 而根据经验法则, 训练时用的数据越多, 系统性能就越好[130].对于非公开的基准库, 由于后面的研究不能使用, 这里没有列出.表 1是目前常见的一些公开样本库列表.CAD研究领域, 有标注的医学图像样本库是训练分类器必须的, 特别是深度学习算法需要大规模统一标准的样本.由于征象样本图像生成成本高, 目前征象库的规模都是百张到千张左右, 各库的产生标准不尽相同, 征象覆盖也不全面.对于训练鲁棒的视觉特征分类模型来说, 样本不充足, 更不能满足深度学习算法的应用.
(2) 系统的性能不好评估.
公用的基准库是公平、正确地对比衡量CAD系统性能的基础条件之一.目前, 医学类的公共征象库有美国肺部图像数据库联盟的LIDC/IDRI库、日本放射技术学会的JSRT库、弗雷德里克国家实验室的RIDER、ELCAP公用库、南佛罗里达州大学的DDSM、Mini-MIAS乳腺征象库、NCIA、TCIA Collection库、promise12前列腺库, 国内的有LISS[265]库.由于标注量和征象种类的限制, 这些库只能满足某些CAD系统的评估.而且大部分现有的CAD系统研究文献中的CAD性能评估也不是建立在这些公共的库上[266].有些研究是基于上面所提到的公共库做的, 但一般只选用了库中的一部分图像, 没有说明使用了哪些图像或选择使用的标准, 实验环境无法重现.大部分文献中的系统性能评估使用的是自己的基准库, 不同基准库图像的产生设备和电气条件不同, 库的规模和征象类别的比例等也不同.除此之外, 也没有一个通用、可行的性能评估流程标准.正确地衡量一个系统的性能是非常困难的.
(3) CAD系统应用于临床使用有很多困难.
首先, 由于身体器官的医学图像构成复杂, 如肺部器官多、内部结构显示多态化, 医学图像上各种组织灰度相近.乳腺的图像相对简单, 图像上没有其他器官、症状的干扰, 目前有少量临床的商用系统.一个实验室的CAD研究实验往往针对某一类征象, 而临床应用要检测所有征象, 甚至是几种疾病同时伴发的征像组合.由于医院的商用医学影像系统的接口不对外开放, 开发的CAD系统很难与医院医生所用的系统无缝结合.医院的日常医疗工作任务重, 医生没有单独的时间对系统试用评估.目前在医学图像领域开展的研究较多, 但临床应用难度较大.
(4) CAD系统的应用效果还不理想.
尽管有的系统实验结果性能非常好, 但这只是小样本量、特定案例下的测试, 一旦实验对象变成普通随机病例, 如临床的医学图像, 情况会复杂得多[267], 性能就会不令人满意.
目前, 商用的系统有IQQA-Chest、CyclopusCAD® mammo、SecondLook和ImageChecker等.尽管已取得了一些成果, 但CAD系统的临床检测和诊断的正确率还偏低, 不少临床研究显示, 现有的CAD方法或系统的应用效果并不明显[114, 268-270].Lee等人[271]对IQQA-Chest系统对观察者的性能的影响进行了评估, 结论是此系统也许会对提高观察者的性能有帮助.在讨论部分指出, 病例的选择也许是结节太明显了, 所以得到的观察者敏感度和特异度比以前的实验结果明显要高.Qin等人[47]对商用的CAD系统IQQA®Chest的临床应用效果进行了对比实验.讨论中指出:用5位医生独立阅片和参考CAD阅片, 两种方式之间差异的统计P>0.05, 没有统计学意义, 即CAD系统对胸片的诊断结果没有明显影响.CAD系统的平均阅片时间为80s, 略高于医生未使用CAD系统的平均阅片时间72s.Cascio等人在文献[134]中对两个商用的乳腺CAD系统的诊断性能进行了对比分析.CyclopusCAD® mammo和SecondLook®的总体敏感度分别为83.1%和66.2%.但文献[134]在讨论部分指出:实验中没有与放射科医生的诊断性能进行比较, 数据库的生成仅限于肿块直径为12±5mm, 微钙化病变簇直径为11±5mm的案例.Murakami等人[123]对SecondLook®做了回顾性分析, 结论显示, CAD系统有很高的敏感度.但他们没有评估CAD对工作流程的影响, 不能确定是否能够提高放射医生的工作效率.Taylor等人[135]在对R2 ImageChecker CAD系统在乳腺筛查中的作用进行评估时也讨论, 他们提到的几项测试研究都没有证据证明CAD在临床应用中是有效的.
8.2 未来展望基于医学图像的CAD系统研究还处于未成熟阶段, 有很多工作需要我们去探索和认识.对未来医学图像CAD系统的可能发展趋势, 我们展望如下.
(1) 既检测又诊断的CAD系统能够为医生提供更丰富的信息.
CADe系统只对病变进行检测, 而不做出诊断.对于医生来说, 临床应用程序不提供病变的影像学特征, 体现的信息是不全面的.CADx系统如果只显示病变的良恶性诊断结果而不显示检测到的病变信息也不完善.未来的CAD系统应该是CADe系统和CADx系统的功能结合.
(2) 与医院HIS系统或PACS系统相结合的临床应用.
目前的研究中基于实验室样本库的较多, 仅有少数商用系统[134, 272]的临床应用研究.临床应用是CAD系统的最终目标, 结合临床应用, 可以检测CAD系统在复杂应用场景下的鲁棒性和实用性.
(3) 半监督学习.
传统的分类器只使用标记数据进行训练, 但充足的标记样本通常难以获得(如医学图像)、昂贵或耗时, 因为它们需要经验丰富的注释人员的努力.同时, 未标记的数据可能相对容易收集, 但几乎没有办法加以使用[273].半监督学习通过使用标记的数据及大量未标记的数据来解决这个问题, 构建更好的分类器[274].由于半监督学习需要较少的人力且能达到更高的准确性[275], 所以研究其在医学图像CAD领域的应用[135, 249-251], 对解决训练样本不足的现状非常有意义.
(4) 每种算法都有其局限性, 目前的单一分类器都不能完全解决所有的问题或者达到应用系统的要求.
多算法融合, 综合运用各种方法的优势、扬长避短, 组合起来可以得到更高鲁棒性的系统[252].算法融合不是算法简单地相加或捆绑.用合适的策略把不同的算法有机结合起来,得到一个更高效、鲁棒的CAD系统,是值得关注的.
(5) 在CAD系统中引入专家系统的思想, 使用时医生可以与系统交互.
在分割、检测阶段, 医生对环节的输出结果进行更准确的修改.在分类阶段对分类正确的结果给予奖励, 对分类错误的结果给予惩罚.系统根据回馈结果再进行强化学习, 逐步达到完美的性能.
(6) 深度学习.
基于视觉语义的医学图像分类一直以来都是充满挑战的研究领域, 不仅待识别的图像种类繁多, 在每一类图像的内部也存在诸多变数, 包括光照变化、不匹配不对齐、形变、遮挡等因素.针对这些变数, 学者们做了各种各样的努力, 提出了各种各样的特征来应对这些变化, 如比较典型的SIFT和HOG特征.虽然这些特征能够很好地应对特定情况下的数据处理任务, 但这些特征的泛化能力有限[276].自2006年Hinton在Science上发表论文提出训练深层网络的新思路开始, 深度学习就在图像特征学习方面展示出了卓越的性能, 也产生了不同的深度学习框架, 如Overfeat、ResNet、GoogLeNet、Alexnet、VGG、DeepID-Net、network in network、SPP-net等.在2015年的ImageNet图像识别大赛中, 深度学习算法已经超过了人类对图像的识别水平.深层CNN从原始数据(如图像)自动获得中级和高级的抽象(特征)[277], 同时考虑上下文之间的关系[253], 能够实现端到端的目标检测[278], 有效地避免了由分割和手工设计特征提取模型带来的误差[255].近年来的研究结果表明, CNN提取的通用描述符在医学图像检测中也非常有效[94, 277].世界各地的医学图像分析小组正在快速进入该领域并取得了良好的成果[87, 253, 254, 279, 280].在此架构上出现的“迁移学习”[256]和“微调”[277, 257]技术一方面能够提高检测准确率, 另一方面也为医学图像研究领域样本不足问题提供了有效的解决方案.深度学习自动学习特征和高识别准确度的特性, 一定会在医学图像领域得到不凡的表现.
9 结论基于医学图像的CAD技术研究具有重要的医疗和社会价值.本文以不同的身体部位为主线, 按照医学图像产生的技术分类, 调研了2012年以来研究较多的各类医学图像CAD系统, 并进行多维度归纳梳理, 还有一些其他身体部位的医学CAD技术研究, 如脑部[258, 259]、肝部[260, 281]、骨骼[261, 282]和皮肤[48, 262].CAD实现方法与文中的系统有很多相似之处, 文献相对较为分散, 本文未作综述.作为一个具有很强实用价值的研究领域, 基于医学图像的CAD系统具有光明的发展前景, 目前, 在系统每一个环节的算法、统一完善的公用样本库、标准的性能评估指标流程体系和无缝高效的临床应用方面, 还有很多有待完善、深入和拓展的研究空间.
在很长一段时间内, 计算机辅助检测/诊断将是医学图像处理领域的研究热点.建立鲁棒和高性能的CAD系统, 能够更好地辅助医生对疾病的检测与诊断, 提高患者的生存率, 改善其生活质量, 具有广阔的应用前景.
附录英文缩写与全称对照表
| 缩写 | 全称 |
| A2INET | Adaptive artificial immune network |
| ACS | American cancer society |
| AFE | Autofluorescence endoscopy |
| ANN | Artificial neural network |
| AUC | Area under curve |
| bCT | Breast computed tomography |
| BFGS | Broyden-fletcher-goldfarb-shanno |
| BPNN | Back propagation neural network |
| CADe | Computer-Aided detection |
| CADx | Computer-Aided diagnosis |
| CBIR | Content-Based image retrieval |
| CC | Craniocaudal |
| CFS | Correlation-Based feature subset selection for machine learning |
| CLAHE | Contrast limited adaptive histogram equalization |
| CNN | Convolutional neural network |
| CRC | Colorectal cancer |
| CT | Computerized tomography |
| CTC | Ct colonography |
| CV | Chan-Vese |
| CXR | Chest X radiograph |
| DBN | Deep belief network |
| DDSM | The digital database for screening mammography |
| ELCAP | The end-use load and consumer assessment program |
| ELM | Extreme learning machine |
| ESN | Echo state network |
| FCM | Fuzzy c-means |
| FDR | Fisher linear discriminant ratio |
| FFNN | Feed-Forward neural network |
| FFT | Fast fourier transform |
| FLD | Fisher linear decriminant |
| FLDA | Fisher discriminant analysis |
| FP | False positive |
| FPR | False positive rate |
| FROC | Free-Response roc curve |
| GA | Genetic algorithm |
| GGO | Ground-glass opacity |
| GMM | Gaussian mixture model |
| GLCM | Gray-Level co-occurrence matrix |
| GLRLM | Gray level run length matrix |
| IDRI | The infectious disease research institute |
| JSRT | Japanese society of radiological technology |
| kNN | K-Nearest neighbor |
| LDA | Linear discriminant analysis |
| LI | Location index |
| LIDC | Lung image database consortium |
| LISS | Lung ct imaging signs |
| LVQ | Learning vector quantization |
| MFFNN | Multi-Layer feed forward neural network |
| MIAS | The mammographic image analysis society |
| MLO | Medio lateral-oblique |
| MLP | Multilayer preceptor |
| MR | Magnetic resonance |
| MRI | Magnetic resonance imaging |
| MTANN | Massive-Training artificial neuralnetwork |
| MTB | Mycobacterium tuberculosis |
| NC | Nearest centroid |
| NCIA | National cancer image archive |
| NN | Neural network |
| OC | Optical colonoscopy |
| PACS | Picture archiving and communication systems |
| PCA | Principal component analysis |
| PNN | Probabilistic neural network |
| PPV | Positive predictive value |
| RF | Random forest |
| RFNC | Relief-f non-correlated |
| RIF | Resistance to Rifampicin |
| ROC | Receiver operating characteristic |
| ROI | Regions of interest |
| RRF | Rocchio relevance feedback algorithm |
| SCLGM | Square centroid lines gray level distribution method |
| SDAE | Stacked denoising autoencoder |
| SFS | Sequential forward selection |
| SVM | Support vector machine |
| TCIA | The cancer imaging archive |
| TNR | True negative tate |
| TPR | True positive rate |
| VOI | Volume of intretest |
| wkNN | Weighted knn |
| [1] |
Spiegel PK. The first clinical X-ray made in America-100 years. AJR American Journal of Roentgenology, 1995, 164(1): 241–243.
[doi:10.2214/ajr.164.1.7998549] |
||
| [2] |
Tao DC. Medical imaging technology in the new century. Chinese Journal of Medical Imaging Technology, 2000, 16(1): 1–2(in Chinese with English abstract).
http://kns.cnki.net/KCMS/detail/detail.aspx?filename=zyxx200001000&dbname=CJFD&dbcode=CJFQ |
||
| [3] |
Tearney GJ, Brezinski ME, Bouma BE, Boppart SA, Pitris C, Southern JF, Fujimoto JG. In vivo endoscopic optical biopsy with optical coherence tomography. Science, 1997, 276(5321): 2037–2039.
[doi:10.1126/science.276.5321.2037] |
||
| [4] |
Hendee WR. The impact of future technology on oncologic diagnosis:Oncologic imaging and diagnosis. Int'l Journal of Radiation Oncology* Biology* Physics, 1983, 9(12): 1851–1865.
[doi:10.1016/0360-3016(83)90353-X] |
||
| [5] |
Bunge RE, Herman CL. Usage of diagnostic imaging procedures:A nationwide hospital study. Radiology, 1987, 163(2): 569–573.
[doi:10.1148/radiology.163.2.3550886] |
||
| [6] |
Sluimer I, Schilham A, Prokop M, van Ginneken B. Computer analysis of computed tomography scans of the lung:A survey. IEEE Trans. on Medical Imaging, 2006, 25(4): 385–405.
[doi:10.1109/TMI.2005.862753] |
||
| [7] |
Quekel LG, Kessels AG, Goei R, van Engelshoven JM. Miss rate of lung cancer on the chest radiograph in clinical practice. CHEST Journal, 1999, 115(3): 720–724.
[doi:10.1378/chest.115.3.720] |
||
| [8] |
Li F, Sone S, Abe H, MacMahon H, Armato SG, Doi K. Lung cancers missed at low-dose helical CT screening in a general population:Comparison of clinical, histopathologic, and imaging findings. Radiology, 2002, 225(3): 673–683.
[doi:10.1148/radiol.2253011375] |
||
| [9] |
Li Q, Li F, Suzuki K, Shiraishi J, Abe H, Engelmann R, Nie Y, MacMahon H, DoiK. Computer-Aided diagnosis in thoracic CT. In: Proc. of the Seminars in Ultrasound, CT and MRI. Elsevier, 2005. 357-363. [doi: 10.1053/j.sult.2005.07.001] |
||
| [10] |
Suzuki K, Sheu I, Epstein M, Kohlbrenner R, Lostumbo A, Rockey DC, Dachman AH. An MTANN CAD for detection of polyps in false-negative CT colonography cases in a large multicenter clinical trial: Preliminary results. In: Proc. of the Medical Imaging. Int'l Society for Optics and Photonics, 2008. [doi: 10.1117/12.769824] |
||
| [11] |
Jr Engle RL. Attempts to use computers as diagnostic aids in medical decision making:A thirty-year experience. Perspectives in Biology and Medicine, 1992, 35(2): 207–219.
[doi:10.1353/pbm.1992.0011] |
||
| [12] |
Doi K. Current status and future potential of computer-aided diagnosis in medical imaging. The British Journal of Radiology, 2014, 78(Suppl_1): 3–19.
http://www.ncbi.nlm.nih.gov/pubmed/15917443 |
||
| [13] |
Doi K. Computer-Aided diagnosis in medical imaging:Historical review, current status and future potential. Computerized Medical Imaging and Graphics, 2007, 31(4): 198–211.
[doi:10.1016/j.compmedimag.2007.02.002] |
||
| [14] |
Giger ML, Chan HP, Boone J. Anniversary paper:History and status of CAD and quantitative image analysis:The role of medical physics and AAPM. Medical Physics, 2008, 35(12): 5799–5820.
[doi:10.1118/1.3013555] |
||
| [15] |
Doi K, MacMahon H, Katsuragawa S, Nishikawa RM, Jiang Y. Computer-Aided diagnosis in radiology:Potential and pitfalls. European Journal of Radiology, 1999, 31(2): 97–109.
[doi:10.1016/S0720-048X(99)00016-9] |
||
| [16] |
Kerlikowske K, Carney PA, Geller B, Mandelson MT, Taplin SH, Malvin K, Ernster V, Urban N, Cutter G, Rosenberg R, Ballard-Barbash R. Performance of screening mammography among women with and without a first-degree relative with breast cancer. Annals of Internal Medicine, 2000, 133(11): 855–863.
[doi:10.7326/0003-4819-133-11-200012050-00009] |
||
| [17] |
Suzuki K. A review of computer-aided diagnosis in thoracic and colonic imaging. Quantitative Imaging in Medicine And Surgery, 2012, 2(3): 163–176.
[doi:10.3978/j.issn.2223-4292.2012.09.02] |
||
| [18] |
Becker HC, Jr Nettleton WJ, Meyers PH, Sweeney JW, Jr Nice CM. Digital computer determination of a medical diagnostic index directly from chest X-ray images. IEEE Trans. on Biomedical Engineering, 1964, BME-11(3): 67–72.
[doi:10.1109/TBME.1964.4502309] |
||
| [19] |
Lodwick GS, Haun CL, Smith WE, Keller RF, Robertson ED. Computer diagnosis of primary bone tumors:A preliminary report. Radiology, 1963, 80(2): 273–275.
[doi:10.1148/80.2.273] |
||
| [20] |
Lodwick GS, Keats TE, Dorst JP. The coding of roentgen images for computer analysis as applied to lung cancer. Radiology, 1963, 81(2): 185–200.
[doi:10.1148/81.2.185] |
||
| [21] |
Meyers PH, Jr Nice CM, Becker HC, Jr Nettleton WJ, Sweeney JW, Meckstroth GR. Automated computer analysis of radiographic images. Radiology, 1964, 83(6): 1029–1034.
[doi:10.1148/83.6.1029] |
||
| [22] |
Redman A, Lowes S, Leaver A. Imaging techniques in breast cancer. Surgery (Oxford), 2016, 34(1): 8–18.
[doi:10.1016/j.mpsur.2015.10.004] |
||
| [23] |
Hambrock T, Vos PC, Hulsbergen-van de Kaa CA, Barentsz JO, Huisman HJ. Prostate cancer:Computer-Aided diagnosis with multiparametric 3-T MR imaging-Effect on observer performance. Radiology, 2013, 266(2): 521–530.
[doi:10.1148/radiol.12111634] |
||
| [24] |
Kashikura Y, Nakayama R, Hizukuri A, Noro A, Nohara Y, Nakamura T, Ito M, Kimura H, Yamashita M, Hanamura N, Ogawa T. Improved differential diagnosis of breast masses on ultrasonographic images with a computer-aided diagnosis scheme for determining histological classifications. Academic Radiology, 2013, 20(4): 471–477.
[doi:10.1016/j.acra.2012.11.007] |
||
| [25] |
Mookiah MRK, Acharya UR, Chua CK, Lim CM, Ng EYK, Laude A. Computer-Aided diagnosis of diabetic retinopathy:A review. Computers in Biology and Medicine, 2013, 43(12): 2136–2155.
[doi:10.1016/j.compbiomed.2013.10.007] |
||
| [26] |
Petrick N, Sahiner B, Armato Ⅲ SG, Bert A, Correale L, Delsanto S, Freedman MT, Fryd D, Gur D, Hadjiiski L, Huo Z, Jiang Y, Morra L, Paquerault S, Raykar V, Samuelson F, Summers RM, Tourassi G, Yoshida H, Zheng B, Zhou C, Chan HP. Evaluation of computer-aided detection and diagnosis systemsa. Medical Physics, 2013, 40(8): No.087001.
[doi:10.1118/1.4816310] |
||
| [27] |
Zeng Q, Miao FF, Yu CHH, Zhang GS. Current status and trends of cancer incidence and mortality in China. Chinese Health Statistics, 2016, 33(2): 321–323(in Chinese with English abstract).
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=zgwstj201602046 |
||
| [28] |
Chen W, Zheng R, Baade PD, Zhang S, Zeng H, Bray F, Jemal A, Yu XQ, He J. Cancer statistics in China, 2015. CA:A Cancer Journal for Clinicians, 2016, 66(2): 115–132.
[doi:10.3322/caac.21338] |
||
| [29] |
American Cancer Society (ACS). Report on lung cancer. 2014. https://www.cancer.org/research/cancer-facts-statistics/all-cancer-facts-figures/cancer-facts-figures-2014.html |
||
| [30] |
Parveen SS, Kavitha C. A review on computer aided detection and diagnosis of lung cancer nodules. Int'l Journal of Computers & Technology, 2012, 3(3): 393–400.
http://citeseerx.ist.psu.edu/viewdoc/summary?doi=10.1.1.299.4528 |
||
| [31] |
Henschke CI, Yankelevitz DF, Libby DM, Pasmantier MW, Smith JP, Miettinen OS. Survival of patients with stage I lung cancer detected on CT screening. The New England Journal of Medicine, 2006, 355(17): 1763–1771.
[doi:10.1056/NEJMoa060476] |
||
| [32] |
Suleiman WI, Georgian-Smith D, Evanoff MG, Lewis S, McEntee MF. A comparison of Australian and USA radiologists' performance in detection of breast cancer. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2014. [doi: 10.1117/12.2042968] |
||
| [33] |
Wang H, Li L, Peng H, Han H, Song B, Wang Y, Gu X, Liang Z. A novel computer aided detection (CADe) scheme for colonic polyps based on the structure decomposition. In: Proc. of the Abdominal Imaging Computation and Clinical Applications. Springer-Verlag, 2013. 63-72. [doi: 10.1007/978-3-642-41083-3_8] |
||
| [34] |
Bogoni L, Cathier P, Dundar M, Jerebko A, Lakare S, Liang J, Periaswamy S, Baker ME, Macari M. Computer-Aided detection (CAD) for CT colonography: A tool to address a growing need. The British Journal of Radiology, 2014, 78 Spec No 1(Suppl_1): S57-S62. http://www.birpublications.org/doi/full/10.1259/bjr/25777270 |
||
| [35] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2016. CA:A Cancer Journal for Clinicians, 2016, 66(1): 7–30.
[doi:10.3322/caac.21332] |
||
| [36] |
DeSantis CE, Lin CC, Mariotto AB, Siegel RL, Stein KD, Kramer JL, Alteri R, Robbins AS, Jemal A. Cancer treatment and survivorship statistics, 2014. CA:A Cancer Journal for Clinicians, 2014, 64(4): 252–271.
[doi:10.3322/caac.21235] |
||
| [37] |
Firmino M, Morais AH, Mendoça RM, Dantas M, Hekis H, Valentim R. Computer-Aided detection system for lung cancer in computed tomography scans:Review and future prospects. Biomedical Engineering Online, 2014, 13(1): 1–16.
[doi:10.1186/1475-925X-13-1] |
||
| [38] |
Bogoni L, Ko JP, Alpert J, Anand V, Fantauzzi J, Florin CH, Mason D, Rom W, Shiau M, Salganicoff M, Naidich DP. Impact of a computer-aided detection (CAD) system integrated into a picture archiving and communication system (PACS) on reader sensitivity and efficiency for the detection of lung nodules in thoracic CT exams. Journal of Digital Imaging, 2012, 25(6): 771–781.
[doi:10.1007/s10278-012-9496-0] |
||
| [39] |
Kominami Y, Yoshida S, Tanaka S, Sanomura Y, Hirakawa T, Raytchev B, Tamaki T, Koide T, Kaneda K, Chayama K. Computer-Aided diagnosis of colorectal polyp histology by using a real-time image recognition system and narrow-band imaging magnifying colonoscopy. Proc. of the Gastrointestinal Endoscopy, 2016, 83(3): 643–649.
[doi:10.1016/j.gie.2015.08.004] |
||
| [40] |
Fujita H, Zhang X, Kido S, Hara T, Zhou X, Hatanaka Y, Xu R. An introduction and survey of computer-aided detection/diagnosis (CAD). In: Proc. of the 2010 Int'l Conf. on Future Computer, Control and Communication (FCCC 2010). 2010. 200-205. |
||
| [41] |
Dhara AK, Mukhopadhyay S, Khandelwal N. Computer-Aided detection and analysis of pulmonary nodule from CT images:A survey. IETE Technical Review, 2012, 29(4): 265–275.
[doi:10.4103/0256-4602.101306] |
||
| [42] |
Elakkia K, Narendran P. Survey of medical image segmentation using removal of Gaussian noise in medical image. Int'l Journal of Engineering Science, 2016, 6(6): 7593–7595.
[doi:10.4010/2016.1795] |
||
| [43] |
Jacobs C, van Rikxoort EM, Scholten ET, de Jong PA, Prokop M, Schaefer-Prokop C, van Ginneken B. Solid, part-solid, or non-solid? Classification of pulmonary nodules in low-dose chest computed tomography by a computer-aided diagnosis system. Investigative Radiology, 2015, 50(3): 168–173.
[doi:10.1097/RLI.0000000000000121] |
||
| [44] |
Sampat MP, Markey MK, Bovik AC, et al. Computer-Aided detection and diagnosis in mammography. Handbook of Image and Video Processing, 2005, 2(1): 1195–1217.
http://ee.sharif.edu/~miap/Files/MammographyReview1.pdf |
||
| [45] |
Nagata R, Kawaguchi T, Miyake H. A computer-aided diagnosis system for lung nodule detection in chest radiographs using a two-stage classification method based on radial gradient and template matching. In: Proc. of the 20136th Int'l Conf. on Biomedical Engineering and Informatics (BMEI). IEEE, 2013. 80-85. [doi: 10.1109/BMEI.2013.6746911] |
||
| [46] |
Htike ZZ, Naing WYN, Win SL, Khan S. Computer-Aided diagnosis of pulmonary nodules from chest X-rays using rotation forest. In: Proc. of the 2014 Int'l Conf. on Computer and Communication Engineering (ICCCE). 2014. 96-99. [doi: 10.1109/ICCCE.2014.38] |
||
| [47] |
Qin J, Bai HL, Liu C, Yu JQ, Zhang HJ, Zhang ZJ, Li W, Zhang L. Application of computer-aided diagnosis in early detection of pulmonary nodules based on digital chest radiograph. Journal of Biomedical Engineering, 2014, 31(5): 1117–1120(in Chinese with English abstract).
[doi:10.7507/1001-5515.20140210] |
||
| [48] |
Harrison M, Looper J, Armato SG. Adaptive thresholding of chest temporal subtraction images in computer-aided diagnosis of pathologic change. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2016. [doi: 10.1117/12.2217177] |
||
| [49] |
Coppini G, Miniati M, Monti S, Paterni M, Favilla R, Ferdeghini EM. A computer-aided diagnosis approach for emphysema recognition in chest radiography. Medical Engineering & Physics, 2013, 35(1): 63–73.
[doi:10.1016/j.medengphy.2012.03.011] |
||
| [50] |
Cao X, Zhang HX. Research of computer-aided diagnosis about pulmonary interstitial pathology based on wavelet decomposition. Applied Mechanics & Materials, 2014: 611–614.
[doi:10.4028/www.scientific.net/AMM.519-520.611] |
||
| [51] |
Muyoyeta M, Maduskar P, Moyo M, Kasese N, Milimo D, Spooner R, Kapata N, Hogeweg L, van Ginneken B, Ayles H. The sensitivity and specificity of using a computer aided diagnosis program for automatically scoring chest X-rays of presumptive TB patients compared with Xpert MTB/RIF in Lusaka Zambia. PloS One, 2014, 9(4): e93757.
[doi:10.1371/journal.pone.0093757] |
||
| [52] |
Team NLSTR, et al. Reduced lung-cancer mortality with low-dose computed tomographic screening. The New England Journal of Medicine, 2011, 365(5): 395–409.
[doi:10.1056/NEJMoa1102873] |
||
| [53] |
Wiemker R, Rogalla P, Blaffert T, Sifri D, Hay O, Shah E, Truyen R, Fleiter T. Aspects of computer-aided detection (CAD) and volumetry of pulmonary nodules using multislice CT. The British Journal of Radiology, 2005, 78 Spec No 1: S46-S56. http://www.birpublications.org/doi/full/10.1259/bjr/30281702 |
||
| [54] |
Austin J, Müller N, Friedman PJ, Hansell DM, Naidich DP, Remy-Jardin M, Webb WR, Zerhouni EA. Glossary of terms for CT of the lungs:Recommendations of the nomenclature committee of the fleischner society. Radiology, 1996, 200(2): 327–331.
[doi:10.1148/radiology.200.2.8685321] |
||
| [55] |
Henschke CI, Yankelevitz DF, Yip R, Reeves AP, Farooqi A, Xu D, Smith JP, Libby DM, Pasmantier MW, Miettinen OS. Lung cancers diagnosed at annual CT screening:Volume doubling times. Radiology, 2012, 263(2): 578–583.
[doi:10.1148/radiol.12102489] |
||
| [56] |
Xu DM, Yip R, Smith JP, Yankelevitz DF, Henschke CI. Retrospective review of lung cancers diagnosed in annual rounds of CT screening. American Journal of Roentgenology, 2014, 203(5): 965–972.
[doi:10.2214/AJR.13.12115] |
||
| [57] |
Messay T, Hardie RC, Rogers SK. A new computationally efficient CAD system for pulmonary nodule detection in CT imagery. Medical Image Analysis, 2010, 14(3): 390–406.
[doi:10.1016/j.media.2010.02.004] |
||
| [58] |
Darmanayagam SE, Harichandran KN, Cyril SRR, Arputharaj K. A novel supervised approach for segmentation of lung parenchyma from chest CT for computer-aided diagnosis. Journal of Digital Imaging, 2013, 26(3): 496–509.
[doi:10.1007/s10278-012-9539-6] |
||
| [59] |
Ashwin S, Ramesh J, Kumar SA, Gunavathi K. Efficient and reliable lung nodule detection using a neural network based computer aided diagnosis system. In: Proc. of the 2012 Int'l Conf. on Emerging Trends in Electrical Engineering and Energy Management (ICETEEEM). 2012. 135-142. [doi: 10.1109/ICETEEEM.2012.6494454] |
||
| [60] |
Kulkarni SG, Bagal SB. Techniques for lung cancer nodule detection: A survey. 2015. http://www.irjet.net/archives/V2/i9/IRJET-V2I9323.pdf |
||
| [61] |
Al-Tarawneh MS. Lung cancer detection using image processing techniques. Leonardo Electronic Journal of Practices and Technologies, 2012, 11(21): 147–158.
https://www.researchgate.net/publication/265998089_Lung_Cancer_Detection_Using_Image_Processing_Techniques |
||
| [62] |
Cui Y, Wang XP, Yu H, Chen L. Segmentation method for CT image of lung parenchyma. Computer Engineering and Design, 2015, 36(5): 1274–1278(in Chinese with English abstract).
http://or.nsfc.gov.cn/bitstream/00001903-5/96645/1/1000007202240.pdf |
||
| [63] |
Zhao YR, van Ooijen PM, Dorrius MD, Heuvelmans M, de Bock GH, Vliegenthart R, Oudkerk M. Comparison of three software systems for semi-automatic volumetry of pulmonary nodules on baseline and follow-up CT examinations. Acta Radiologica, 2013, 55(6).
[doi:10.1177/0284185113508177] |
||
| [64] |
Lee SLA, Kouzani AZ, Hu EJ. Automated detection of lung nodules in computed tomography images:A review. Machine Vision and Applications, 2012, 23(1): 151–163.
[doi:10.1007/s00138-010-0271-2] |
||
| [65] |
Firmino M, Angelo G, Morais H, Dantas MR, Valentim R. Computer-Aided detection (CADe) and diagnosis (CADx) system for lung cancer with likelihood of malignancy. Biomedical Engineering Online, 2016, 15(1): 2–17.
[doi:10.1186/s12938-015-0120-7] |
||
| [66] |
Mansoor A, Bagci U, Foster B, Xu Z, Papadakis GZ, Folio LR, Udupa JK, Mollura DJ. Segmentation and image analysis of abnormal lungs at CT:Current approaches, challenges, and future trends. RadioGraphics, 2015, 35(4): 1056–1076.
[doi:10.1148/rg.2015140232] |
||
| [67] |
Armato SG, Sensakovic WF. Automated lung segmentation for thoracic CT:Impact on computer-aided diagnosis. Academic Radiology, 2004, 11(9): 1011–1021.
[doi:10.1016/j.acra.2004.06.005] |
||
| [68] |
Chen N, Liu G, Liao Y, Ou C, Yong Y. Research on computer-aided diagnosis of lung nodule. In: Proc. of the 2014 IEEE Workshop on Electronics, Computer and Applications. 2014. 1019-1022. [doi: 10.1109/IWECA.2014.6845796] |
||
| [69] |
Krewer H, Geiger B, Hall LO, Goldgof DB, Gu Y, Tockman M, Gillies RJ. Effect of texture features in computer aided diagnosis of pulmonary nodules in low-dose computed tomography. In: Proc. of the 2013 IEEE Int'l Conf. on Systems, Man, and Cybernetics (SMC). 2013. 3887-3891. [doi: 10.1109/SMC.2013.663] |
||
| [70] |
Wang Q, Kang W, Wu C, Wang B. Computer-Aided detection of lung nodules by SVM based on 3D matrix patterns. Clinical Imaging, 2013, 37(1): 62–69.
[doi:10.1016/j.clinimag.2012.02.003] |
||
| [71] |
Shi Z, Xu B, Zhao M, Zhao J, Wang Y, Liu Y, Zhang M, He L, Suzuki K. A joint ROI extraction filter for computer aided lung nodule detection. Bio-Medical Materials and Engineering, 2015, 26(s1): S1491–S1499.
[doi:10.3233/BME-151448] |
||
| [72] |
Li S, Liu X, Yang A, Pang K, Zhou C, Zhao X, Zhao Y. A novel approach of computer-aided detection of focal ground-glass opacity in 2D lung CT images. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2013. [doi: 10.1117/12.2003594] |
||
| [73] |
Anirudh R, Thiagarajan JJ, Bremer T, Kim H. Lung nodule detection using 3D convolutional neural networks trained on weakly labeled data. In: Proc. of the SPIE Medical Imaging. 2016. [doi: 10.1117/12.2214876] |
||
| [74] |
Demir Ö, Çamurcu AY. Computer-Aided detection of lung nodules using outer surface features. Bio-Medical Materials and Engineering, 2015, 26(s1): S1213–-S1222.
[doi:10.3233/BME-151418] |
||
| [75] |
Zeiler MD, Fergus R. Visualizing and understanding convolutional networks. In: Proc. of the European Conf. on Computer Vision. Springer-Verlag, 2014. 818-833. [doi: 10.1007/978-3-319-10590-1_53] |
||
| [76] |
Wajid SK, Hussain A, Luo B. An efficient computer aided decision support system for breast cancer diagnosis using echo state network classifier. In: Proc. of the 2014 IEEE Symp. on Computational Intelligence in Healthcare and e-health (CICARE). IEEE, 2014. 17-24. [doi: 10.1109/CICARE.2014.7007829] |
||
| [77] |
Tourassi GD, Frederick ED, Markey MK, Jr Floyd CE. Application of the mutual information criterion for feature selection in computer-aided diagnosis. Medical Physics, 2001, 28(12): 2394–2402.
[doi:10.1118/1.1418724] |
||
| [78] |
Elfarra BK, Abuhaiba IS. New feature extraction method for mammogram computer aided diagnosis. Int'l Journal of Signal Processing, Image Processing and Pattern Recognition, 2013, 6(1): 13–36.
http://citeseerx.ist.psu.edu/viewdoc/summary?doi=10.1.1.637.4927 |
||
| [79] |
Chandrashekar G, Sahin F. A survey on feature selection methods. Computers & Electrical Engineering, 2014, 40(1): 16–28.
[doi:10.1016/j.compeleceng.2013.11.024] |
||
| [80] |
Pudil P, Novovičová J, Kittler J. Floating search methods in feature selection. Pattern Recognition Letters, 1994, 15(11): 1119–1125.
[doi:10.1016/0167-8655(94)90127-9] |
||
| [81] |
Yao X, Wang XD, Zhang YX, Quan W. Summary of feature selection algorithms. Control and Decision, 2012, 27(2): 161–166(in Chinese with English abstract).
http://kns.cnki.net/KCMS/detail/detail.aspx?filename=kzyc201202002&dbname=CJFD&dbcode=CJFQ |
||
| [82] |
Liu X, Ma L, Song L, Zhao Y, Zhao X, Zhou C. Recognizing common CT imaging signs of lung diseases through a new feature selection method based on Fisher criterion and genetic optimization. IEEE Journal of Biomedical and Health Informatics, 2015, 19(2): 635–647.
[doi:10.1109/JBHI.2014.2327811] |
||
| [83] |
Cao P, Yang J, Zhao D, Li W, Huang M, Zaiane O. A multi-kernel based framework for heterogeneous feature selection and over-sampling for computer-aided detection of pulmonary nodules. Pattern Recognition, 2017, 64(C): 327–346.
https://www.sciencedirect.com/science/article/pii/S0031320316303557 |
||
| [84] |
Camarlinghi N, Gori I, Retico A, Bellotti R, Bosco P, Cerello P, Gargano G, Lopez Torres E, Megna R, Peccarisi M, Fantacci ME. Combination of computer-aided detection algorithms for automatic lung nodule identification. Int'l Journal of Computer Assisted Radiology and Surgery, 2012, 7(3): 455–464.
[doi:10.1007/s11548-011-0637-6] |
||
| [85] |
Sun T, Zhang R, Wang J, Li X, Guo X. Computer-Aided diagnosis for early-stage lung cancer based on longitudinal and balanced data. PloS One, 2013, 8(5): e63559.
[doi:10.1371/journal.pone.0063559] |
||
| [86] |
Sun T, Wang J, Li X, Lü P, Liu F, Luo Y, Gao Q, Zhu H, Guo X. Comparative evaluation of support vector machines for computer aided diagnosis of lung cancer in CT based on a multi-dimensional data set. Computer Methods and Programs in Biomedicine, 2013, 111(2): 519–524.
[doi:10.1016/j.cmpb.2013.04.016] |
||
| [87] |
Sun W, Zheng B, Qian W. Computer aided lung cancer diagnosis with deep learning algorithms. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2016. [doi: 10.1117/12.2216307] |
||
| [88] |
Han F, Wang H, Song B, Zhang G, Lu H, Moore W, Zhao H, Liang Z. A new 3D texture feature based computer-aided diagnosis approach to differentiate pulmonary nodules. Medical Imaging: Computer-Aided Diagnosis, 2013. http://proceedings.spiedigitallibrary.org/proceeding.aspx?doi=10.1117/12.2007252 |
||
| [89] |
Han F, Wang H, Zhang G, Han H, Song B, Li L, Moore W, Lu H, Zhao H, Liang Z. Texture feature analysis for computer-aided diagnosis on pulmonary nodules. Journal of Digital Imaging, 2015, 28(1): 99–115.
[doi:10.1007/s10278-014-9718-8] |
||
| [90] |
Abdalla AS, Yusuf IA, Mohammed SHAA, Mahmoud MA, Mustafa ZA. A computer-aided diagnosis system for classification of lung tumors. Journal of Clinical Engineering, 2015, 40(3): 130–134.
[doi:10.1097/JCE.0000000000000098] |
||
| [91] |
Niehaus R, Raicu DS, Furst J, Armato Ⅲ S. Toward understanding the size dependence of shape features for predicting spiculation in lung nodules for computer-aided diagnosis. Journal of Digital Imaging, 2015, 28(6): 704–717.
[doi:10.1007/s10278-015-9774-8] |
||
| [92] |
Li QP, Liu H, Su ZY. Modified fuzzy clustering with partial supervision algorithm in classification and recognition of pulmonary nodules. Journal of Graphics, 2015, 36(2): 244–250(in Chinese with English abstract).
http://www.cnki.com.cn/Article/CJFDTOTAL-GCTX201502017.htm |
||
| [93] |
Suzuki K. Pixel-Based machine learning in medical imaging. Journal of Biomedical Imaging, 2012, 2012(4).
http://europepmc.org/articles/PMC3299341/ |
||
| [94] |
van Ginneken B, Setio AA, Jacobs C, Ciompi F. Off-the-Shelf convolutional neural network features for pulmonary nodule detection in computed tomography scans. In: Proc. of the 2015 IEEE 12th Int'l Symp. on Biomedical Imaging (ISBI). IEEE, 2015. 286-289. [doi: 10.1109/ISBI.2015.7163869] |
||
| [95] |
Tajbakhsh N, Gotway MB, Liang J. Computer-Aided pulmonary embolism detection using a novel vessel-aligned multi-planar image representation and convolutional neural networks. In: Proc. of the Int'l Conf. on Medical Image Computing and Computer-Assisted Intervention. Springer-Verlag, 2015. 62-69. [doi: 10.1007/978-3-319-24571-3_8] |
||
| [96] |
Ng K, Muttarak M. Advances in mammography have improved early detection of breast cancer. Journal of the Hong Kong College of Radiologists, 2003, 6(3): 126–131.
https://www.researchgate.net/publication/228756983_Advances_in_mammography_have_improved_early_detection_of_breast_cancer |
||
| [97] | |||
| [98] |
Scharcanski J, Jung CR. Denoising and enhancing digital mammographic images for visual screening. Computerized Medical Imaging and Graphics, 2006, 30(4): 243–254.
[doi:10.1016/j.compmedimag.2006.05.002] |
||
| [99] |
Lavanya R, Nagarajan N, Devi MN. Computer-Aided diagnosis of breast cancer by hybrid fusion of ultrasound and mammogram features. In: Proc. of the Artificial Intelligence and Evolutionary Algorithms in Engineering Systems. Springer-Verlag, 2015. 403-409. [doi: 10.1007/978-81-322-2135-7_43] |
||
| [100] |
Radovic M, Milosevic M, Ninkovic S, Filipovic N, Peulic A. Parameter optimization of a computer-aided diagnosis system for detection of masses on digitized mammograms. Technology and Health Care, 2015, 23(6): 757–774.
[doi:10.3233/THC-151034] |
||
| [101] |
Meenalosini S, Janet J, Kannan E. A novel approach in malignancy detection of computer aided diagnosis. American Journal of Applied Sciences, 2012, 9(7): 1020–1029.
[doi:10.3844/ajassp.2012.1020.1029] |
||
| [102] |
Jain A, Singh S, Bhateja V. A robust approach for denoising and enhancement of mammographic images contaminated with high density impulse noise. Int'l Journal of Convergence Computing, 2013, 1(1): 38–49.
[doi:10.1504/IJCONVC.2013.054659] |
||
| [103] |
Bhateja V, Urooj S, Misra M, Pandey A, Lay-Ekuakille A. A polynomial filtering model for enhancement of mammogram lesions. In: Proc. of the 2013 IEEE Int'l Symp. on Medical Measurements and Applications Proceedings (MeMeA). IEEE, 2013. 97-100. [doi: 10.1109/MeMeA.2013.6549714] |
||
| [104] |
Wang X, Li L, Liu W, Xu W, Lederman D, Zheng B. An interactive system for computer-aided diagnosis of breast masses. Journal of Digital Imaging, 2012, 25(5): 570–579.
[doi:10.1007/s10278-012-9451-0] |
||
| [105] |
Jen CC, Yu SS. Automatic detection of abnormal mammograms in mammographic images. Expert Systems with Applications, 2015, 42(6): 3048–3055.
[doi:10.1016/j.eswa.2014.11.061] |
||
| [106] |
Sharma S, Khanna P. Computer-Aided diagnosis of malignant mammograms using Zernike moments and SVM. Journal of Digital Imaging, 2015, 28(1): 77–90.
[doi:10.1007/s10278-014-9719-7] |
||
| [107] |
Al-Absi HRH, Samir BB, Sulaiman S. A computer aided system for breast cancer detection and diagnosis. In: Proc. of the 2014 Int'l Conf. on Computer and Information Sciences (ICCOINS). 2014. 1-4. [doi: 10.1109/ICCOINS.2014.6868355] |
||
| [108] |
Dhungel N, Carneiro G, Bradley AP. Deep learning and structured prediction for the segmentation of mass in mammograms. In: Proc. of the Int'l Conf. on Medical Image Computing and Computer-Assisted Intervention. 2015. 605-612. [doi: 10.1007/978-3-319-24553-9_74] |
||
| [109] |
Bozek J, Mustra M, Delac K, Grgic M. A survey of image processing algorithms in digital mammography. In: Proc. of the Recent Advances in Multimedia Signal Processing and Communications. Springer-Verlag, 2009. 631-657. [doi: 10.1007/978-3-642-02900-4_24] |
||
| [110] |
Oliver A, Torrent A, Lladó X, Tortajada M, Tortajada L, Sentís M, Freixenet J, Zwiggelaar R. Automatic microcalcification and cluster detection for digital and digitised mammograms. Knowledge-Based Systems, 2012, 28(2): 68–75.
[doi:10.1016/j.knosys.2011.11.021] |
||
| [111] |
Sahiner B, Chan HP, Hadjiiski LM, Helvie MA, Wei J, Zhou C, Lu Y. Computer-Aided detection of clustered microcalcifications in digital breast tomosynthesis:A 3D approach. Medical Physics, 2012, 39(1): 28–39.
http://www.ncbi.nlm.nih.gov/pubmed/22225272 |
||
| [112] |
Malar E, Kandaswamy A, Chakravarthy D, Dharan AG. A novel approach for detection and classification of mammographic microcalcifications using wavelet analysis and extreme learning machine. Computers in Biology and Medicine, 2012, 42(9): 898–905.
[doi:10.1016/j.compbiomed.2012.07.001] |
||
| [113] |
Tan WC, Wang JH, Cai HM, Yang X, Li L. The performance of computer-aided diagnosis for DCIS based on classification of clustered microcalcifications. Journal of Clinical Radiology, 2016, 35(9): 1352–1356(in Chinese with English abstract).
http://kns.cnki.net/KCMS/detail/detail.aspx?filename=lcfs201609017&dbname=CJFD&dbcode=CJFQ |
||
| [114] |
Tang J, Rangayyan RM, Xu J, El Naqa I, Yang Y. Computer-Aided detection and diagnosis of breast cancer with mammography:Recent advances. IEEE Trans. on Information Technology in Biomedicine, 2009, 13(2): 236–251.
[doi:10.1109/TITB.2008.2009441] |
||
| [115] |
Yoshikawa R, Teramoto A, Matsubara T, Fujita H. Automated detection of architectural distortion using improved adaptive Gabor filter. In: Proc. of the Breast Imaging. Springer-Verlag, 2014. 606-611. [doi: 10.1007/978-3-319-07887-8_84] |
||
| [116] |
Singh B, Jain V. Computer aided classification of architectural distortion in mammograms using texture features. Computer, 2015, 9(7).
https://waset.org/abstracts/29952 |
||
| [117] |
Da Cruz CF. Automatic analysis of mammography images[MS. Thesis]. Oporto: Engineering Faculty, Porto University, 2011.https://www.mendeley.com/research-papers/automatic-analysis-mammography-images/ |
||
| [118] |
Scutt D, Lancaster GA, Manning JT. Breast asymmetry and predisposition to breast cancer. Breast Cancer Research, 2006, 8(2).
[doi:10.1186/bcr1388] |
||
| [119] |
Rangayyan RM, Ayres FJ, Desautels JL. A review of computer-aided diagnosis of breast cancer:Toward the detection of subtle signs. Journal of the Franklin Institute, 2007, 344(3): 312–348.
[doi:10.1016/j.jfranklin.2006.09.003] |
||
| [120] |
Magna G, Jayaraman SV, Casti P, Mencattini A, Di Natale C, Martinelli E. Adaptive classification model based on artificial immune system for breast cancer detection. In: Proc. of the AISEM Annual Conf. (2015 XVⅢ). IEEE, 2015. 1-4. [doi: 10.1109/AISEM.2015.7066842] |
||
| [121] |
Kelder A, Zigel Y, Lederman D, Zheng B. A new computer-aided detection scheme based on assessment of local bilateral mammographic feature asymmetry-A preliminary evaluation. In: Proc. of the 201537th Annual Int'l Conf. of the IEEE Engineering in Medicine and Biology Society (EMBC). IEEE, 2015. 6394-6397. [doi: 10.1109/EMBC.2015.7319856] |
||
| [122] |
Wang X, Li L, Xu W, Liu W, Lederman D, Zheng B. Improving performance of computer-aided detection of masses by incorporating bilateral mammographic density asymmetry:An assessment. Academic Radiology, 2012, 19(3): 303–310.
[doi:10.1016/j.acra.2011.10.026] |
||
| [123] |
Murakami R, Kumita S, Tani H, Yoshida T, Sugizaki K, Kuwako T, KiriyamaKenta T, OkazakiKeiko H, IidaShunsuke Y, Tsuchiya H. Detection of breast cancer with a computer-aided detection applied to full-field digital mammography. Journal of Digital Imaging, 2013, 26(4): 768–773.
[doi:10.1007/s10278-012-9564-5] |
||
| [124] |
Taylor P, Given-Wilson R. Evaluation of computer-aided detection (CAD) devices. The British Journal of Radiology, 2005, 78 Spec No 1(925): S26–S30.
http://www.ncbi.nlm.nih.gov/pubmed/15917442 |
||
| [125] |
Zhang P, Verma B, Kumar K. A neural-genetic algorithm for feature selection and breast abnormality classification in digital mammography. In: Proc. of the 2004 IEEE Int'l Joint Conf. on Neural Networks. IEEE, 2004. 2303-2308. [doi: 10.1109/IJCNN.2004.1380985] |
||
| [126] |
Eltoukhy MM, Faye I, Samir BB. A statistical based feature extraction method for breast cancer diagnosis in digital mammogram using multiresolution representation. Computers in Biology and Medicine, 2012, 42(1): 123–128.
[doi:10.1016/j.compbiomed.2011.10.016] |
||
| [127] |
Choi JY, Kim DH, Plataniotis KN, Ro YM. Classifier ensemble generation and selection with multiple feature representations for classification applications in computer-aided detection and diagnosis on mammography. Expert Systems with Applications, 2016, 46(C): 106–121.
[doi:10.1016/j.eswa.2015.10.014] |
||
| [128] |
Bengio Y, Courville A, Vincent P. Representation learning:A review and new perspectives. IEEE Trans. on Pattern Analysis & Machine Intelligence, 2012, 35(8): 1798–1828.
[doi:10.1109/TPAMI.2013.50] |
||
| [129] |
Arevalo J, Gonzalez FA, Ramos-Pollan R, Oliveira JL, Lopez MAG. Convolutional neural networks for mammography mass lesion classification. In: Proc. of the Engineering in Medicine and Biology Society. 2015. 797-800. [doi: 10.1109/EMBC.2015.7318482] |
||
| [130] |
Kooi T, Karssemeijer N. Boosting classification performance in computer aided diagnosis of breast masses in raw full-field digital mammography using processed and screen film images. In: Proc. of the 2014. 2014. [doi: 10.1117/12.2042863] |
||
| [131] |
Zheng B, Sumkin JH, Zuley ML, Lederman D, Wang X, Gur D. Computer-Aided detection of breast masses depicted on full-field digital mammograms:A performance assessment. The British Journal of Radiology, 2012, 85(1014): 153–161.
[doi:10.1259/bjr/51461617] |
||
| [132] |
Verma B, Zakos J. A computer-aided diagnosis system for digital mammograms based on fuzzy-neural and feature extraction techniques. IEEE Trans. on Information Technology in Biomedicine, 2001, 5(1): 46–54.
[doi:10.1109/4233.908389] |
||
| [133] |
Dheeba J, Singh NA, Selvi ST. Computer-Aided detection of breast cancer on mammograms:A swarm intelligence optimized wavelet neural network approach. Journal of Biomedical Informatics, 2014, 49(C): 45–52.
[doi:10.1016/j.jbi.2014.01.010] |
||
| [134] |
Cascio D, Fauci F, Iacomi M, Raso G, Magro R, Castrogiovanni D, Filosto G, Ienzi R, Vasile MS. Computer-Aided diagnosis in digital mammography:Comparison of two commercial systems. Imaging in Medicine, 2014, 6(1): 13–20.
[doi:10.2217/iim.13.68] |
||
| [135] |
Peng L, Chen W, Zhou W, Li F, Yang J, Zhang J. An immune-inspired semi-supervised algorithm for breast cancer diagnosis. Computer Methods and Programs in Biomedicine, 2016, 134(C): 259–265.
[doi:10.1016/j.cmpb.2016.07.020] |
||
| [136] |
Yosinski J, Clune J, Bengio Y, Lipson H. How transferable are features in deep neural networks? In: Advances in Neural Information Processing Systems 27(NIPS 2014). 2014. 3320-3328.http://dl.acm.org/citation.cfm?id=2969197 |
||
| [137] |
Carneiro G, Nascimento J, Bradley AP. Unregistered Multiview Mammogram Analysis with Pre-Trained Deep Learning Models. Springer Int'l Publishing, 2015: 159–169.
[doi:10.1007/978-3-319-24574-4_78] |
||
| [138] |
Zaheeruddin Z, Jaffery Z, Singh L. Detection and shape feature extraction of breast tumor in mammograms. In: Proc. of the World Congress on Engineering, Vol. 2. 2012. 1-6.http://www.oalib.com/paper/2151587 |
||
| [139] |
Marcomini KD, Carneiro AO, Schiabel H. Development of a computer tool to detect and classify nodules in ultrasound breast images. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2014. [doi: 10.1117/12.2043048] |
||
| [140] |
Jalalian A, Mashohor SB, Mahmud HR, Saripan MIB, Ramli ARB, Karasfi B. Computer-Aided detection/diagnosis of breast cancer in mammography and ultrasound:A review. Clinical Imaging, 2013, 37(3): 420–426.
[doi:10.1016/j.clinimag.2012.09.024] |
||
| [141] |
Moon WK, Lo CM, Huang CS, Chen JH, Chang RF. Computer-Aided diagnosis based on speckle patterns in ultrasound images. Ultrasound in Medicine & Biology, 2012, 38(7): 1251–1261.
[doi:10.1016/j.ultrasmedbio.2012.02.029] |
||
| [142] |
Alam S, Feleppa E, Rondeau M, Kalisz A, Garra B. Computer-Aided diagnosis of solid breast lesions using an ultrasonic multi-feature analysis procedure. Bangladesh Journal of Medical Physics, 2013, 4(1): 1–10.
https://www.researchgate.net/publication/314400360_Computer-Aided_Diagnosis_of_Solid_Breast_Lesions_Using_an_Ultrasonic_Multi-Feature_Analysis_Procedure |
||
| [143] |
Huang Q, Yang F, Liu L, Li X. Automatic segmentation of breast lesions for interaction in ultrasonic computer-aided diagnosis. Information Sciences, 2015, 314(C): 293–310.
[doi:10.1016/j.ins.2014.08.021] |
||
| [144] |
Cheng JZ, Ni D, Chou YH, Qin J, Tiu CM, Chang YC, Huang CS, Shen D, Chen CM. Computer-Aided diagnosis with deep learning architecture:Applications to breast lesions in us images and pulmonary nodules in CT scans. Scientific Reports, 2016.
[doi:10.1038/srep24454] |
||
| [145] |
Ray S, Prionas ND, Lindfors KK, Boone JM. Analysis of breast CT lesions using computer-aided diagnosis: An application of neural networks on extracted morphologic and texture features. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2012, 8315(2): 145-150. [doi: 10.1117/12.910982] |
||
| [146] |
Kuo HC, Giger ML, Reiser I, Boone JM, Lindfors KK, Yang K, Edwards A. Level set segmentation of breast masses in contrast-enhanced dedicated breast CT and evaluation of stopping criteria. Journal of Digital Imaging, 2014, 27(2): 237–247.
[doi:10.1007/s10278-013-9652-1] |
||
| [147] |
Dromain C, Boyer B, Ferre R, Canale S, Delaloge S, Balleyguier C. Computed-Aided diagnosis (CAD) in the detection of breast cancer. European Journal of Radiology, 2013, 82(3): 417–423.
[doi:10.1016/j.ejrad.2012.03.005] |
||
| [148] |
Wang TC, Huang YH, Huang CS, Chen JH, Huang GY, Chang YC, Chang RF. Computer-Aided diagnosis of breast DCE-MRI using pharmacokinetic model and 3-D morphology analysis. Magnetic Resonance Imaging, 2014, 32(3): 197–205.
[doi:10.1016/j.mri.2013.12.002] |
||
| [149] |
Pang Z, Zhu D, Chen D, Li L, Shao Y. A computer-aided diagnosis system for dynamic contrast-enhanced MR images based on level set segmentation and ReliefF feature selection. In: Proc. of the Computational and Mathematical Methods in Medicine. 2015.http://www.ncbi.nlm.nih.gov/pubmed/25628755 |
||
| [150] |
Gallego-Ortiz C, Martel AL. Improving the accuracy of computer-aided diagnosis for breast MR imaging by differentiating between mass and nonmass lesions. Radiology, 2016, 278(3).
[doi:10.1148/radiol.2015150241] |
||
| [151] |
Agha M, Eid AF, Nouh M. 3T MRI of the breast with computer aided diagnosis, can it help to avoid unnecessary invasive procedures?. Alexandria Journal of Medicine, 2016, 52(1): 9–18.
[doi:10.1016/j.ajme.2014.12.005] |
||
| [152] |
Cancer Research UK. Worldwide cancer incidence statistics. 2015. http://www.cancerresearchuk.org/cancerinfo/cancerstats/world/incidence/ |
||
| [153] |
Cancer Research UK. Worldwide cancer mortality statistics. 2015. http://www.cancerresearchuk.org/cancerinfo/cancerstats/world/mortality/ |
||
| [154] | |||
| [155] |
Devi KG, Radhakrishnan R, Rajamani K. Computer aided diagnosis scheme for polyp detection in CT colonography using K-means clustering and SVM. World Journal of Medical Sciences, 2013, 9(4): 273–281.
[doi:10.5829/idosi.wjms.2013.9.4.11605] |
||
| [156] |
Hu Y, Song B, Ma M, Liang Z. A mixture classifier for computer aided diagnosis of polyp malignancy for CT colonography. In: Proc. of the 2014 IEEE Nuclear Science Symp. and Medical Imaging Conf. (NSS/MIC). IEEE, 2014. 1-5. [doi: 10.1109/NSSMIC.2014.7430959] |
||
| [157] |
Pickhardt PJ, Choi JR, Hwang I, Butler JA, Puckett ML, Hildebrandt HA, Wong RK, Nugent PA, Mysliwiec PA, Schindler WR. Computed tomographic virtual colonoscopy to screen for colorectal neoplasia in asymptomatic adults. New England Journal of Medicine, 2003, 349(23): 2191–2200.
[doi:10.1056/NEJMoa031618] |
||
| [158] |
Xu Y, Zhao J. Computer-Aided detection for CT colonography. Journal of Shanghai Jiaotong University (Science), 2014, 19(5): 531–537.
[doi:10.1007/s12204-014-1536-0] |
||
| [159] |
Yang X, Ye X, Slabaugh G. Multilabel region classification and semantic linking for colon segmentation in CT colonography. IEEE Trans. on Biomedical Engineering, 2015, 62(3): 948–959.
[doi:10.1109/TBME.2014.2374355] |
||
| [160] |
Lu L, Zhang D, Li L, Zhao J. Fully automated colon segmentation for the computation of complete colon centerline in virtual colonoscopy. IEEE Trans. on Biomedical Engineering, 2012, 59(4): 996–1004.
[doi:10.1109/TBME.2011.2182051] |
||
| [161] |
Wang H, Liang Z, Li L, Peng H. A novel computer aided detection (CADe) scheme for colonic polyps based on colon structure decomposition. In: Proc. of the Nuclear Science Symp. and Medical Imaging Conf. 2013. 1-5. [doi: 10.1109/NSSMIC.2013.6829287] |
||
| [162] |
Chen MH, Blevins M, Herman BA, Johnson CD. National CT colonography trial (ACRIN 6664): Are CT colonography, colonoscopy, and pathology giving the same measures for polyp size? In: Proc. of the Radiological Society of North America 2008 Scientific Assembly and Meeting. 2008. 279-288.https://www.researchgate.net/publication/266118870_National_CT_Colonography_Trial_ACRIN_6664_Are_CT_Colonography_Colonoscopy_and_Pathology_Giving_the_Same_Measures_for_Polyp_Size |
||
| [163] |
Song B, Zhang G, Zhu H, Zhu W, Lu H, Liang Z. A feasibility study of high order volumetric texture features for computer aided diagnosis of polyps via CT colonography. In: Proc. of the 2012 IEEE Nuclear Science Symp. and Medical Imaging Conf. (NSS/MIC). IEEE, 2012. 3940-3943. [doi: 10.1109/NSSMIC.2012.6551903] |
||
| [164] |
Zhang GP, Liao QM, Jiao C, Li BJ, Liu Y, Lu HB. Study of computer aided diagnosis technology in virtual colonoscopy. Journal of Xidian University, 2015, 42(2): 157–161(in Chinese with English abstract).
http://doi.wanfangdata.com.cn/10.3969/j.issn.1001-2400.2015.02.026 |
||
| [165] |
Sun YP, Sargent D. Colonoscopic polyp detection using convolutional neural networks. In: Proc. of the SPIE Medical Imaging. 2016. [doi: 10.1117/12.2217148] |
||
| [166] |
Giger ML, Chan HP, Boone J. Anniversary paper:History and status of CAD and quantitative image analysis:The role of medical physics and AAPM. Medical Physics, 2008, 35(12): 5799–5820.
[doi:10.1118/1.3013555] |
||
| [167] |
Xu JW, Suzuki K. Max-AUC feature selection in computer-aided detection of polyps in CT colonography. IEEE Journal of Biomedical and Health Informatics, 2014, 18(2): 585–593.
[doi:10.1109/JBHI.2013.2278023] |
||
| [168] |
Hu Y, Han H, Zhu W, Li L, Pickhardt PJ, Liang Z. An integrated classifier for computer-aided diagnosis of colorectal polyps based on random forest and location index strategies. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2016. [doi: 10.1117/12.2216353] |
||
| [169] |
Aihara H, Saito S, Inomata H, Ide D, Tamai N, Ohya TR, Kato T, Amitani S, Tajiri H. Computer-Aided diagnosis of neoplastic colorectal lesions using "real-time" numerical color analysis during autofluorescence endoscopy. European Journal of Gastroenterology & Hepatology, 2013, 25(4): 488–494.
[doi:10.1097/MEG.0b013e32835c6d9a] |
||
| [170] | |||
| [171] |
Mcneal JE, Redwine EA, Freiha FS, Stamey TA. Zonal distribution of prostatic adenocarcinoma:Correlation with histologic pattern and direction of spread. American Journal of Surgical Pathology, 1988, 12(12): 897–906.
[doi:10.1097/00000478-198812000-00001] |
||
| [172] |
Giannini V, Vignati A, Mazzetti S, Luca MD, Bracco C, Stasi M, Russo F, Armando E, Regge D. A prostate CAD system based on multiparametric analysis of DCE T1-w, and DW automatically registered images. In: Proc. of the SPIE Medical Imaging. 2013. [doi: 10.1117/12.2006336] |
||
| [173] |
Wang S, Burtt K, Turkbey B, Choyke P, Summers RM. Computer aided-diagnosis of prostate cancer on multiparametric MRI: A technical review of current research. In: Proc. of the BioMed Research Int'l. 2014. [doi: 10.1155/2014/789561] |
||
| [174] |
Peng Y, Jiang Y, Yang C, Brown JB, Antic T, Sethi I, Schmid-Tannwald C, Giger ML, Eggener SE, Oto A. Quantitative analysis of multiparametric prostate MR images:Differentiation between prostate cancer and normal tissue and correlation with Gleason score——A computer-aided diagnosis development study. Radiology, 2013, 267(3): 787–796.
[doi:10.1148/radiol.13121454] |
||
| [175] |
Lemaître G, Martí R, Freixenet J, Vilanova JC, Walker PM, Meriaudeau F. Computer-Aided detection and diagnosis for prostate cancer based on mono and multi-parametric MRI:A review. Computers in Biology and Medicine, 2015, 60(2015): 8–31.
[doi:10.1016/j.compbiomed.2015.02.009] |
||
| [176] |
Liu P, Wang S, Turkbey B, Grant K, Pinto P, Choyke P, Wood BJ, Summers RM. A prostate cancer computer-aided diagnosis system using multimodal magnetic resonance imaging and targeted biopsy labels. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2013. [doi: 10.1117/12.2007927] |
||
| [177] |
Litjens G, Karssemeijer N, Huisman H. A multi-atlas approach for prostate segmentation in MR images. In: Proc. of the MICCAI Grand Challenge: Prostate MR Image Segmentation. 2012. 1-8. |
||
| [178] |
Toth R, Ribault J, Sperling D, Madabhushi A. Simultaneous segmentation of prostatic zones using active appearance models with multiple coupled levelsets. Computer Vision & Image Understanding, 2013, 117(9): 1051–1060.
[doi:10.1016/j.cviu.2012.11.013] |
||
| [179] |
Qiu W, Yuan J, Ukwatta E, Sun Y, Rajchl M, Fenster A. Efficient 3D Multi-region Prostate MRI Segmentation Using Dual Optimization. Berlin, Heidelberg: Springer-Verlag, 2013: 304-315. DOI:10.1007/978-3-642-38868-2_26
|
||
| [180] |
Yuan J, Ukwatta E, Qiu W, Rajchl M, Sun Y, Tai XC, Fenster A. Jointly segmenting prostate zones in 3D MRIs by globally optimized coupled level-sets. In: Proc. of the Int'l Workshop on Energy Minimization Methods in Computer Vision and Pattern Recognition. 2013. 12-25. [doi: 10.1007/978-3-642-40395-8_2] |
||
| [181] |
Rampun A, Malcolm P, Zwiggelaar R. Computer-Aided diagnosis method for MRI-guided prostate biopsy within the peripheral zone using grey level histograms. In: Proc. of the 7th Int'l Conf. on Machine Vision (ICMV 2014). Int'l Society for Optics and Photonics, 2015. [doi: 10.13140/2.1.5144.3523] |
||
| [182] |
Niaf E, Rouvière O, Mège-Lechevallier F, Bratan F, Lartizien C. Computer-Aided diagnosis of prostate cancer in the peripheral zone using multiparametric MRI. Physics in Medicine and Biology, 2012, 57(12): 3833–3851.
[doi:10.1088/0031-9155/57/12/3833] |
||
| [183] |
Zhou T, Lu H. Multi-Features prostate tumor aided diagnoses based on ensemble-SVM. In: Proc. of the IEEE Int'l Conf. on Granular Computing. 2013. 297-302. [doi: 10.1109/GrC.2013.6740425] |
||
| [184] |
Chen Q, Xu X, Hu S, Li X, Zou Q, Li Y. A transfer learning approach for classification of clinical significant prostate cancers from mpMRI scans. In: Proc. of the SPIE Medical Imaging. 2017. [doi: 10.1117/12.2279021] |
||
| [185] |
Matlaga BR, Eskew LA, Mccullough DL. Prostate biopsy:Indications and technique. Journal of Urology, 2003, 169(1): 12–19.
[doi:10.1016/S0022-5347(05)64024-4] |
||
| [186] |
Welch HG, Fisher ES, Gottlieb DJ, Barry MJ. Detection of prostate cancer via biopsy in the medicare-SEER population during the PSA era. Journal of the National Cancer Institute, 2007, 99(18): 1395–1400.
[doi:10.1093/jnci/djm119] |
||
| [187] |
Gleason DF. Classification of prostatic carcinomas. Cancer Chemotherapy Reports, 1966, 50(3): 125–128.
http://europepmc.org/abstract/MED/5948714 |
||
| [188] |
Bostwick DG, Meiers I. Prostate biopsy and optimization of cancer yield. European Urology, 2006, 49(3): 415–417.
[doi:10.1016/j.eururo.2005.12.052] |
||
| [189] |
Jr Allsbrook WC, Mangold KA, Johnson MH, Lane RB, Lane CG, Epstein JI. Interobserver reproducibility of Gleason grading of prostatic carcinoma:General pathologist. Human Pathology, 2001, 32(1): 81–88.
[doi:10.1053/hupa.2001.21135] |
||
| [190] |
Epstein JI, Walsh PC, Sanfilippo F. Clinical and cost impact of second-opinion pathology. Review of prostate biopsies prior to radical prostatectomy. . American Journal of Surgical Pathology, 1996, 20(7): 851–857.
[doi:10.1097/00000478-199607000-00008] |
||
| [191] |
Doyle S, Feldman M, Tomaszewski J, Madabhushi A. A boosted Bayesian multiresolution classifier for prostate cancer detection from digitized needle biopsies. IEEE Trans. on Biomedical Engineering, 2012, 59(5): 1205–1218.
[doi:10.1109/TBME.2010.2053540] |
||
| [192] |
Han G, Liu X, Han F, Santika I, Zhao Y, Zhao X, Zhou C. The LISS-A public database of common imaging signs of lung diseases for computer-aided detection and diagnosis research and medical education. IEEE Trans. on Biomedical Engineering, 2015, 62(2): 648–656.
[doi:10.1109/TBME.2014.2363131] |
||
| [193] |
Venmathi A, Ganesh E, Kumaratharan N. A review of medical image classification and evaluation methodology for breast cancer diagnosis with computer aided mammography. Int'l Journal of Applied Engineering Research, 2015, 10(11): 30045–30054.
https://www.researchgate.net/profile/Drenganesh/publication/288808029_A_Review_of_Medical_Image_Classification_and_Evaluation_Methodology_For_Breast_Cancer_Diagnosis_With_Computer_Aided_Mammography/links/5683c8e408ae1e63f1f1bb03.pdf?inViewer=0&pdfJsDownload=0&origin=publication_detail |
||
| [194] |
Robin X, Turck N, Hainard A, Tiberti N, Lisacek F, Sanchez JC, Müller M. pROC:An open-source package for R and S+ to analyze and compare ROC curves. BMC Bioinformatics, 2011, 12(1): 1–8.
[doi:10.1186/1471-2105-12-1] |
||
| [195] |
Bunch PC, Hamilton JF, Sanderson GK, Simmons AH. A free response approach to the measurement and characterization of radiographic observer performance. In: Proc. of the Application of Optical Instrumentation in Medicine VI. Int'l Society for Optics and Photonics, 1977. 124-135. [doi: 10.1117/12.955926] |
||
| [196] |
Kong YH, Jing ML. Research of the classification method based on confusion matrixes and ensemble learning. Computer Engineering and Science, 2012, 34(6): 111–117(in Chinese with English abstract).
http://www.cnki.com.cn/Article/CJFDTotal-JSJK201206026.htm |
||
| [197] |
Shen W, Zhou M, Yang F, Yang C, Tian J. Multi-Scale convolutional neural networks for lung nodule classification. In: Proc. of the Int'l Conf. on Information Processing in Medical Imaging. Springer-Verlag, 2015. 588-599. [doi: 10.1007/978-3-319-19992-4_46] |
||
| [198] |
Watanabe CY, Ribeiro MX, Traina AJ, Traina C. A statistical associative classifier with automatic estimation of parameters on computer aided diagnosis. In: Proc. of the 201211th Int'l Conf. on Machine Learning and Applications (ICMLA). IEEE, 2012. 564-567. [doi: 10.1109/ICMLA.2012.103] |
||
| [199] |
Moon WK, Lo CM, Cho N, Chang JM, Huang CS, Chen JH, Chang RF. Computer-Aided diagnosis of breast masses using quantified BI-RADS findings. Computer Methods and Programs in Biomedicine, 2013, 111(1): 84–92.
[doi:10.1016/j.cmpb.2013.03.017] |
||
| [200] |
Bhooshan N, Giger M, Medved M, Li H, Wood A, Yuan Y, Lan L, Marquez A, Karczmar G, Newstead G. Potential of computer-aided diagnosis of high spectral and spatial resolution (HiSS) MRI in the classification of breast lesions. Journal of Magnetic Resonance Imaging, 2014, 39(1): 59–67.
[doi:10.1002/jmri.24145] |
||
| [201] |
Jiang M, Zhang S, Li H, Metaxas DN. Computer-Aided diagnosis of mammographic masses using scalable image retrieval. IEEE Trans. on Biomedical Engineering, 2015, 62(2): 783–792.
[doi:10.1109/TBME.2014.2365494] |
||
| [202] |
Kowal M, Filipczuk P, Obuchowicz A, Korbicz J, Monczak R. Computer-Aided diagnosis of breast cancer based on fine needle biopsy microscopic images. Computers in Biology and Medicine, 2013, 43(10): 1563–1572.
[doi:10.1016/j.compbiomed.2013.08.003] |
||
| [203] |
Peng Y, Jiang Y, Antic T, Giger ML, Eggener S, Oto A. A study of T2-weighted MR image texture features and diffusion-weighted MR image features for computer-aided diagnosis of prostate cancer. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2013. http://proceedings.spiedigitallibrary.org/proceeding.aspx?articleid=1657356[doi: 10.1117/12.2007979] |
||
| [204] |
Lai YC, Huang YS, Wang DW, Tiu CM, Chou YH, Chang RF. Computer-Aided diagnosis for 3-D power Doppler breast ultrasound. Ultrasound in Medicine & Biology, 2013, 39(4): 555–567.
[doi:10.1016/j.ultrasmedbio.2012.09.020] |
||
| [205] |
Ko CC, Tsai CY, Lin CH, Liao KS. A computer-aided diagnosis system of breast intraductal lesion using histopathological images. In: Proc. of the 29th Int'l Conf. on Image and Vision Computing. ACM Press, 2014. 212-217. [doi: 10.1145/2683405.2683430] |
||
| [206] |
Dalmış MU, Gubern-Mérida A, Vreemann S, Karssemeijer N, Mann R, Platel B. A computer-aided diagnosis system for breast DCE-MRI at high spatiotemporal resolution. Medical Physics, 2016, 43(1): 84–94.
[doi:10.1118/1.4937787] |
||
| [207] |
Shrivastava VK, Londhe ND, Sonawane RS, Suri JS. Computer-Aided diagnosis of psoriasis skin images with HOS, texture and color features:A first comparative study of its kind. Computer Methods and Programs in Biomedicine, 2016, 126(C): 98–109.
[doi:10.1016/j.cmpb.2015.11.013] |
||
| [208] |
Sugimoto K, Shiraishi J, Tanaka H, Tsuchiya K, Aso K, Kobayashi Y, Iijima H, Moriyasu F. Computer-Aided diagnosis for estimating the malignancy grade of hepatocellular carcinoma using contrast-enhanced ultrasound:An ROC observer study. Liver Int'l, 2016, 36(7): 1026–1032.
[doi:10.1111/liv.13043] |
||
| [209] |
Litjens GJ, Barentsz JO, Karssemeijer N, Huisman HJ. Clinical evaluation of a computer-aided diagnosis system for determining cancer aggressiveness in prostate MRI. European Radiology, 2015, 25(11): 3187–3199.
[doi:10.1007/s00330-015-3743-y] |
||
| [210] |
Huang YH, Chang YC, Huang CS, Wu TJ, Chen JH, Chang RF. Computer-Aided diagnosis of mass-like lesion in breast MRI:Differential analysis of the 3-D morphology between benign and malignant tumors. Computer Methods and Programs in Biomedicine, 2013, 112(3): 508–517.
[doi:10.1016/j.cmpb.2013.08.016] |
||
| [211] |
Moon WK, Huang YS, Lo CM, Huang CS, Bae MS, Kim WH, Chen JH, Chang RF. Computer-Aided diagnosis for distinguishing between triple-negative breast cancer and fibroadenomas based on ultrasound texture features. Medical Physics, 2015, 42(6): 3024–3035.
[doi:10.1118/1.4921123] |
||
| [212] |
Preim U, Glaber S, Preim B, Fischbach F, Ricke J. Computer-Aided diagnosis in breast DCE-MRI-Quantification of the heterogeneity of breast lesions. European Journal of Radiology, 2012, 81(7): 1532–1538.
[doi:10.1016/j.ejrad.2011.04.045] |
||
| [213] |
Han F, Wang H, Song B, Zhang G, Lu H, Moore W, Liang Z, Zhao H. Efficient 3D texture feature extraction from CT images for computer-aided diagnosis of pulmonary nodules. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2014. [doi: 10.1117/12.2043221] |
||
| [214] |
Agliozzo S, De Luca M, Bracco C, Vignati A, Giannini V, Martincich L, Carbonaro LA, Bert A, Sardanelli F, Regge D. Computer-Aided diagnosis for dynamic contrast-enhanced breast MRI of mass-like lesions using a multiparametric model combining a selection of morphological, kinetic, and spatiotemporal features. Medical Physics, 2012, 39(4): 1704–1715.
[doi:10.1118/1.3691178] |
||
| [215] |
Xiao Y, Zeng J, Niu L, Zeng Q, Wu T, Wang C, Zheng R, Zheng H. Computer-Aided diagnosis based on quantitative elastographic features with supersonic shear wave imaging. Ultrasound in Medicine & Biology, 2014, 40(2): 275–286.
[doi:10.1016/j.ultrasmedbio.2013.09.032] |
||
| [216] |
Azizi N, Tlili-Guiassa Y, Zemmal N. A computer-aided diagnosis system for breast cancer combining features complementarily and new scheme of SVM classifiers fusion. Int'l Journal of Multimedia & Ubiquitous Engineering, 2013, 8(4): 45–58.
https://www.researchgate.net/profile/Nabiha_Azizi/publication/259007838_A_computer-aided_diagnosis_system_for_breast_cancer_combining_features_complementarily_and_new_scheme_of_SVM_classifiers_fusion/links/00b495343dd0b4aa85000000.pdf?inViewer=true&disableCoverPage=true&origin=publication_detail |
||
| [217] |
Lu L, Devarakota P, Vikal S, Wu D, Zheng Y, Wolf M. Computer aided diagnosis using multilevel image features on large-scale evaluation. In: Proc. of the Medical Computer Vision Large Data in Medical Imaging. Springer-Verlag, 2013. 161-174. [doi: 10.1007/978-3-319-05530-5_16] |
||
| [218] |
Al-Yousef A, Samarasinghe S. Improved ultrasound based computer aided diagnosis system for breast cancer incorporating a new feature of mass central regularity degree (CRD). In: Proc. of the Artificial Neural Network Modelling. Springer-Verlag, 2016. 213-233. [doi: 10.1007/978-3-319-28495-8_10] |
||
| [219] |
Levman J, Warner E, Causer P, Martel A. Semi-Automatic region-of-interest segmentation based computer-aided diagnosis of mass lesions from dynamic contrast-enhanced magnetic resonance imaging based breast cancer screening. Journal of Digital Imaging, 2014, 27(5): 670–678.
[doi:10.1007/s10278-014-9723-y] |
||
| [220] |
Liu B, Jiang Y. A multitarget training method for artificial neural network with application to computer-aided diagnosis. Medical Physics, 2013, 40(1).
[doi:10.1118/1.4772021] |
||
| [221] |
Meyer-Baese A, Barbu A, Lobbes M, Hoffmann S, Burgeth B, Kleefeld A, Meyer-Bäse U. Computer-Aided diagnosis of breast MRI with high accuracy optical flow estimation. In: Proc. of the SPIE Sensing Technology+Applications. Int'l Society for Optics and Photonics, 2015. [doi: 10.1117/12.2181815] |
||
| [222] |
Tan T, Platel B, Huisman H, Sanchez CI, Mus R, Karssemeijer N. Computer-Aided lesion diagnosis in automated 3-D breast ultrasound using coronal spiculation. IEEE Trans. on Medical Imaging, 2012, 31(5): 1034–1042.
[doi:10.1109/TMI.2012.2184549] |
||
| [223] |
Levman JED, Warner E, Causer P, Martel AL. A vector machine formulation with application to the computer-aided diagnosis of breast cancer from DCE-MRI screening examinations. Journal of Digital Imaging, 2014, 27(1): 145–151.
[doi:10.1007/s10278-013-9621-8] |
||
| [224] |
Jiang M, Zhang S, Liu J, Shen T, Metaxas DN. Computer-Aided diagnosis of mammographic masses using vocabulary tree-based image retrieval. In: Proc. of the 2014 IEEE 11th Int'l Symp. on Biomedical Imaging (ISBI). 2014. 1123-1126. [doi: 10.1109/ISBI.2014.6868072] |
||
| [225] |
Liu H, Tan T, van Zelst J, Mann R, Karssemeijer N, Platel B. Incorporating texture features in a computer-aided breast lesion diagnosis system for automated three-dimensional breast ultrasound. Journal of Medical Imaging, 2014, 1(2).
[doi:10.1117/1.JMI.1.2.024501] |
||
| [226] |
Miranda GHB, Felipe JC. Computer-Aided diagnosis system based on fuzzy logic for breast cancer categorization. Computers in Biology and Medicine, 2015, 64(C): 334–346.
[doi:10.1016/j.compbiomed.2014.10.006] |
||
| [227] |
Kowal M, Filipczuk P. Nuclei segmentation for computer-aided diagnosis of breast cancer. Int'l Journal of Applied Mathematics and Computer Science, 2014, 24(1): 19–31.
[doi:10.2478/amcs-2014-0002] |
||
| [228] |
Takahashi Y, Takashima S, Hakucho T, Miyake C, Morimoto D, Jiang B, Numasaki H, Tomita Y, Nakanishi K, Higashiyama M. Diagnosis of regional node metastases in lung cancer with computer-aided 3D measurement of the volume and CT-attenuation values of lymph nodes. Academic Radiology, 2013, 20(6): 740–745.
[doi:10.1016/j.acra.2013.01.013] |
||
| [229] |
Liu Y, Cheng HD, Huang JH, Zhang YT, Tang XL, Tian JW, Wang Y. Computer aided diagnosis system for breast cancer based on color Doppler flow imaging. Journal of Medical Systems, 2012, 36(6): 3975–3982.
[doi:10.1007/s10916-012-9869-4] |
||
| [230] |
Jian W, Sun X, Luo S. Computer-Aided diagnosis of breast microcalcifications based on dual-tree complex wavelet transform. Biomedical Engineering Online, 2012, 11(1).
[doi:10.1186/1475-925X-11-96] |
||
| [231] |
Cary TW, Cwanger A, Venkatesh SS, Conant EF, Sehgal CM. Comparison of naïve Bayes and logistic regression for computer-aided diagnosis of breast masses using ultrasound imaging. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2012. [doi: 10.1117/12.911916] |
||
| [232] |
Retter F, Plant C, Burgeth B, Botella G, Schlossbauer T, Meyer-Bäse A. Computer-Aided diagnosis for diagnostically challenging breast lesions in DCE-MRI based on image registration and integration of morphologic and dynamic characteristics. EURASIP Journal on Advances in Signal Processing, 2013, 2013(1): 1–9.
[doi:10.1186/1687-6180-2013-1] |
||
| [233] |
Jing H, Yang Y, Nishikawa RM. Retrieval boosted computer-aided diagnosis of clustered microcalcifications for breast cancer. Medical Physics, 2012, 39(2): 676–685.
[doi:10.1118/1.3675600] |
||
| [234] |
Cho H, Hadjiiski L, Sahiner B, Chan HP, Paramagul C, Helvie M, Nees AV. Interactive content-based image retrieval (CBIR) computer-aided diagnosis (CADx) system for ultrasound breast masses using relevance feedback. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2012, 8315(2): 145-150. [doi: 10.1117/12.912164] |
||
| [235] |
Gomez W, Rodriguez A, Pereira W, Infantosi A. Feature selection and classifier performance in computer-aided diagnosis for breast ultrasound. In: Proc. of the 201310th Int'l Conf. and Expo on Emerging Technologies for a Smarter World (CEWIT). IEEE, 2013. 1-5. [doi: 10.1109/CEWIT.2013.6713755] |
||
| [236] |
Riely A, Sablan K, Xiaotao T, Furst J, Raicu D. Reducing annotation cost and uncertainty in computer-aided diagnosis through selective iterative classification. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2015. [doi: 10.1117/12.2082480] |
||
| [237] |
Lee SH, Kim DH, Choi JY, Ro YM. Improving positive predictive value in computer-aided diagnosis using mammographic mass and microcalcification confidence score fusion based on co-location information. In: Proc. of the SPIE. 2013. [doi: 10.1117/12.2007771] |
||
| [238] |
Elbially M, Ahmed MS, Abdelgawwad M, Ali FA, Botros F, Kadah YM. K8. Hand-Held computer aided diagnosis system with application in mammography. In: Proc. of the 30th National Radio Science Conf. (NRSC). IEEE, 2013. 549-556. [doi: 10.1109/NRSC.2013.6587962] |
||
| [239] |
Kowal M. Computer-Aided diagnosis for breast tumor classification using microscopic images of fine needle biopsy. In: Proc. of the Intelligent Systems in Technical and Medical Diagnostics. Springer-Verlag, 2014. 213-224. [doi: 10.1007/978-3-642-39881-0_17] |
||
| [240] |
Taher F, Werghi N, Al-Ahmad H. Computer aided diagnosis system for early lung cancer detection. Algorithms, 2015, 8(4): 1088–1110.
[doi:10.3390/a8041088] |
||
| [241] |
Patel BC, Sinha G. Abnormality detection and classification in computer-aided diagnosis (CAD) of breast cancer images. Journal of Medical Imaging and Health Informatics, 2014, 4(6): 881–885.
[doi:10.1166/jmihi.2014.1349] |
||
| [242] |
Plant C, Ngo D, Retter F, Zavala O, Schlossbauer T, Lobbes M, Lockwood M, Meyer-Bäse A. Computer-Aided diagnosis of small lesions and non-masses in breast MRI. Proc. of the SPIE Defense, Security, and Sensing, 2012, 8367(2): 741–770.
[doi:10.1117/12.921937] |
||
| [243] |
George YM, Zayed HH, Roushdy MI, Elbagoury BM. Remote computer-aided breast cancer detection and diagnosis system based on cytological images. Systems Journal, IEEE, 2014, 8(3): 949–964.
[doi:10.1109/JSYST.2013.2279415] |
||
| [244] |
Lee JH, Seong YK, Chang CH, Ko EY, Cho BH, Ku J, Woo KG. Computer-Aided lesion diagnosis in B-mode ultrasound by border irregularity and multiple sonographic features. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2013. [doi: 10.1117/12.2007452] |
||
| [245] |
Pack C, Shin S, Son SH, Jeon SI. Computer aided breast cancer diagnosis system with fuzzy multiple-parameter support vector machine. In: Proc. of the 2015 Conf. on Research in Adaptive and Convergent Systems. ACM Press, 2015. 172-176. [doi: 10.1145/2811411.2811504] |
||
| [246] |
Gedik N, Atasoy A. A computer-aided diagnosis system for breast cancer detection by using a curvelet transform. Turkish Journal of Electrical Engineering & Computer Sciences, 2013, 21(4): 1002–1014.
[doi:10.3906/elk-1201-8] |
||
| [247] |
Calderon-Contreras JD, Chacon-Murguia MI, Villalobos-Montiel AJ, Ortega-Maynez L. A fuzzy computer aided diagnosis system using breast thermography. In: Proc. of the 2015 IEEE 12th Int'l Symp. on Biomedical Imaging (ISBI). IEEE, 2015. 105-108. [doi: 10.1109/ISBI.2015.7163827] |
||
| [248] |
Parveen SS, Kavitha C. Detection of lung cancer nodules using automatic region growing method. In: Proc. of the 20134th Int'l Conf. on Computing, Communications and Networking Technologies (ICCCNT). IEEE, 2013. 1-6. [doi: 10.1109/ICCCNT.2013.6726669] |
||
| [249] |
Sun W, Tseng TLB, Zhang J, Qian W. Computerized breast cancer analysis system using three stage semi-supervised learning method. Computer Methods and Programs in Biomedicine, 2016, 135(C): 77–88.
[doi:10.1016/j.cmpb.2016.07.017] |
||
| [250] |
Mahapatra D. Semi-Supervised learning and graph cuts for consensus based medical image segmentation. Pattern Recognition, 2017, 63(2017): 700–709.
[doi:10.1016/j.patcog.2016.09.030] |
||
| [251] |
Deng C, Guo MZ. A new co-training-style random forest for computer aided diagnosis. Journal of Intelligent Information Systems, 2011, 36(3): 253–281.
[doi:10.1007/s10844-009-0105-8] |
||
| [252] |
Choi JY, Kim DH, Plataniotis KN, Ro YM. Classifier ensemble generation and selection with multiple feature representations for classification applications in computer-aided detection and diagnosis on mammography. Expert Systems with Applications, 2016, 46(C): 106–121.
[doi:10.1016/j.eswa.2015.10.014] |
||
| [253] |
Pereira S, Pinto A, Alves V, Silva CA. Brain tumor segmentation using convolutional neural networks in MRI images. IEEE Trans. on Medical Imaging, 2016, 35(5): 1240–1251.
[doi:10.1109/TMI.2016.2538465] |
||
| [254] |
Shin HC, Roth HR, Gao M, Lu L, Xu Z, Nogues I, Yao J, Mollura D, Summers RM. Deep convolutional neural networks for computer-aided detection:CNN architectures, dataset characteristics and transfer learning. IEEE Trans. on Medical Imaging, 2016, 35(5): 1285–1298.
[doi:10.1109/TMI.2016.2528162] |
||
| [255] |
Arevalo J, González FA, Ramos-Pollán R, Oliveira JL, Lopez MAG. Representation learning for mammography mass lesion classification with convolutional neural networks. Computer Methods and Programs in Biomedicine, 2016, 127(C): 248–257.
[doi:10.1016/j.cmpb.2015.12.014] |
||
| [256] |
Samala RK, Chan HP, Hadjiiski L, Helvie MA, Wei J, Cha K. Mass detection in digital breast tomosynthesis:Deep convolutional neural network with transfer learning from mammography. Medical Physics, 2016, 43(12): 6654–6666.
[doi:10.1118/1.4967345] |
||
| [257] |
Tajbakhsh N, Shin JY, Gurudu SR, Hurst RT, Kendall CB, Gotway MB, Gotway MB, Liang J. Convolutional neural networks for medical image analysis:Full training or fine tuning?. IEEE Trans. on Medical Imaging, 2016, 35(5): 1299–1312.
[doi:10.1109/TMI.2016.2535302] |
||
| [258] |
Ramírez J, Górriz J, Salas-Gonzalez D, Romero A, López M, lvarez I, Gómez-Río M. Computer-Aided diagnosis of Alzheimer's type dementia combining support vector machines and discriminant set of features. Information Sciences, 2013, 237(10): 59–72.
[doi:10.1016/j.ins.2009.05.012] |
||
| [259] |
Charbonnier JP, Smit EJ, Viergever MA, Velthuis BK, Vos PC. Computer-Aided diagnosis of acute ischemic stroke based on cerebral hypoperfusion using 4D CT angiography. In: Proc. of the SPIE Medical Imaging. Int'l Society for Optics and Photonics, 2013. [doi: 10.1117/12.2007735] |
||
| [260] |
Kumar SS, Moni RS, Rajeesh J. An automatic computer-aided diagnosis system for liver tumours on computed tomography images. Computers & Electrical Engineering, 2013, 39(5): 1516–1526.
[doi:10.1016/j.compeleceng.2013.02.008] |
||
| [261] |
Nagarajan MB, Coan P, Huber MB, Diemoz PC, Glaser C, Wismuller A. Computer-Aided diagnosis in phase contrast imaging X-ray computed tomography for quantitative characterization of ex vivo human patellar cartilage. IEEE Trans. on Biomedical Engineering, 2013, 60(10): 2896–2903.
[doi:10.1109/TBME.2013.2266325] |
||
| [262] |
Garnavi R, Aldeen M, Bailey J. Computer-Aided diagnosis of melanoma using border-and wavelet-based texture analysis. IEEE Trans. on Information Technology in Biomedicine, 2012, 16(6): 1239–1252.
[doi:10.1109/TITB.2012.2212282] |
||
| [263] |
Niaf E, Lartizien C, Bratan F, Roche L, Rabilloud M, Mège-Lechevallier F, Rouvière O. Prostate focal peripheral zone lesions:Characterization at multiparametric MR imaging-Influence of a computer-aided diagnosis system. Radiology, 2014, 271(3): 761–769.
[doi:10.1148/radiol.14130448] |
||
| [264] |
Moura DC, López MAG, Cunha P, de Posada NG, Pollan RR, Ramos I, Loureiro JP, Moreira IC, de Araújo BMF, Fernandes TC. Benchmarking datasets for breast cancer computer-aided diagnosis (CADx). In: Proc. of the Progress in Pattern Recognition, Image Analysis, Computer Vision, and Applications. Springer-Verlag, 2013. 326-333. [doi:
[265] |
| |
| [266] |
Gonçalves VM, Delamaro ME, Nunes F, de L dos S. A systematic review on the evaluation and characteristics of computer-aided diagnosis systems. Revista Brasileira de Engenharia Biomédica, 2014, 30(4): 355–383.
[doi:10.1590/1517-3151.0517] |
||
| [267] |
Uemura K, Miyoshi Y, Kawahara T, Yoneyama S, Hattori Y, Teranishi J, Kondo K, Moriyama M, Takebayashi S, Yokomizo Y, Yao M, Uemura H, Noguchi K. Prognostic value of a computer-aided diagnosis system involving bone scans among men treated with docetaxel for metastatic castration-resistant prostate cancer. BMC Cancer, 2016, 16(1): 1–9.
[doi:10.1186/s12885-015-2026-y] |
||
| [268] |
Ganesan K, Acharya UR, Chua CK, Min LC, Abraham KT, Ng KH. Computer-Aided breast cancer detection using mammograms:A review. IEEE Reviews in Biomedical Engineering, 2013, 6: 77–98.
[doi:10.1109/RBME.2012.2232289] |
||
| [269] |
Qian W, Sun W, Zheng B. Improving the efficacy of mammography screening:The potential and challenge of developing new computer-aided detection approaches. Expert Review of Medical Devices, 2015, 12(5): 497–499.
[doi:10.1586/17434440.2015.1068115] |
||
| [270] |
Lehman CD, Wellman RD, Buist DS, Kerlikowske K, Tosteson AN, Miglioretti DL. Diagnostic accuracy of digital screening mammography with and without computer-aided detection. JAMA Internal Medicine, 2015, 175(11): 1828–1837.
[doi:10.1001/jamainternmed.2015.5231] |
||
| [271] |
Lee KH, Goo JM, Park CM, Lee HJ, Jin KN. Computer-Aided detection of malignant lung nodules on chest radiographs:Effect on observers' performance. Korean Journal of Radiology, 2012, 13(5): 564–571.
[doi:10.3348/kjr.2012.13.5.564] |
||
| [272] |
De Boo DW, van Hoorn F, van Schuppen J, Schijf L, Scheerder MJ, Freling NJ, Mets O, Weber M, Schaefer-Prokop CM. Observer training for computer-aided detection of pulmonary nodules in chest radiography. European Radiology, 2012, 22(8): 1659–1664.
[doi:10.1007/s00330-012-2412-7] |
||
| [273] |
Zhu X. Semi-Supervised Learning Literature Survey. 2005. http://citeseerx.ist.psu.edu/viewdoc/download?doi=10.1.1.99.9681&rep=rep1&type=pdf |
||
| [274] |
Zhu X, Goldberg AB. Introduction to semi-supervised learning. Synthesis Lectures on Artificial Intelligence and Machine Learning, 2009, 3(1): 1–130.
http://ieeexplore.ieee.org/xpls/abs_all.jsp?arnumber=6813505 |
||
| [275] |
de Bruijne M. Machine learning approaches in medical image analysis: From detection to diagnosis. In: Proc. of the Elsevier. 2016. [doi: 10.1016/j.media.2016.06.032] |
||
| [276] |
Chan TH, Jia K, Gao S, Lu J, Zeng Z, Ma Y. PCANet:A simple deep learning baseline for image classification? IEEE Trans. on Image Processing, 2015, 24(12): 5017–5032.
[doi:10.1109/TIP.2015.2475625] |
||
| [277] |
Greenspan H, van Ginneken B, Summers RM. Guest editorial deep learning in medical imaging:Overview and future promise of an exciting new technique. IEEE Trans. on Medical Imaging, 2016, 35(5): 1153–1159.
[doi:10.1109/TMI.2016.2553401] |
||
| [278] |
LeCun Y, Bengio Y, Hinton G. Deep learning. Nature, 2015, 521(7553): 436–444.
[doi:10.1038/nature14539] |
||
| [279] |
Dou Q, Chen H, Yu L, Zhao L, Qin J, Wang D, Mok VC, Shi L, Heng PA. Automatic detection of cerebral microbleeds from MR images via 3D convolutional neural networks. IEEE Trans. on Medical Imaging, 2016, 35(5): 1182–1195.
[doi:10.1109/TMI.2016.2528129] |
||
| [280] |
Anthimopoulos M, Christodoulidis S, Ebner L, Christe A, Mougiakakou S. Lung pattern classification for interstitial lung diseases using a deep convolutional neural network. IEEE Trans. on Medical Imaging, 2016, 35(5): 1207–1216.
[doi:10.1109/TMI.2016.2535865] |
||
| [281] |
Dankerl P, Cavallaro A, Tsymbal A, Costa MJ, Suehling M, Janka R, Uder M, Hammon M. A retrieval-based computer-aided diagnosis system for the characterization of liver lesions in CT scans. Academic Radiology, 2013, 20(12): 1526–1534.
[doi:10.1016/j.acra.2013.09.001] |
||
| [282] |
Heye T, Davenport MS, Horvath JJ, Feuerlein S, Breault SR, Bashir MR, Merkle EM, Boll DT. Reproducibility of dynamic contrast-enhanced MR imaging. Part Ⅰ:Perfusion characteristics in the female pelvis by using multiple computer-aided diagnosis perfusion analysis solutions. Radiology, 2013, 266(3): 801–811.
[doi:10.1148/radiol.12120278] |
||
| [2] |
陶笃纯. 迈向新世纪的医学影像技术. 中国医学影像技术, 2000, 16(1): 1–2.
http://kns.cnki.net/KCMS/detail/detail.aspx?filename=zyxx200001000&dbname=CJFD&dbcode=CJFQ
|
||
| [27] |
曾倩, 崔芳芳, 宇传华, 张干深. 中国癌症发病、死亡现状与趋势分析. 中国卫生统计, 2016, 33(2): 321–323.
http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=zgwstj201602046
|
||
| [47] |
秦菊, 白红利, 刘畅, 余建群, 张洪静, 张泽江, 李为民, 张丽芝. 计算机辅助诊断在数字化胸片肺结节早期检出中的应用. 生物医学工程学杂志, 2014, 31(5): 1117–1120.
[doi:10.7507/1001-5515.20140210]
|
||
| [62] |
崔颖, 王小鹏, 于挥, 陈璐. 肺实质CT图像分割方法. 计算机工程与设计, 2015, 36(5): 1274–1278.
http://or.nsfc.gov.cn/bitstream/00001903-5/96645/1/1000007202240.pdf
|
||
| [81] |
姚旭, 王晓丹, 张玉玺, 权文. 特征选择方法综述. 控制与决策, 2012, 27(2): 161–166.
http://kns.cnki.net/KCMS/detail/detail.aspx?filename=kzyc201202002&dbname=CJFD&dbcode=CJFQ
|
||
| [92] |
李秋萍, 刘慧, 苏志远. 基于改进的半监督FCM聚类算法的肺结节分类与识别. 图学学报, 2015, 36(2): 244–250.
http://www.cnki.com.cn/Article/CJFDTOTAL-GCTX201502017.htm
|
||
| [113] |
谭婉嫦, 王金花, 蔡洪明, 杨曦, 李立. 基于微钙化检测的计算机辅助诊断系统对于乳腺导管原位癌的诊断价值. 临床放射学杂志, 2016, 35(9): 1352–1356.
http://kns.cnki.net/KCMS/detail/detail.aspx?filename=lcfs201609017&dbname=CJFD&dbcode=CJFQ
|
||
| [164] |
张国鹏, 廖琪梅, 焦纯, 李宝娟, 刘洋, 卢虹冰. 虚拟结肠镜的计算机辅助诊断技术. 西安电子科技大学学报, 2015, 42(2): 157–161.
http://doi.wanfangdata.com.cn/10.3969/j.issn.1001-2400.2015.02.026
|
||
| [196] |
孔英会, 景美丽. 基于混淆矩阵和集成学习的分类方法研究. 计算机工程与科学, 2012, 34(6): 111–117.
http://www.cnki.com.cn/Article/CJFDTotal-JSJK201206026.htm
|
 2018, Vol. 29
2018, Vol. 29


