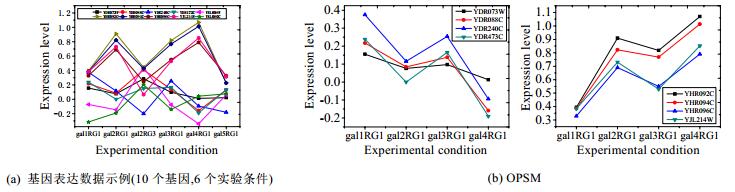
基因芯片技术加快了基因表达数据的积累, 数据的积累又促进了大量用来挖掘新的有用知识的高性能算法的设计与实现.基因表达数据广泛应用于基因组研究, 因为其方便了疾病亚型的发现与诊断.基因表达数据一般组织成矩阵的形式, 其中的行代表基因, 列代表实验条件, 每个元素代表某个基因在特定实验条件下的表达水平.图 1(a)给出一个真实的基因表达数据集(http://genomebiology.com/content/supplementary/gb-2003-4-5-r34-s8.txt
|
Fig. 1 Toy example 图 1 举例 |
目前, 聚类方法已广泛应用于基因表达数据分析领域, 但是其要求所有实验条件必须在同一聚类中.然而, 相关基因不一定在所有实验条件下共表达.基于上述观点与现实, 聚类不必过分强调聚类中包含全部的实验条件或者基因.相反地, 两个维度可以同时聚类, 该方法称为双聚类或者子空间聚类.它可以挖掘出在部分实验条件下具有相似表达水平或趋势的若干基因的集合.双聚类的概念最初由Hartigan等人[2]提出, 其作为对矩阵中的行与列同时聚类的一种方法, 并将其命名为Direct聚类.Cheng等人[3]提出了基因表达数据的双聚类, 并引入了元素残差以及子矩阵的均方残差的概念.Ben-Dor等人[1]介绍了一种特殊的双聚类模型OPSM, 并证明了它是NP难问题.随后, 研究者们提出了基于定量测度[4-14]和定性测度[15-33]的OPSM挖掘方法.定量测度包括均方残差MSR[3]、平均相关值ACV[10]、平均斯皮尔曼秩相关系数ASR[10]、平均一致性相关指数ACSI[10]等.定性测度包括上升、下降和无变化等衡量标准[24, 28].上述方法大多数是批量挖掘多种类型的OPSM, 在搜索或者查询特定的某个OPSM时显然太过笨拙.因为其或者经过多轮迭代或者批量挖掘之后从中选取目标聚类, 所以其在查询OPSM的过程中性能较差.尽管基于查询的双聚类方法[34]可以在一定程度上满足需求, 但是其输入的关键词和参数并不能真正表达分析者的领域知识.从文献中可知, 分析者的领域知识可以用约束来表示.现有的约束在基因表达数据分析[35-37]中也有应用, 其主要辅助共表达聚类的批量挖掘, 目前, 约束与查询[38]还没有共同使用.基于约束查询的OPSM分析对探索基因协同表达、调控网络的创建具有重要意义.本文主要解决的问题是, 从OPSM数据集中搜索与查询特定的符合分析者要求的若干OPSM.
基于约束条件的OPSM查询存在以下挑战.
(1) OPSM数据量庞大, 如何快速遍历如此大的数据具有难度.因为OPSM间具有交叉重叠, 所以基因或实验条件会出现在多个OPSM中.虽然具有冗余, 但又是必须的, 因为其代表了不同的生物学功能团.
(2) 如何规范而有效地将分析者的领域知识转化为自定义约束条件以及如何规范有效地利用这些规范, 都是需要解决的问题.现有文献中的自定义约束主要有must-link与cannot-link两类[37].如果用户主观上要求基因g4与g7必须同时出现在一个功能团中, 就可以应用must-link加以约束, 以减少不相关的搜索.同样地, 若生物学家意在搜寻含有基因g4但无g1的功能团, 就可以应用cannot-link加以限制.另外, 我们观察到本研究与现有方法的不同点, 即对表达值进行排序与实验条件(列)标签的替换, 所以可以在实验条件的表达值上加偏序之后, 定义一个interval约束(实验条件之间的间隔数目的限制)来减少搜索空间.若同时对OPSM的行列数目加以约束, 则同样能够加速搜索速度.
(3) 如何建立数据量小且查询速度快的索引具有挑战性.我们观察到, 每个基因名序列编码较长, 若在匹配过程中直接利用, 会极大地降低搜索速度.另外, 实验条件序列有序且较长, 也不能简单地通过变换编码的方式来解决.为此, 亟待设计高效的索引与搜索算法来解决OPSM的检索问题.
为了解决上述问题, 本文首先将OPSM中的基因名序列转换成0/1数字签名, 并将其放入Tire树中建立sTrie索引, 减少了内存的消耗.又对sTrie中的单分支节点进行压缩, 设计了cTrie索引.接着, 针对OPSM中的实验条件序列提出了基于原始或枚举实验条件的索引tTrie.然后, 为了快速通过自定义约束进行搜索, 提出了基于上述两类索引的自顶向下与自底向上搜索方式相结合的查询方法.其旨在利用一类索引从另一类索引中搜索出来的候选集, 从而加速搜索.以自顶向下搜索的sTrie(或cTrie)与以自底向上的tTrie搜索方式相结合后, 比蛮力等初步方法在性能上有较大的提升.其搜索时间复杂度为O(ζb+ωm), 其中的ζ为平均搜索分支数, b为数字签名位数, ω为候选分支数, m为平均分支长度.为了进一步解决sTrie和cTrie所产生的候选集数目庞大以及遍历效率低的问题, 提出了以自顶向下搜索的tTrie与以自底向上的sTrie(或cTrie)搜索方式相结合的方法.其搜索时间复杂度为O(ε+ζb), 其中, e为平均搜索节点数.
本文的主要工作和创新点如下.
(1) 提出了基于数字签名和Trie的基因序列与实验条件序列索引方法(第2节), 其中包括数字签名位数的计算方法、0/1 Trie的分支/节点的压缩方法以及表头的创建方法.
(2) 提出了自顶向下与自底向上相结合的查询方法(第3节), 它能够保证不同情况下的查询效率.
(3) 在真实数据集上做了大量实验, 实验结果表明, 所提出的方法具有良好的查询精确性和可扩展性(第4节).
1 问题描述本节主要介绍相关概念与解决OPSM约束型查询所用到的索引和查询框架, 用到的相关符号见表 1.
| Table 1 Notations used throughout the paper 表 1 本文用到的相关符号 |
如果后文中没有特殊说明, 基因与行、实验条件和列将交替使用, 表示同样的含义.
定义1(保序子矩阵(OPSM)).给定数据D(n×m的矩阵), Mi(g, t)是D中的一个子矩阵, 且g⊆G, t⊆T.若Mi是一个保序子矩阵, 则有g中的每一行数据e关于列标签子集t的排列严格单调递增, 即ei1≤ei2≤…≤eij≤…≤eik, 其中, (i1, ..ij, …, ik)是列标签t的一个排列.
例1:图 1(b)和图 1(c)是从图 1(a)中挖掘出来的两个OPSM, 其按照原始实验条件的先后顺序来表示.实际上, 在发现过程中, 先将每个基因的表达值按大小排序, 接着替换为列标签, 最后寻找列标签序列的最长公共子序列.
定义2(保序子矩阵的约束查询).给定自定义约束集合C(例如必须连接、不能连接、间隔约束和数量约束等)与OPSM的集合M, 保序子矩阵的约束查询是在M中寻找符合C的保序子矩阵集合QC的过程.
定义3(必须连接(must-link)).若行ga和gb(或列ta和tb)参与必须连接约束, 表示为cmust(ga, gb)(或cmust(ta, tb)), 那么必须返回处于同一个行聚类Mg(或同一个列聚类Mt)的OPSM.
例2:从表 2数据中查询g4, g5, g7所在的某功能团, 将记住的基因名g5, g7作为必须连接约束(即cmst(g5, g7))提前输入.在查询时, 将需要遍历的数据从8条减少到2条(即M0和M2), 减少了不相关数据的干扰, 降低了计算量.
| Table 2 A toy example of OPSM dataset 表 2 OPSM数据集举例 |
定义4(不能连接(cannot-link)).若行ga和gb(或列ta和tb)参与不能连接约束, 表示为ccant(ga, gb)(或ccant(ta, tb)), 那么必须返回不在同一个行聚类Mg(或同一个列聚类Mt)的OPSM.
例3:从表 2数据中查询g4但非g1所在的某个功能团, 将基因名g4, g1作为不能连接约束(即ccant(g4, g1))提前输入.在查询时, 将需遍历的数据从8条减少到1条(即M2), 减少了不相关数据的干扰, 降低了计算量.
定义5(间隔约束(interval)).若在列集上定义一个偏序( < ), 则该集合上的间隔约束表示为cint(ta, tb, i), i为非负整数, 表示ta和tb在列聚类Mt的每一个子集lj中的间隔必须为i.如果i为0, 则表示ta和tb紧挨着; 如果i为正整数, 表示ta和tb间距为i.
例4:从表 2数据中查询间隔为1的实验条件4和条件3所在的某个功能团, 将其转化为间隔约束cint(t4, t3, 1) (即间隔1的条件4和条件3在聚类Mt中).在查询时, 将候选集从8条减少到2条(即M0和M2), 减少了数据的干扰与计算量.
定义6(数量约束(count)).给定行阈值|g|、列阈值|t|以及二元关系u, v∈{≠, =, ≤, ≥, < , > ,
例5:从表 2数据中查询行列数目分别为3和不小于5的某个功能团, 将其转化为数量约束ccnt(3=, 5≥).在查询时, 将需要遍历的数据从8条减少到2条(即M2和M7), 减少了候选集的数量, 也降低了计算量.
通常, 序列查询可以转换为集合包含问题[39].然而, OPSM查询问题转化为集合包含问题之后, 其中的实验条件还需要鉴定顺序.为此, 针对OPSM基因部分的查询, 利用基于数字签名(生成方法见第2.1节)的Trie来解决.对于OPSM实验条件部分的查询, 利用基于枚举序列的前缀树索引来解决.OPSM查询分为以下两个主要步骤.
1) 创建索引.这是一个对OPSM中基因名与实验条件部分进行预处理的过程.
(ⅰ) 首先, 将基因名转换为数字签名, 如果关键词g包含于某个OPSM, 那么g的数字签名也包含于该OPSM的数字签名之中, 且利用数字签名的查询更快捷; 然后, 将数字签名放在Trie中, 这样可以节省部分内存空间.
(ⅱ) 首先, 枚举不超过一定长度的实验条件序列; 然后, 将该序列放置于前缀树之中; 最后, 建立一个辅助表头, 方便对索引的遍历与定位.
2) 查询处理.包含搜索与验证两个子步骤.
(ⅰ) 搜索, 利用基因关键词的数字签名或者实验条件序列或者子序列来遍历Trie与前缀树, 并获取相应的聚类Mi作为候选集Cq中的元素.
(ⅱ) 验证, 检测候选集Cq中的每个聚类Mi是否真正满足所有约束.
2 索引方法实现OPSM约束查询的最简单的方法是蛮力搜索, 即每次查询都完整地扫描一遍数据集, 从中选择符合条件的结果.该方法只适用于小数据集, 对大规模数据集却无能为力.为了使搜索更高效, 首先分析如表 2所示的OPSM数据集, 知道完成OPSM的约束查询需要对基因名(行)与实验条件(列)两部分分别进行处理与匹配.为此, 根据基因名与实验条件序列各自的特点, 分别提出了两种索引方法.由于基因名序列没有元素的先后之分, 设计了基于数字签名与Trie的索引方法.根据实验条件有先后顺序的特点, 提出了基于枚举序列的索引策略.
2.1 基于数字签名与Trie的索引(sTrie)数字签名signature[39]是一个长度为b的位域, 其用来代表或者近似代表一个集合.假如一个集合t中有x个元素, 且每一个元素用0到x-1之间的整数来表示, 那么这个集合的数字签名的生成方法为:将第(i mod b)位置置为1.生成的数字签名即为集合t映射到b位上的一个压缩位图.表 2数字签名列展示了为每一个基因名序列设计的4位数字签名.本文将数字签名的最左侧作为第0位, 左侧第2位作为第1位, 依次下去.在映射过程中, g0映射到第0位, g1映射到第1位, 以此类推.注意, M2与M3中的不同基因名序列具有相同的数字签名1101.
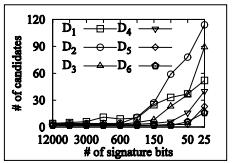
为了寻找最优的数字签名位数, 利用实验部分表 3所示的数据, 测试在12 000, 6 000, 3 000, 1 000, 600, 300, 150, 75, 50, 25位数字签名情况下的候选集中元素的数目.从图 2中可以看出:由列数较少的基因表达数据生成的OPSM数据集D1和D2, 随着数字签名位数的减少, 候选集中元素数目增加的速度相对较快.在600位数字签名之前, 候选集数目增加相对平稳, 而在其后急速增加.由列数较多的基因表达数据生成的OPSM数据集(D3, D4, D5, D6)则在150位数字签名之后才快速增加.由此得出如下结论:对由列数较少的基因表达数据生成的OPSM数据集进行数字签名转化时, 数字签名的位数应该稍多一些.
| Table 3 Details of gene expression datasets 表 3 基因表达数据集的详细情况 |
|
Fig. 2 Candidate numbers with different bits of signature 图 2 不同位数的数字签名下的候选集中元素数目 |
基于数字签名的Trie索引的创建过程(算法1) 如下:对于每一个OPSM中的基因名序列, 首先将数字签名sig中的相应位置置为1, 并将当前节点指向根节点(第2行); 接着, 对于sig中的每一位(第3行), 如果当前节点的孩子中没有等于该值的节点, 就创建一个具有该值的孩子节点, 并将当前节点指向该节点(第4行、第5行), 否则只需指向该孩子节点即可(第5行); 直到sig的最后一位时, 将当前节点设为叶子节点并存储OPSM的ID号集合(第6行).对于每一个id号(第7行), 如果opsmIdTable表头中存在该id, 则将当前节点加入以id为键的集合中; 否则, 新建一个分别以id为键、当前节点地址为值的键值对放入opsmIdTable表头中(第8行).创建表头的目的是为了方便后文的通过获取到的id编号以自底向上的方式来检验候选id是否符合条件.
算法1.基于数字签名的Trie(sTrie).
输入:OPSM数据集M.
输出:基于数字签名的Trie sTrie.
1. while M中存在下一条数据do
2. sig←0; sig
3. for i←0 to sig.length-1 do
4. if curNode的孩子节点中不存在sig[i] then将sig[i]加入到curNode的孩子节点中;
5. curNode←curNode指向孩子节点sig[i];
6. 将curNode设为末节点; 将M中该OPSM的ID存入curNode;
7. for ID中的每一个id do
8. if opsmIdTable中存在id then将curNode添加到opsmIdTable中id所在单元;
9. else将(id, curNode)加入opsmIdTable;
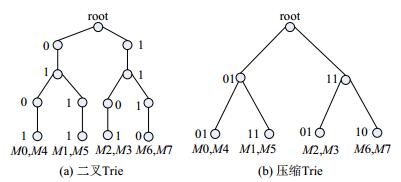
例6(sTrie索引的创建):表 2展示了一个OPSM样例数据集, 该数据将用来作为后文创建索引与演示查询方法的示例.基于数字签名的Trie的创建结果如图 3(a)所示.
|
Fig. 3 0/1 Trie example 图 3 0/1 Trie例子 |
引理1.基于数字签名的索引方法能够保证查询结果的完整性.
证明:根据数字签名的性质, 如果基因名约束转换后的集合c包含于某一个OPSM的基因名集合g中, 即c⊆g, 则有sigc⊆sigg, 其中, sigc⊆sigg:=sigc & ~sigg=0, & 与~分别是二进制中的按位与和求补操作.定理得证.
因为索引(包括后文索引)的更新方法与索引的创建方法类似, 所以在更新索引时只需调用类似于索引创建的更新方法即可.由于索引的创建方法已有详细介绍, 这里不再给出具体的算法.
2.2 基于数字签名与Trie的压缩索引(cTrie)第2.1节的基于数字签名的sTrie索引方法的一个缺点是其生成了许多不必要的节点, 这些节点仅有1个孩子, 也叫做单分支节点.从图 3(a)中也可以发现上述现象.对于k个拥有b位的数字签名, 如果没有上述单分支节点, 那么该sTire应该包含2k个节点.然而通常情况下, 数字签名越长, sTire的单分支节点就越多, 节点越多, 需要占据的内存也就越多.同时, 在遍历过程中也可能消耗更多的CPU时间.为了解决上述问题, 对上述方法进行改进, 将每个节点的单分支节点压缩到一个节点中.
为了避免单节点分支的出现, 将二分sTrie中的单分支节点放入一个节点中, 这样就保证了除叶子节点之外的所有节点都有两个分支.下面用一个例子来说明如何建立压缩Trie(即cTrie).图 3(b)中展示了一个图 3(a)中对应的压缩索引cTire.对于左分支, 只有第2个位置有分裂, 所以将第1节点、第2节点合并成一个节点01, 其后的两个分支分别合并成01和11.右分支也只有第2个位置有分裂, 所以将前两个节点合并成一个节点11, 其后的两个分支分别合并成01和10.这样就将图 3(a)中的13个节点减少到图 3(b)中的7个节点.
下面给出基于数字签名的压缩Trie索引(cTrie)的创建过程(算法2) 与算法1的不同之处, 其中, 算法1的第3行~第5行替换为算法2的第3行~第16行.在处理第1个sig时, 直接将该sig序列放入根节点的一个孩子中(第4行、第5行).对于之后的sig序列, 首先获取当前节点(首先为根节点)的孩子节点child, 并找到节点child中所存储的sigold序列与新的signew最长公共前缀lcp(第8行).如果lcp的长度为0, 则将该signew作为当前节点的另外一个孩子节点(第15行).如果节点child中的某个孩子节点为lcp, 则将当前节点指向child节点的lcp节点(第14行).如果lcp长度大于1且sigold片段有剩余, 则将现有节点的key换为lcp, 同时创建一个新的节点作为现有节点的孩子节点(第11行~第13行).如果lcp长度大于0, 则将标志位置为false(第16行).opsmIdTable表头的创建过程与sTrie类似(第18行、第19行).
算法2.基于数字签名的压缩Trie(cTrie).
输入:OPSM数据集M.
输出:基于数字签名的压缩Trie cTrie.
1. while M中存在下一条数据do
2. sig←0; sig
3. while sig不为空do
4. if curNode为根节点且没有孩子节点then
5. 将sig[i]加入到curNode的孩子节点中; curNode←curNode指向孩子节点sig[i]; sig←“Φ”;
6. else if curNode有孩子节点 then flag←true;
7. while flag为真且curNode中存在下一个孩子do
8. key←获取currentChild基因名; lcp←寻找LongestCommonPrefix(key, sig);
9. if lcp.length=sig.length then sig←“Φ”; else sig←sig中截取从lcp.length到sig.length序列;
10. if lcp.length < key.length then key_remain←key中截取从lcp.length到key.length的序列;
11. if lcp.length > 0 & key_remain.length > 0 then val←currentChild; curNode孩子节点去除key;
12. 将lcp加入curNode孩子节点; 将(key_remain, val)放入curNode中孩子lcp的孩子;
13. curNode←curNode中的孩子节点lcp;
14. else if lcp.length > 0 & key_remain.length=0 then curNode←curNode中的孩子节点lcp;
15. else if lcp.length=0 then将sig加入curNode孩子节点; curNode指向孩子节点lcp; sig←“Φ”;
16. if lcp.length > 0 then flag←false;
17. 将curNode设为末节点; 将M中该OPSM的ID存入curNode;
18. for ID中的每一个id do
19. if opsmIdTable中存在id then将curNode添加到opsmIdTable中id所在单元;
20. else将(id, curNode)加入opsmIdTable;
例7(cTrie索引的创建):利用表 2所示的数据集创建的基于数字签名的压缩树cTrie如图 3(b)所示.
2.3 基于序列的索引(tTrie)若直接将实验条件序列放入前缀树中, 遍历过程则会比较耗时.为了减少根据约束条件等关键词的定位时间, 本文直接枚举每个OPSM的实验条件部分, 将枚举的片段作为前缀树中的中间节点, 而id号放入枚举序列片段最后一个序列所在的节点中.虽然枚举序列可以加速定位, 但是枚举序列的个数随着元素数目的增加成指数级增长.通过观察得知, 经常用到的约束中关键词的长度不超过4, 所以只枚举不超过4个元素的序列.
引理2.如果将长度为m的序列枚举成长度不大于l(l < m)的子序列, 那么其空间复杂度为O(ml).
证明:对于每一序列, 其长度不大于l的序列片段的个数为
| $ O\left( {\sum\nolimits_{i = 1}^l {A_m^i} } \right) = O(l{m^l}) = O({m^l}). $ |
实验条件tTrie索引(tTrie)的创建过程(算法3) 如下.对于每一个枚举出来的特征f(第1行), 首先将当前节点指向根节点tRoot, 并取出该特征中的实验条件(第2行); 接着, 对于f中的每个实验条件(第3行), 如果当前节点的孩子中没有等于该值的节点, 就创建一个具有该值的孩子节点, 并将当前节点指向该节点(第4行、第5行), 否则只需指向该孩子节点即可(第5行); 直到f的最后一个实验条件时, 取出f中的OPSM的ID号集合(第6行); 然后, 对于f中的每个id号(第7行), 如果opsmIdTable中存在该id, 就将当前节点加入该id键所在的值中(第8行), 否则将该id作为键而当前节点作为值, 放入opsmIdTable中(第9行).
算法3.实验条件Trie(tTrie).
输入:枚举序列F.
输出:实验条件tire tTrie.
1. while F中存在下一条数据f do
2. curNode←tRoot; cols←f.cols;
3. for i←0 to cols.length-1 do
4. if curNode中不存在孩子cols[i] then将cols[i]加入curNode的孩子节点中;
5. curNode←curNode中的孩子节点cols[i];
6. 将curNode设为末节点; 将f中该OPSM的ID存入ID;
7. for i←0 to ID.size-1 do
8. if opsmIdTable中存在ID[i] then将curNode添加到opsmIdTable中ID[i]所在单元;
9. else将(ID[i], curNode)加入opsmIdTable;
例8(tTrie索引的创建):利用表 2中的数据集, 枚举的长度不大于4的序列见图 4(a).由于数量大, 只展示了以4开头的序列片段.基于枚举序列和原始序列的tTrie索引如图 4(a)和图 4(b)所示, 后者用于后文自底向上的遍历方法中.

|
Fig. 4 tTrie with different datasets 图 4 不同数据集下的tTrie |
2.4 代价分析
代价分析:在创建索引过程中, 主要关注的是预处理与索引的创建时间:
| $ {T_{index}} = {T_{sig}} + {T_{sig}}_{\_Trie} + {T_{enum}} + {T_{seq}}_{\_tree} $ | (1) |
其中, Tsig是数字签名的时间代价, Tsig_Trie是数字签名Trie的生成时间, Tenum是实验条件序列的枚举时间, Tseq_tree是索引枚举序列的时间.显然, 提高公式(1) 的性能需要依靠减少数字签名的生成时间和降低序列的枚举时间.
3 查询方法提出自顶向下和自底向上两类查询方法, 前者适用于给定关键词来定位候选集的情形, 后者适用于给定候选集后进而缩小候选集的情况.二者结合使用能够提高查询效率, 同时, 两类索引的前后顺序也影响搜索速度.以上两类方法经过组合得到以下4种方法.
(1) 基于sTrie的自顶向下与tTrie的自底向上相结合的查询方法(即GT);
(2) 基于cTrie的自顶向下与tTrie的自底向上相结合的查询方法(即GcT);
(3) 基于tTrie的自顶向下与sTrie的自底向上相结合的查询方法(TG);
(4) 基于tTrie的自顶向下与cTrie的自底向上相结合的查询方法(即TGc).
3.1 自顶向下的查询自顶向下的查询包括基于基因名数字签名索引(即sTrie和cTrie)和实验条件枚举枚举序列索引(即tTrie)这两类查询方法.
3.1.1 基于sTrie的自顶向下OPSM约束查询基于sTrie的自顶向下OPSM约束查询过程(算法4) 如下:算法的输入为sTrie索引的某个节点, 由约束转化而来的sigc以及sigc的某一个下标.如果当前所遍历的节点未到叶子节点且sigc的当前位点为1, 那么继续遍历该节点的右子树(第1行); 如果当前所遍历的节点未到叶子节点且sigc的当前位点为0, 那么继续遍历该节点的左右子树(第2行、第3行); 如果当前所遍历的节点为叶子节点, 则将该节点中的OPSM的ID号放入候选集R中(第4行).
算法4.基于sTrie的自顶向下OPSM约束查询(sTrie-TD).
输入:sTrie的某个节点, 由约束转化的数字签名sigc, sigc的位标签.
输出:OPSM ID列表R.
1. if node.level < sig.length & sigc[sig_idx]=1 then sTrie-TQ(node.right, sigc, sig_idx+1);
2. else if node.level < sig.length & sigc[sig_idx]=0 then
3. sTrie-TQ(node.left, sigc, sig_idx+1);sTrie-TQ(node.right, sigc, sig_idx+1);
4. else if node.level=sig.length-1 then将节点node中保存的ID加入R;
例9(基于sTrie索引的自顶向下查询):给定关键词g4, g5, g7, 其转化成的sigc为1101, 那么第1次迭代时的算法输入就是根节点、1101(sigc)以及0(sigc的第1位).由于当前节点是根节点, 取出其左右孩子(0和1).因为sigc的第1位为1, 所以将当前节点指向右孩子.然后, 类似地经过3次迭代后, 得到M2和M3所在分支符合条件.
定义7(投入(drop)).给定两个集合c与g以及相应的数字签名sigc与sigg, 其上的预测θ, 即sigcθsigg, 称为一个投入drop.如果sigcθsigg与cθg同时成立, 则该预测为正确的投入(right drop).如果sigcθsigg成立但cθg不成立, 则该预测为错误的投入(false drop).
引理3.给定两个集合c与g以及相应的数字签名sigc与sigg, 错误投入的概率为(1-e-k/b)k, 其中, b为数字签名的位数, k为集合的基数(本文中c和g的基数相同).
证明:具体方法类似于文献[39], 此处从略.
3.1.2 基于cTrie索引的自顶向下OPSM约束查询基于cTrie索引的自顶向下OPSM约束查询过程(算法5) 如下:采用迭代的方式处理, 其输入为cTrie索引的某个节点和由约束转化而来的sigc.若迭代中sig为空, 则返回该节点中存储的OPSM ID号(第6行); 否则(第1行), 若当前节点有孩子节点, 则返回这些孩子节点(第2行).对于每一个孩子节点, 获取其基因名序列sigold、节点地址以及与sigold同长度的sigc, 并将or初始化, 用来存储sigold与sigc的比对结果(第3行).对于其中的每一位, 若前者不小于后者, 则存储前者; 否则, 存储后者(第4行).接下来比对or与sigold:若相同, 则进入新一轮的迭代(第5行).
算法5.基于cTrie索引的自顶向下OPSM约束查询(cTrie-TD).
输入:cTrie的某个节点, 由约束转化的数字签名sigc.
输出:OPSM ID列表R.
1. if sig不为空then
2. if node中存在孩子节点then all←node的所有孩子节点;
3. for child: all do key←node中的基因名; val←node节点; str←sig从0到key.length的序列; or←“Φ”;
4. for i←0 to key.length-1 do if key[i] > =str[i] then or←or+key[i]; else or←or+str[i];
5. if or的值与key相同then cTrie-TQ(val, sig.substring(key.length, sig.length));
6. else将节点node中保存的ID加入R;
例10(基于cTrie索引的自顶向下查询):查询的输入同例9(根节点、1101(sigc)以及0(sigc的第1位)).由于当前节点是根节点, 且有两个孩子节点(01和11).因为sigc的前两位为11, 所以经过两次比较之后, 将当前节点指向11节点.接着进入第2次迭代, 经过类似的比较之后, 最后得到M2和M3所在的分支符合条件.
引理4.基于sTrie与cTrie的自顶向下的搜索时间复杂度都为O(ζb), 其中, ζ为平均搜索分支数.
3.1.3 基于tTrie索引的自顶向下OPSM约束查询基于tTrie索引的自顶向下OPSM约束查询过程(算法6) 如下:算法的输入为必须连接约束, 用到的数据为枚举的特征数据.首先将当前节点指向根节点, 必须连接的游标初始化为0, 并将分支是否遍历的标志位设为真(第1行).对于每个分支, 如果标志位为真, 则判断该节点是否含有第idx个必须约束的孩子:若有且游标idx没有到达最后一位, 则将当前节点指向该孩子节点(第3行、第4行); 如果idx指向最后一位, 则将当前节点中存储的OPSM的ID号放入结果集合R中, 并将标志位设为假(第5行).
算法6.基于tTrie索引的自顶向下OPSM约束查询(tTrie-TD).
输入:必须连接tMust.
输出:OPSM ID列表R.
1. curNode←tTrie的根节点, idx←0, flag←true;
2. while flag为真do
3. if curNode的孩子中存在tMust.get(idx)且idx不等于|tMust|-1 then
4. curNode←urNode的孩子节点tMust.get(idx);
5. idx++; if idx等于|tMust|-1 then将节点curNode中保存的ID加入R; flag←false;
例11(基于tTrie索引的自顶向下查询):给定关键词4, 5, 3(必须连接), 首先将当前节点指向根节点, 接着从其孩子节点中找到4节点, 然后找到4的孩子节点5, 接下来找到5的孩子节点3.此时关键词已用完, 就从当前节点中取出ID号(M2)作为候选集.
引理5.基于tTrie的自顶向下的搜索时间复杂度为O(ε), 其中, ε为平均搜索节点数.
3.2 自底向上的查询包括基于基因名数字签名索引(即sTrie和cTrie)和实验条件枚举枚举序列索引(即tTrie)的两类查询方法.
3.2.1 基于sTrie的自底向上OPSM约束查询基于sTrie的自底向上OPSM约束查询过程(算法7) 如下:算法的输入为由约束转化而来的sigc和候选集ID.对于候选集中的每个id, 首先初始化支持它的分支的个数, 并将数字签名中为1的位置数字放入链表rows中(第1行).如果id在opsmIdTable中不为空, 则取出所有分支末节点(不一定是叶子节点), 并以自底向上的方式检验该分支是否符合条件(第2行).对于取出来的每个分支节点, 首先进行初始化工作(第3行); 从该分支节点自底向上遍历(第4行), 当节点标号idx_brc等于关键词rows中的第idx_key个元素(第5行)时, 若检测到当前节点中的元素为0, 则将标志位flag置为flase, 且终止对该分支的遍历, 若rows没有检测完(idx_key大于0), 将idx_key减少1 (第6行).如果节点标号idx_brc等于关键词rows中的第idx_key个元素(第5行), 则将idx_brc减少1, 并将当前节点指向父节点(第7行).在遍历完每一分支后, 若flag为真, 则将支持id的分支数目idBrcCnt加1(第8行).在遍历完每一id的所有分支后, 若其支持数量idBrcCnt为0, 则从ID中删除(第9行).最后, 将ID加入R(第10行).
算法7.基于sTrie的自底向上OPSM约束查询(sTrie-BU).
输入:由约束转化的数字签名sigc, OPSM候选集ID.
输出:OPSM ID列表R.
1. for id: ID do for i←0 to sigc.length-1 do if sigc[i]为1 then将i加入rows;
2. if opsmIdTable中存在id then nodes←opsmIdTable表id单元中的节点;
3. for node: nodes do curNodes←node; idx_brc←sigc.length-1; idx_key←rows.length-1; flag←true;
4. while curNode不为sTrie的根节点do
5. if idx_brc等于rows中第idx_key个元素then
6. if curNode.getgName=0 then flag←false; break; if idx_key > 0 then idx_key--;
7. idx_brc--; curNode←curNode的父节点;
8. if flag then IdBrcCnt++;
9. if idBrcCnt=0 then从ID中去除id;
10.将ID中的所有元素加入R;
例12(基于sTrie索引的自底向上查询):给定与例9相同的关键词g4, g5, g7(sigc为1101) 以及例11获取到的候选集M2.首先, 将sigc中为1的位数记录在rows链表中, 则rows存储的为0, 1, 3;接着, 从opsmIdTable表头中取出M2所在分支的叶节点; 然后, 以自底向上的顺序检测到该分支3, 1, 0所在位置为1, 所以M2符合条件.
3.2.2 基于cTrie的自底向上OPSM约束查询基于cTrie索引的自底向上OPSM约束查询(算法8) 与算法7大致相同, 不同之处如下:第3行中加入了判断rows中关键词是否用完的标志位flag_key, 并在第4行的while中判断该标志位; 第5行~第7行替换为首先判断当前节点中所存储位点的左分支是否小于idx_key, 若小于等于idx_key, 则取出当前节点中idx_key所对应的位点; 接着, 如果该位点为0, 则将标志位flag置为flase, 且终止对该分支的遍历; 若rows中的关键词没有检测完(idx_key大于0), 则将idx_key减1(第6行); 若rows中的关键词已经检测完(idx_key等于0), 则将标志位flag_key置为flase, 且终止对该分支的遍历.如果节点标号idx_brc_left小于关键词rows中的第idx_key个元素(第5行), 则将idx_brc_left赋值为idx_brc_left减少该节点存储的数字签名位数后的值, 并将当前节点指向父节点(第7行).
算法8.基于cTrie的自底向上OPSM约束查询(cTrie-BU).
输入:由约束转化的数字签名sigc, OPSM候选集ID.
输出:OPSM ID列表R.
1~4.同算法7.
5. if idx_brc_left≤row的第idx_key个元素then bit←curNode中基因名数字签名的第idx_key位;
6. if bit=0 then flag←false; break; if idx_key > 0 then idx_key--; if idx_key=0 then flag_key←false; break;
7. else
idx_brc_left←idx_brc_
8~10.同算法7.
例13(基于cTrie索引的自底向上查询):给定与例9相同的关键词g4, g5, g7(sigc为1101) 以及例11获取到的候选集M2.搜索与比对方式与例12类似, 不同之处为:本方法是每次获取到的数字签名位数不确定, 需要在比对过程中做额外的对齐下标的处理.最后得到的结果同样为M2.
引理6.基于sTrie与cTrie的自底向上的搜索时间复杂度都为O(ζb), 其中, ζ为平均搜索分支数.
3.2.3 基于tTrie的自底向上OPSM约束查询基于tTrie索引的自底向上OPSM约束查询过程(算法9) 如下:算法的输入为OPSM的集合ID、必须连接和不能连接约束条件.用到的数据为完整的实验条件序列, 而不是枚举的实验条件序列.原因是可以减少候选集中元素的个数, 减少后续的验证工作.对于每个由tTrie索引得到的OPSM(第1行), 如果opsmIdTable表头中存在该id, 就返回包含该id的节点(第2行).对于上述获取到的每个节点, 首先获取每个节点为当前节点(第3行), 接着判断该节点中的列标签是否在不能连接约束中:如果在, 则终止该分支的遍历; 否则, 将当前列标签放入最长公共子序列lcs中.之后, 指向当前节点的父节点, 直到根节点(第4行、第5行).如果获取到的lcs与必须连接约束的反序列的lcs的长度大于1, 则将该id的lcs计数加1(第6行).对于lcs计数为0的id号, 从ID中删除, 并将ID的长度与下标i的数值减1(第7行).最后, 将ID赋值给OPSM列表R, 并作为结果返回(第8行).
算法9.基于tTrie的自底向上OPSM约束查询(tTrie-BU).
输入:OPSM候选集ID, 必须连接tMust, 不能连接tNot.
输出:OPSM ID列表R.
1. for i←0, len←ID.length to len-1 do
2. if opsmIdTable中存在ID[i] then nodes←opsmIdTable获取ID[i]中的所有节点;
3. for j←0 to nodes.size-1 do curNode←nodes的第j个元素;
4. while curNode不为根节点do if tNot包含curNode.col then break; else将curNode.col加入lcs;
5. curNode←curNode的父节点;
6. if lcs与tMust的反序列的LCS长度 > 1 then count++;
7. if count=0 then从ID中去除ID[i]; --len; --i;
8. R←ID;
例14(基于tTrie索引的自底向上查询):给定与例11相同的关键词4, 5, 3(必须连接), 1, 6(不能连接)以及例9(例10) 获取到的候选集M2, M3.首先找到M2所在分支的节点, 接着以自底向上的方式比对关键词, 依次遍历节点0, 3, 5, 4, 2, 其中包括关键词4, 5, 3, 且没有关键词1, 6, 所以该分支符合条件.接下来搜索M2所在的分支.由于在5之前没检测到3(自底向上方式, 所以关键词顺序相反), 所以提前终止该分支的遍历.最后只有M2符合条件.
引理7.基于tTrie的自底向上搜索的时间复杂度为O(ωm), 其中, ω为候选分支数, m为平均分支长度.
3.3 性能优化规则1(基于关键词顺序的剪枝).在遍历sTrie与cTrie的过程中, 如果某一位关键词位置为0, 那么此分支中剩下的元素就不必遍历.同样, 在遍历tTrie的过程中, 按照输入的列关键词顺序和相应分支中的元素比对.如果此分支中的元素顺序和关键词顺序不一致, 那么此分支中剩下的元素就不必遍历.
搜索方法给出相关结果之后, 还不能保证结果相关性较高的排在前面.为此, 给出了对结果排名用到的标准与定义.
定义8(排名).对于符合所有约束的结果, 按照每个Mi中行列数目和数目约束的接近程度来定义其排名, 即越接近数目约束的排名越靠前.其计算方法如公式(2) 所示.
| $ {R_{{M_i}}} = {\left| {|M_i^r|-|C_{cnt}^r|} \right|^2} + {\left| {|M_i^t|-|C_{cnt}^t|} \right|^2} $ | (2) |
例15:利用表 2中的数据和例14中的查询结果为M2.如果还有另外一个结果M8(g4 g5 g7:2 4 0 3 6 5), 那么根据定义8有, M0排在M6前边, 因为
代价分析:在OPSM约束查询处理中, 主要关注的是查询响应时间:
| $ {T_{response}} = {T_{search}} + |{C_q}| \times {T_{cst\_test}} $ | (3) |
其中, Tsearch是搜索部分的耗时, Tcst_test是约束型检测所需要的平均时间.在验证部分, 剔除候选集Cq中的所有假阳性Mi需要|Cq|×Tcst_test时间.通常, 验证时间占据响应时间公式(3) 的主要部分, 主要是因为|Cq|的数目比较庞大.另外, 对于不同的约束查询, Tcst_test的变化并不明显.因此, 减小约束查询响应时间的关键工作就是最小化候选集Cq的大小.如果OPSM数据集太大而不能存储于内存, Tsearch是将占据查询相应时间的重大部分.
4 实验评估本节主要评估以下6种方法的有效性与可扩展性:(1) 暴力搜索法(BF); (2) 基于sTrie的自顶向下与tTrie的自底向上相结合的查询方法(GT); (3) 基于cTrie的自顶向下与tTrie的自底向上相结合的查询方法(GcT); (4) 基于tTrie的自顶向下与sTrie的自底向上相结合的查询方法(TG); (5) 基于tTrie的自顶向下与cTrie的自底向上相结合的查询方法(TGc); (6) KiWi[40].实验中用到了两种数据:真实数据和生成数据.大多数实验是在真实数据上进行的, 因为它是真实需求的来源.实验用到的机器的是1.87GHz频率, 16GB内存, 且运行着Ubuntu 12.04的浪潮服务器(在分布式并行情况下, 可利用的最大节点数为9).3种方法用Java语言编写, 由Eclipse 4.3编译运行.
数据生成方法:先从网站[41]上下载如表 3所示的6个数据集; 接着, 利用快速排序法对每一行的表达值排序; 然后, 将每一个表达值替换成对应的列标签, 使其变成序列数据[42]; 最后, 利用文献[38, 43]中的列模糊查询方法(关键词为相应数据集中所有的列标签, 列和行阈值都为2) 生成表 4中描述的6个数据集.
| Table 4 Details of OPSM datasets 表 4 OPSM数据集的详细情况 |
枚举序列(特征)见表 4.
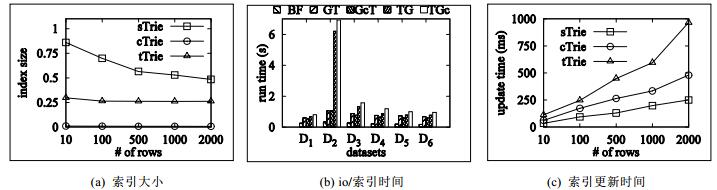
4.1 单机性能 4.1.1 索引性能首先测试sTrie, cTrie和tTrie这3种方法在索引10行, 100行, 500行, 1 000行和2 000行的D2数据集的情况下的索引大小, 其中, sTrie和cTrie用到的数据为表 3中的D2数据集, tTrie用到的是表 4中的D2数据集.索引大小定义为索引的数据量与原始数据量的比值.3种索引的索引大小如图 5(a)所示.当索引的D2中的数据量由10行增加到2 000行的过程中, sTrie索引的数据量占原始数据的比重大小由86%下降到48%, cTrie索引的数据量占原始数据的比重大小由1%下降到0.67%, tTrie索引的数据量占原始数据的比重大小由30%下降到26%.sTrie与cTrie利用的是同样的数据而索引压缩比却有很大差别, 这是因为cTrie对sTrie中的单分支节点进行了大幅度的压缩.tTrie索引较小的原因是tTrie利用的数据为实验条件枚举序列, 其最大长度为4, 所以会有较多的可重复利用节点, 因而索引较小.
|
Fig. 5 Performance evaluation of index methods on a single machine 图 5 索引方法在单机上的性能评估 |
接着评估5种方法在表 3和表 4所示的数据集上的性能.5种方法在6种数据集上所消耗的I/O或者索引创建时间如图 5(b)所示, BF的I/O在0.2s左右, 虽然不大, 但是由于其没有索引, 致使在每次执行查询时这一耗时都是必须的.GT创建索引耗时在0.5s~1s之间, 其压缩索引GcT索引耗时也在0.5s~1s之间.TG创建索引耗时在0.6s~6s之间, 其压缩索引TcG索引耗时也在0.8s~6s之间.TG和TcG耗时大于GT和GcT的原因是:前者索引的是实验条件的枚举序列(见表 4), 数据量相对较大; 后者索引的是原始的实验条件序列(见表 3), 数据量相对较小.TG与TcG索引中用到的特征数据分别来自6个OPSM数据集合中的实验条件部分, 且实验条件的数目由少到多, 数据量D2 > D3 > D4 > D5 > D6 > D1.
最后测试sTrie, cTrie和tTrie这3种索引在插入10行, 100行, 500行, 1 000行和2 000行的D2数据集的情况下的性能.索引更新时间如图 5(c)所示, sTrie由35ms平缓增加到250ms, cTrie由62ms缓慢增加到480ms, tTrie由112ms快速增加到966ms.由于cTrie是sTrie的压缩索引, 其在节点的压缩方面要比后者多付出一些代价, 所以cTire耗时多于sTrie.因为tTrie索引的是特征数据, 而特征数据的key较短但value较多, 每次都要检测分支的末节点中是否已经含有value中的每个值, 所以tTrie索引更新耗时相对较多.
4.1.2 查询性能首先测试5种方法在枚举表 4所示数据时的性能, 如图 6(a)所示.D2数据上的枚举时间最长, 因为其数据量远远多于其他数据.D1数据上的枚举时间最短, 因为其数据量远远少于其他数据.其他数据上的枚举时间基本相同, 这是因为虽然数据量的顺序为D3 > D4 > D5 > D6, 但是列数顺序则恰好相反.5种方法在6种数据集上进行同一种查询(3个must-link、3个cannot-link、2个interval和2个count约束)所消耗的时间如图 6(b)所示, BF的查询时间在158ms~206ms之间, GT的查询时间在36ms~115ms之间, GcT的查询时间略高于GT, 在74ms~143ms之间, TG与TcG的查询耗时基本相同, 分别在1ms~17ms与0ms~14ms之间.TG与TcG的查询性能要优于另外3种方法, 原因是TG与TcG的定位速度与候选集数目都要小于另外3种方法.
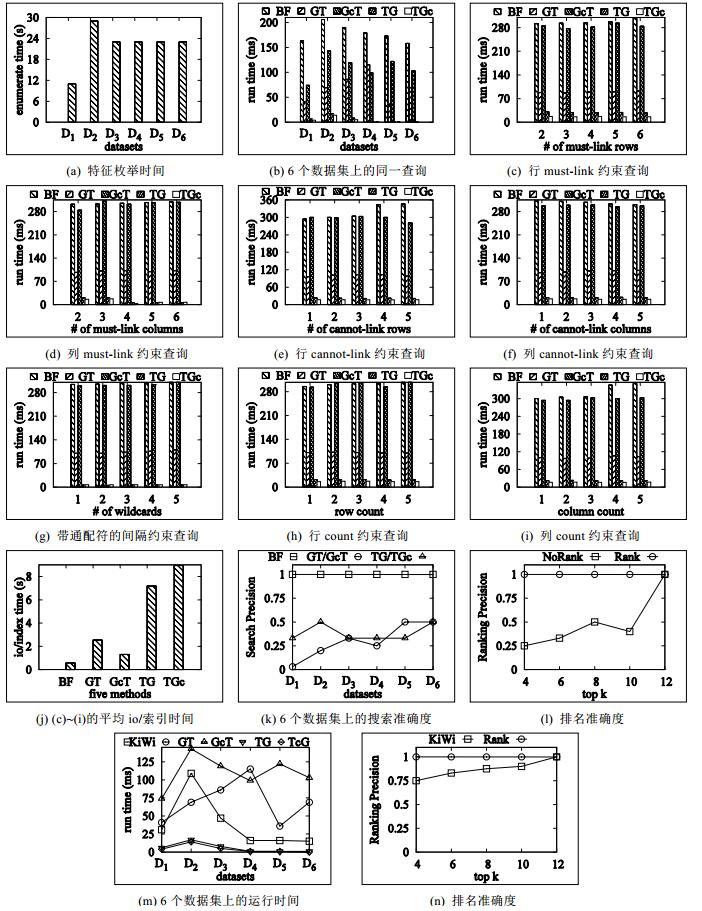
|
Fig. 6 Performance evaluation of 5 methods on a single machine 图 6 5种方法在单机上的性能评估 |
接着进行must-link查询的性能评估, 其性能如图 6(c)和图 6(d)所示.当行的must-link关键词数目由2增加到6的过程中, BF的查询时间由292ms增加到308ms, GT的查询时间由86增加到92ms, GcT的查询时间在277ms~293ms之间徘徊.GcT是GT的压缩索引, 但GcT的查询耗时却远大于GT, 基本与BF相当.原因是GcT失去了GT的二分查找特性, 基本要将所有分支都遍历一下.TG的查询时间在27ms~31ms之间, TcG的查询耗时在16ms~17ms之间.TG与TcG的性能远远优于BF, GT与GcT.同样地, 当列的must-link关键词数目由2增加到6的过程中, BF的查询时间由302ms增加到309ms, GT的查询时间由97ms增加到102ms, GcT的查询时间在284ms~312ms之间徘徊.GT性能优于GcT的原因同上.TG的查询时间在6ms~20ms之间, TcG的查询耗时在3ms~17ms之间.同样地, TG与TcG的性能远远优于BF, GT与GcT.
下面进行cannot-link查询的性能评估, 其性能如图 6(e)和图 6(f)所示.在行的cannot-link关键词数目由1增加到5的过程中, BF的查询时间由292增加到308ms, GT的查询耗时在96ms~103ms之间, GcT的查询时间在281ms~303ms之间.TG的查询时间在21ms~23ms之间, TcG的查询耗时在16ms~17ms之间.TG与TcG的性能远远优于BF, GT与GcT.在列的cannot-link关键词数目由1增加到5的过程中, 性能和行关键词上的测试相同, 且TG与TcG的性能远远优于BF, GT与GcT.
接下来评估interval约束查询中通配符的使用对查询性能的影响, 其性能如图 6(g)所示.在interval约束中, 通配符的数目由1增加到5的过程中, BF的查询时间由304ms增加到310ms, 其原因是通配符越多, 需要遍历的数据就越多, 进而耗时也就越多; GT的查询耗时由100增加至110ms, GcT的查询时间由300到310ms.TG的查询时间在5ms~7ms之间, TcG的查询耗时维持在7ms.同样地, TG与TcG的性能远远优于BF, GT与GcT.
接着评估count约束查询中行列count的多少对查询性能的影响, 其性能如图 6(h)和图 6(i)所示.在行count由1增加到5的过程中, BF的查询时间由295ms增加到307ms, GT的查询耗时在100ms~102ms, GcT的查询时间在293ms~307ms之间, TG的查询时间在21ms~22ms之间, TcG的查询耗时维持在16ms~17ms.
图 6(j)展示了上述4种约束查询之前扫描数据或者对数据进行索引的耗时.BF扫描一遍数据所需要的平均时间约为0.6s, 但每次查询都要扫描数据.假如数据量非常庞大, 那么这部分耗时会急剧增加, 所以此方法不可取.GT方法在0/1数字签名与实验条件枚举序列数据上建立索引所消耗的平均时间约为2.54s, 实验条件序列枚举代价是对最大长度为4的特征进行索引所耗费的时间.如果全部长度的特征都要索引, 则所需耗时会更长. GcT索引没有单节点分支, 即每个节点存储较多数据使得开辟新节点个数更少, 所以耗时约为GT的一半(1.3s).TG和TcG索引的数据同为原始实验条件序列与0/1数字签名序列, 二者耗时基本相同, 分别为7.2s与8.9s.
然后, 评估所提出方法的准确性.图 6(k)展示了BF, GT, GcT, TG和TcG这5种方法在6种不同数据集上执行同一查询所返回候选集的准确度.GT和GcT方法在6种数据集上的准确度由0.03提高到0.5, TG和TcG方法则维持在0.33和0.5之间, 而BF方法则一直为1.这是因为BF方法是对每个OPSM利用4种约束进行搜索, 所以搜索到的结果都是正确的, 而另外4种方法是没有完全利用4种约束做搜索, 剩余的约束在后续验证中才用到.图 6(l)给出了ES和CQ两种方法在有无利用排名技术的情况下所返回最终结果排名的准确度.由于前者(没有利用排名技术)返回的结果是随机排列的, 而后者(利用排名技术)是将最重要的首先返回, 所以后者对最终结果的排名更为准确.
最后, 比较文献[20, 29]中的KiWi方法(代码下载地址见文献[40])与本文所提出所有方法在时间效率和准确度方面的性能.图 6(m)展示了KiWi和GT等5种方法在6种数据集上执行同一查询的运行时间.KiWi在D2数据集上的运行时间最长(109ms), 在D3和D1数据集上的运行时间稍短(分别为47ms和31ms), 在D4, D5和D6数据集上的运行时间最短(基本为16ms).同样地, TG和TcG在D2数据集上的运行时间最长(17ms和14ms), 在D3和D1数据集上的运行时间稍短(7ms), 在D4, D5和D6数据集上的运行时间最短(1ms).GcT的趋势稍有不同, 在D2数据集上的运行时间最长(143ms), 在D3和D5数据集上的运行时间稍短(120ms), 在D4和D5数据集上的运行时间更短些(100ms), 在D1数据集上的运行时间最短(74ms).GT的趋势则有较大的不同, 在D4数据集上的运行时间最长(115ms), 在D3数据集上的运行时间稍短(86ms), 在D2和D6数据集上的运行时间更短些(69ms), 在D1和D5数据集上的运行时间最短(40ms).图 6(n)给出了KiWi和Rank两种方法在前k个结果中的准确度.在top-k中的k由4增加到12的过程中, KiWi所返回结果的准确度由0.75逐步提高到1, 且接近于Rank的准确度1.因为前者首先返回列长的结果再返回行多的结果, 而后者要综合考虑行和列的数目, 先返回行和列数目相对较多的, 再返回较少的.
4.2 并行性能上一节已经验证了5种方法在单机上的性能, 本节将测试3种方法在分布式并行平台下(包括1个master节点、8个slave节点)的表现.图 7(a)给出了BF, GT, GcT, TG和TcG这5种方法执行同样的约束查询(3个must-link、3个cannot-link、2个interval和2个count约束)在6种数据集上的运行时间.BF在D2数据集上的运行时间最长(117ms), 原因是D2数据集中的行数是最多的.D3, D4和D5数据集上的查询时间随着行数的减少而减少.D1数据集的行数是最少的, 但是其上的查询时间却排名前三位, 原因是行所拥有的数据量远多于其他数据集.GT方法基本与BF类似, 但是在D5和D6数据集上例外.同样地, GcT方法基本与BF类似, 但是在D3和D4数据集上例外. TG和TcG两种方法运行速度很快, 最长耗时为4ms, 最短耗时为0ms.此实验证明了5种方法在不同数据集上执行查询的可扩展性.TG和TcG方法的查询性能最好, 因为索引的结合顺序减少了候选集数目、缩短了查询时间.

|
Fig. 7 Performance evaluation of 5 methods on Hadoop 图 7 5种方法在Hadoop上的性能评估 |
图 7(b)展示了BF, GT, GcT, TG和TcG这5种方法在18 748行数据上执行不同的列must-link关键词而其他关键词约束相同的查询所消耗的时间.BF方法最小运行时间为232ms, 最大运行时间为245ms, 基本符合随着关键词的增加, 查询时间在增加的预测.GT方法受关键词影响较小, 基本稳定在70ms.虽然GcT方法只是对GT方法索引做了压缩, 但是其失去了GT方法的遍历优势, 所以非常耗时, 甚至超过了BF方法.由于TG和TcG方法具有对候选集的良好过滤能力, 所以二者的查询时间基本维持在3ms左右.此实验证明了5种方法在不同数目的关键词下执行查询的可扩展性.
最后验证BF, GT, GcT, TG和TcG这5种方法在不同集群节点(1, 2, 4, 8个节点)上执行同一种约束查询的可扩展性.如图 7(c)所示, 随着节点数目的增加, BF方法的查询时间由690ms减少到244ms, 性能提高了将近3倍; GT方法的查询时间基本维持在65ms~70ms范围内; GcT方法的查询时间由1 347ms减少到216ms, 性能提高了6倍以上; TG方法的查询时间由21ms减少到5ms, 性能提高了4倍以上; TcG方法的查询时间由21ms减少到3ms, 性能提高了7倍.此实验说明, BF, GT, GcT, TG和TcG这5种方法随着节点的增加, 都具备有良好的可扩展性.
5 相关工作本节主要回顾和分析与约束型查询有关的文献, 主要分3类:(1) 基于定量和定性测度的子空间聚类; (2) 基
于查询的双聚类; (3) 约束型双聚类.对每一类工作只做简单介绍, 更详细的描述可查阅Sim等人的综述[44-48].
1) 基于定量和定性测度的子空间聚类
双聚类的概念最初由Hartigan等人[2]提出, 作为对矩阵中的行与列同时聚类的一种方法, 并将其命名为Direct聚类.Cheng等人[3]提出了基因表达数据的双聚类, 并引入了元素残差以及子矩阵的均方残差MSR[3]的概念.他们提出了一种贪婪方法.首先, 将整个数据矩阵作为初始化数据; 接着, 删除元素残差或者均方残差最大元素或者行列, 依次递归下去, 直到剩余矩阵的MSR低于某个阈值; 然后, 增加部分元素或者行列, 保证所得矩阵的MSR也低于该阈值.该方法效率较低, 因为一次只能挖掘1个双聚类.Ben-Dor[1]介绍了一种特殊的双聚类模型OPSM, 并证明了其是NP难问题.随后, 研究者们提出了基于定量测度[4-14]和定性测度[15-33]的OPSM挖掘方法.定量测度包括均方残差MSR[1]、平均相关值ACV[10]、平均斯皮尔曼秩相关系数ASR[10]、平均一致性相关指数ACSI[10]等.定性测度包括上升、下降和无变化[24, 28].
基于Cheng等人[2]提出的δ-bicluster模型, Yang等人[4]为了减少数据缺失值的影响, 给出一种δ-cluster模型. Cho等人[5]介绍了两种与MSR相似的平方残差测度, 同时提出了两种有效的基于k-means的双聚类算法.Divina等人[6]给出了一种基于进化计算的双向聚类方法, 用来发现尺寸较大、重叠较少且MSR小于某阈值的双聚类. Deodhar等人[7]提出了一种鲁棒的有重叠的双聚类方法ROCC, 能够有效地从大量的含有噪声的数据中挖掘出稠密的、任意位置的有覆盖的聚类.Cho等人[8]给出了数据转换的方法, 以解决现有的平方残差和测度方法只能有效地挖掘出在数值上具有偏移的双聚类, 却不能很好地解决在数值上有缩放的双聚类问题.Odibat等人[9]发现, 现有方法并不能有效地挖掘矩阵数据中任意位置有重叠的双聚类, 提出了确定性双聚类算法.该算法可以有效地发现正负相关的任意位置上有重叠的双聚类.Ayadi等人[10]利用平均一致性相关指数ACSI来评估相干双聚类, 并利用有向无环图组建这些双聚类.Truong等人[11]提出了一种算法, 用来生成若干个覆盖度小于阈值的双聚类, 在一定程度上能够发现无冗余的双聚类.Ayadi等人[12]给出了模因双聚类算法MBA, 以发现生物学意义上的重要的负相关双聚类.Chen等人[13]利用最小均方错误MMSE测度来鉴别所有类型的线性模式(偏移、缩放、偏移与缩放联合模式).Denitto等人[14]利用Max-Sum测度来提升双聚类的质量.
Wang等人[15]提出并设计了基于pScore测度和pCluster模型的方法, 以挖掘具有相似升降趋势的模式.Liu等人[16]通过寻找在部分维度下表达值排序相同的基因等对象来挖掘OPSM.Wang等人[17]给出了一种基于最近邻的新的测度方法来挖掘相似模式.Kriegel等人[18]提出了一种局部密度阈值的OPSM挖掘方法, 试图改变现有的基于全局密度阈值的方法并不能适用于每个OPSM的现状.Jiang等人[19]给出了一种质量驱动的top-k模式挖掘方法, 以提升发现的有重叠的OPSM的质量.Gao等人[20, 29]提出了一种KiWi框架, 利用k和w两个参数来约束计算资源和搜索空间.Zhang等人[21]发现, 现有的方法都假设基因表达数据是同质的, 给出了称为F-cluster的模型来挖掘异质数据中的相干模式.Yan等人[22]为挖掘非线性相关的模式, 设计了适用于时序基因表达数据的联合聚类方法MI-TSB.Zhang等人[23]提出了一种近似保序聚类模型AOPC, 以减少数据中噪声的影响.Chui等人和Yip等人[24, 30]利用多份数据模型OPSM-RM来消除数据噪声的影响.Zhao等人[25]提出了一种最大化子空间聚类算法, 来挖掘具有正相关和负相关的共调控基因聚类.Trapp等人[26]为挖掘最优OPSM, 给出了一种基于线性规划的挖掘方法.Fang等人[27]为挖掘放松的OPSM, 提出了包含以行或列为中心的OPSM-Growth方法.随后, Fang等人[28, 32]提出了基于桶和概率的方法, 挖掘出放松的OPSM.An等人[31]利用互信息和核密度进行双聚类.Cho等人[33]给出了一种基于坏字符规则的KMP算法, 试图快速匹配保序模式.
2) 基于查询的双聚类[49]
该方法来自生物信息领域[50-53], 应用对象是基因表达数据.首先, 由用户根据经验来提供功能相关或共表达的种子基因; 接着, 利用该种子对搜索空间剪枝或双聚类进行指导.为了使现有挖掘方法能利用先验知识并回答指定的问题, Dhollander等人[54]提出了基于贝叶斯的查询驱动的双聚类方法QDB.同时, 给出了一种基于实验条件列表的联合方法, 以实现关键词的多样性并免除必须事先定义阈值等问题.随后, Zhao等人[55]对QDB方法进行了改进, 提出了ProBic方法.虽然二者在概念上相似, 但是也有不同之处.QDB方法利用概率关系模型扩展贝叶斯框架, 并用基于期望最大化的直接指定方法来学习该概率模型.Alqadah等人[34]提出了一种利用低方差和形式概念分析优势相组合的方法, 以发现在部分实验条件下具有相同表达趋势的基因.为了便于OPSM的查询, Jiang等人[38]提出了带有行列表头的前缀树索引pIndex, 同时给出4种OPSM查询方法.
3) 约束型双聚类
目前, 该问题的相关研究相对较少, 是一种对挖掘与分析基因表达数据的新方法.Pensa等人[35, 36]提出了一种从局部到整体的方法来建立间隔约束的二分分区.该方法是通过扩展从0/1数据集中提取出来的一些局部模式来实现的.其基本思想是:首先, 将间隔约束转换成一个放松的局部模式; 然后, 利用k均值算法来获得一个局部模式的分区; 最后, 对上述分区做后续处理来确定数据之上的协同聚类结构.随后, Pensa等人[37]对文献[35, 36]进行了扩展, 主要的不同点有:(ⅰ)作者同时在行列之上应用目标函数来评价双聚类的好坏; (ⅱ)新工作[37]将文献[35, 36]中的数据从0/1矩阵扩展到了实数数据; (ⅲ)提升了must-link与cannot-link两类约束在行列之上的处理性能.Tseng等人[56]提出了基于相关约束完整链接的约束型双聚类方法.
6 结论针对基因表达数据中保序子矩阵的约束查询问题, 提出了适用于不同情形的索引和查询方法, 即提出了基于数字签名和Trie的基因序列与实验条件序列索引方法, 以及自顶向下与自底向上相结合的查询方法.索引方法极大地减小了索引的数据量并提升了索引速度; 查询方法方便了不同类型的查询目的, 且提升了查询性能.在真实数据集上的实验结果证明了所提出的方法具有很好的查询精确性和良好的可扩展性.
致谢 在此, 向百忙之中评审我们论文的专家学者和对本文的工作给予支持与建议的同行表示感谢.| [1] | Ben-Dor A, Chor B, Karp R, Yakhini Z. Discovering local structure in gene expression data:The order-preserving submatrix problem. In:Proc. of the 6th Annual Int'l Conf. on Computational Biology (RECOMB). ACM Press, 2002. 49-57.[doi:10.1145/565196.565203] |
| [2] | Hartigan JA. Direct clustering of a data matrix. Journal of the American Statistical Association, 1972, 67(337): 123–129 . [doi:10.1080/01621459.1972.10481214] |
| [3] | Cheng Y, Church GM. Biclustering of expression data. In:Proc. of the 8th Int'l Conf. on Intelligent Systems for Molecular Biology (ISMB). AAAI Press, 2000. 93-103. |
| [4] | Yang J, Wang W, Wang H, Yu PS. δ-Clusters:Capturing subspace correlation in a large data sets. In:Proc. of the 18th Int'l Conf. on Data Engineering (ICDE). IEEE Press, 2002. 517-528.[doi:10.1109/ICDE.2002.994771] |
| [5] | Cho H, Dhillon IS, Guan Y, Sra S. Minimum sum-squared residue co-clustering of gene expression data. In:Proc. of the 12th SIAM Int'l Conf. on Data Mining (SDM). SIAM Press, 2004. 114-125.[doi:10.1137/1.9781611972740.11] |
| [6] | Divina F, Aguilar-Ruiz JS. Biclustering of expression data with evolutionary computation. IEEE Trans. on Knowledge and Data Engineering, 2006, 18(5): 590–602 . [doi:10.1109/TKDE.2006.74] |
| [7] | Deodhar M, Gupta G, Ghosh J, Cho H, Dhillon IS. A scalable framework for discovering coherent co-clusters in noisy data. In:Proc. of the 26th Annual Int'l Conf. on Machine Learning (ICML). ACM Press, 2009. 241-248.[doi:10.1145/1553374.1553405] |
| [8] | Cho H. Data transformation for sum squared residue. In:Proc. of the 14th Pacific-Asia Conf. on Advances in Knowledge Discovery and Data Mining (PAKDD). Berlin, Heidelberg:Springer-Verlag, 2010. 48-55.[doi:10.1007/978-3-642-13657-3_8] |
| [9] | Odibat O, Reddy CK. A generalized framework for mining arbitrarily positioned overlapping co-clusters. In:Proc. of the 19th SIAM Int'l Conf. on Data Mining (SDM). SIAM Press, 2011. 343-354.[doi:10.1137/1.9781611972818.30] |
| [10] | Ayadi W, Elloumi M, Hao JK. BicFinder:A biclustering algorithm for microarray data analysis. Knowledge and Information Systems, 2012, 30(2): 341–358 . [doi:10.1007/s10115-011-0383-7] |
| [11] | Truong DT, Battiti R, Brunato M. Discovering non-redundant overlapping biclusters on gene expression data. In:Proc. of the 13th IEEE Int'l Conf. on Data Mining (ICDM). IEEE Press, 2013. 747-756.[doi:10.1109/ICDM.2013.36] |
| [12] | Ayadi W, Hao JK. A memetic algorithm for discovering negative correlation biclusters of DNA microarray data. Neurocomputing, 2014, 145: 14–22 . [doi:10.1016/j.neucom.2014.05.074] |
| [13] | Chen S, Liu J, Zeng T. MMSE:A generalized coherence measure for identifying linear patterns. In:Proc. of the IEEE Int'l Conf. on Bioinformatics and Biomedicine (BIBM). IEEE Press, 2014. 489-492.[doi:10.1109/BIBM.2014.6999206] |
| [14] | Denitto M, Farinelli A, Bicego M. Biclustering gene expressions using factor graphs and the max-sum algorithm. In:Proc. of the 24th Int'l Conf. on Artificial Intelligence (IJCAI). AAAI Press, 2015. 925-931. |
| [15] | Wang H, Wang W, Yang J, Yu PS. Clustering by pattern similarity in large data sets. In:Proc. of the 28th ACM SIGMOD Int'l Conf. on Management of Data (SIGMOD). ACM Press, 2002. 394-405.[doi:10.1145/564691.564737] |
| [16] | Liu J, Wang W. OP-Clustering by tendency in high dimensional space. In:Proc. of the 3th IEEE Int'l Conf. on Data Mining (ICDM). IEEE Press, 2003. 187-194.[doi:10.1109/ICDM.2003.1250919] |
| [17] | Wang H, Pei J, Yu PS. Pattern-Based similarity search for microarray data. In:Proc. of the 11th ACM SIGKDD Int'l Conf. on Knowledge Discovery and Data Mining (KDD). ACM Press, 2005. 814-819.[doi:10.1145/1081870.1081978] |
| [18] | Kriegel HP, Kroger P, Renz M, Wurst SHR. A generic framework for efficient subspace clustering of high-dimensional data. In:Proc. of the 5th IEEE Int'l Conf. on Data Mining (ICDM). IEEE Press, 2005. 250-257.[doi:10.1109/ICDM.2005.5] |
| [19] | Jiang D, Pei J, Zang A. A general approach to mining quality pattern-based clusters from expression data. In:Proc. of the 10th Int'l Conf. on Database Systems for Advanced Applications (DASFAA). Springer-Verlag, 2005. 188-200.[doi:10.1007/11408079_18] |
| [20] | Gao BJ, Griffith OL, Ester M, Jones SJM. Discovering significant OPSM subspace clusters in massive gene expression data. In:Proc. of the 12th ACM SIGKDD Int'l Conf. on Knowledge Discovery and Data Mining (KDD). ACM Press, 2006. 922-928.[doi:10.1145/1150402.1150529] |
| [21] | Zhang X, Wang W. An efficient algorithm for mining coherent patterns from heterogeneous microarrays. In:Proc. of the 19th Int'l Conf. on Scientific and Statistical Database Management (SSDBM). IEEE Computer Society Press, 2007. 32.[doi:10.1109/SSDBM.2007.30] |
| [22] | Yan L, Sun Z, Wu Y, Zhang B. Biclustering nonlinearly correlated time series gene expression data. Journal of Computer Research and Development, 2008, 45(11): 1865–1873 (in Chinese with English abstract). http://www.cnki.com.cn/Article/CJFDTOTAL-JFYZ200811008.htm |
| [23] | Zhang M, Wang W, Liu J. Mining approximate order preserving clusters in the presence of noise. In:Proc. of the 24th Int'l Conf. on Data Engineering (ICDE). IEEE Press, 2008. 160-168.[doi:10.1109/ICDE.2008.4497424] |
| [24] | Chui CK, Kao B, Yip KY, Lee SD. Mining order-preserving submatrices from data with repeated measurements. In:Proc. of the 8th IEEE Int'l Conf. on Data Mining (ICDM). IEEE Press, 2008. 133-142.[doi:10.1109/ICDM.2008.12] |
| [25] | Zhao Y, Yu J, Wang G, Chen L, Wang B, Yu G. Maximal subspace coregulated gene clustering. IEEE Trans. on Knowledge and Data Engineering, 2008, 20(1): 83–98 . [doi:10.1109/TKDE.2007.190670] |
| [26] | Trapp AC, Prokopyev OA. Solving the order-preserving submatrix problem via integer programming. INFORMS Journal on Computing, 2010, 22(3): 387–400 . [doi:10.1287/ijoc.1090.0358] |
| [27] | Fang Q, Ng W, Feng J. Discovering significant relaxed order-preserving submatrices. In:Proc. of the 16th ACM SIGKDD Int'l Conf. on Knowledge Discovery and Data Mining. ACM Press, 2010. 433-442.[doi:10.1145/1835804.1835861] |
| [28] | Fang Q, Ng W, Feng J, Li Y. Mining bucket order-preserving submatrices in gene expression data. IEEE Trans. on Knowledge and Data Engineering, 2012, 24(12): 2218–2231 . [doi:10.1109/TKDE.2011.180] |
| [29] | Gao BJ, Griffith OL, Ester M, Xiong H, Zhao Q, Jones SJM. On the deep order-preserving submatrix problem:A best effort approach. IEEE Trans. on Knowledge and Data Engineering, 2012, 24(2): 309–325 . [doi:10.1109/TKDE.2010.244] |
| [30] | Yip KY, Kao B, Zhu X, Chui CK, Lee SD, Cheung DW. Mining order-preserving submatrices from data with repeated measurements. IEEE Trans. on Knowledge and Data Engineering, 2013, 25(7):1587-1600.[doi:10.1109/TKDE.2011.167] |
| [31] | An P. Research on biclustering methods for gene expression data analysis[MS. Thesis]. Suzhou:Soochow University, 2013(in Chinese with English abstract).http://cdmd.cnki.com.cn/Article/CDMD-10285-1013229909.htm |
| [32] | Fang Q, Ng W, Feng J, Li Y. Mining order-preserving submatrices from probabilistic matrices. ACM Trans. on Database Systems, 2014, 39(1) . [doi:10.1145/2533712] |
| [33] | Cho S, Na JC, Park K, Sim JS. A fast algorithm for order-preserving pattern matching. Information Processing Letters, 2015, 115(2): 397–402 . [doi:10.1016/j.ipl.2014.10.018] |
| [34] | Alqadah F, Bader JS, Anand R, Reddy CK. Query-Based biclustering using formal concept analysis. In:Proc. of the 12th SIAM Int'l Conf. on Data Mining (SDM). SIAM Press, 2012. 648-659.[doi:10.1137/1.9781611972825.56] |
| [35] | Pensa RG, Robardet C, Boulicaut JF. Towards constrained co-clustering in ordered 0/1 data sets. In:Proc. of the 16th Int'l Symp. on Methodologies for Intelligent Systems (ISMIS). Springer-Verlag, 2006. 425-434.[doi:10.1007/11875604_49] |
| [36] | Pensa RG, Robardet C, Boulicaut JF. Constraint-Driven co-clustering of 0/1 data. In:Proc. of the Constrained Clustering:Advances in Algorithms, Data Mining and Knowledge Discovery Series. 2008. 123-148.[doi:10.1201/9781584889977.ch6] |
| [37] | Pensa RG, Boulicaut JF. Constrained co-clustering of gene expression data. In:Proc. of the 8th SIAM Int'l Conf. on Data Mining (SDM). SIAM Press, 2008. 25-36.[:doi10.1137/1.9781611972788.3] |
| [38] | Jiang T, Li Z, Chen Q, Li K, Wang Z, Pan W. Towards order-preserving submatrix search and indexing. In:Proc. of the 20th Int'l Conf. on Database Systems for Advanced Applications (DASFAA). Part Ⅱ. Berlin, Heidelberg:Springer-Verlag, 2015. 309-326.[doi:10.1007/978-3-319-18123-3_19] |
| [39] | Helmer S, Moerkotte G. Evaluation of main memory join algorithm for joins with subset join predicates. In:Proc. of the 23rd Int'l Conf. on Very Large Database (VLDB). ACM Press, 1997. 386-395. |
| [40] | KiWi Software 1.0. 2012. http://www.bcgsc.ca/platform/bioinfo/ge/kiwi/ |
| [41] | Broad Institute. Datasets.rar and 5q_gct_file.gct. 1999. http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi |
| [42] | Jiang T, Li Z, Chen Q, Wang Z, Li K, Wang Z. Parallel partitioning and mining gene expression data with butterfly network. In:Proc. of the 24th Int'l Conf. on Database and Expert Systems Applications (DEXA). Part Ⅰ. Berlin, Heidelberg:Springer-Verlag, 2013. 129-144.[doi:10.1007/978-3-642-40285-2_13] |
| [43] | Jiang T, Li Z, Chen Q, Wang Z, Li K, Pan W. OMEGA:An order-preserving submatrix mining, indexing and search. In:Proc. of the European Conf. on Machine Learning and Principles and Practice of Knowledge Discovery in Databases (ECML/PKDD). Part Ⅲ. Berlin, Heidelberg:Springer-Verlag, 2015. 303-307.[doi:10.1007/978-3-319-23461-8_35] |
| [44] | Sim K, Gopalkrishnan V, Zimek A, Cong G. A survey on enhanced subspace clustering. Data Mining and Knowledge Discovery, 2013, 26: 332–397 . [doi:10.1007/s10618-012-0258-x] |
| [45] | Kriegel HP, Kroger P, Zimek A. Clustering of high-dimensional data:A survey on subspace clustering, pattern-based clustering, and correlation clustering. ACM Trans. on Knowledge Discovery from Data, 2009, 3(1): 1–58 . [doi:10.1145/1497577.1497578] |
| [46] | Yue F, Sun L, Wang K, Wang Y, Zuo W. State-of-the-Art of cluster analysis of gene expression data. Acta Automatica Sinica, 2008, 34(2): 113–120 (in Chinese with English abstract). [doi:10.3724/SP.J.1004.2008.00113] |
| [47] | Jiang D, Tang C, Zhang AD. Cluster analysis for gene expression data:A survey. IEEE Trans. on Knowledge and Data Engineering, 2004, 16(11): 1370–1386 . [doi:10.1109/TKDE.2004.68] |
| [48] | Madeira SC, Oliveira AL. Biclustering algorithms for biological data analysis:A survey. IEEE Trans. on Computational Biology and Bioinformatics, 2004, 1(1): 24–45 . [doi:10.1109/TCBB.2004.2] |
| [49] | Smet RD, Marchal K. An ensemble method for querying gene expression compendia with experimental lists. In:Proc. of the 2010 IEEE Int'l Conf. on Bioinformatics and Biomedicine (BIBM). IEEE Press, 2010. 314-318.[doi:10.1109/BIBM.2010.5706583] |
| [50] | Zou Q, Guo M, Liu Y, Wang J. A classification method for class-imbalanced data and its application on bioinformatics. Journal of Computer Research and Development, 2010, 47(8): 1407–1414 (in Chinese with English abstract). http://www.cnki.com.cn/Article/CJFDTOTAL-JFYZ201008016.htm |
| [51] | Zou Q, Li X, Jiang W, Lin Z, Li G, Chen K. Survey of mapreduce frame operation in bioinformatics. Briefings in Bioinformatics, 2014, 15(4): 637–647 . [doi:10.1093/bib/bbs088] |
| [52] | Chen W, Cheng Y, Zhang S, Pan Q. Heuristic clustering method based on neighbor-seeds for 454 sequencing data. Ruan Jian Xue Bao/Journal of Software, 2014, 25(5):929-938(in Chinese with English abstract). http://www.jos.org.cn/1000-9825/4547.htm[doi:10.13328/j.cnki.jos.004547]http://www.jos.org.cn/ch/reader/view_abstract.aspx?flag=1&file_no=4547&journal_id=jos |
| [53] | Zou Q, Hu Q, Guo M, Wang G. HAlign:Fast multiple similar DNA/RNA sequence alignment based on the centre star strategy. Bioinformatics, 2015, 31(15): 2475–2481 . [doi:10.1093/bioinformatics/btv177] |
| [54] | Dhollander T, Sheng QZ, Lemmens K, Moor BD, Marchal K, Moreau Y. Query-Driven module discovery in microarray data. Bioinformatics, 2007, 23(19): 2573–2580 . [doi:10.1093/bioinformatics/btm387] |
| [55] | Zhao H, Cloots L, Bulcke TV, Wu Y, Smet RD, Storms V, Meysman P, Engleen K, Marchal K. Query-Based biclustering of gene expression data using probabilistic relational models. BMC Bioinformatics, 2011, 12(s1): S37 . [doi:10.1186/1471-2105-12-S1-S37] |
| [56] | Tseng VS, Chen LC, Kao CP. Constrained clustering for gene expression data mining. In:Proc. of the 12th Pacific-Asia Conf. on Advances in Knowledge Discovery and Data Mining (PAKDD). Berlin, Heidelberg:Springer-Verlag, 2008. 759-766.[doi:10.1007/978-3-540-68125-0_73] |
| [22] | 闫雷鸣, 孙志挥, 吴英杰, 张柏礼. 联合聚类非线性相关的时序基因表达数据. 计算机研究与发展, 2008, 45(11): 1865–1873. http://www.cnki.com.cn/Article/CJFDTOTAL-JFYZ200811008.htm |
| [31] | 安平. 基因表达数据的双聚类分析方法研究[硕士学位论文]. 苏州: 苏州大学, 2013. |
| [46] | 岳峰, 孙亮, 王宽全, 王永吉, 左旺孟. 基因表达数据的聚类分析研究进展. 自动化学报, 2008, 34(2): 113–120. [doi:10.3724/SP.J.1004.2008.00113] |
| [50] | 邹权, 郭茂祖, 刘扬, 王峻. 类别不平衡的分类方法及在生物信息学中的应用. 计算机研究与发展, 2010, 47(8): 1407–1414. http://www.cnki.com.cn/Article/CJFDTOTAL-JFYZ201008016.htm |
| [52] | 陈伟, 程咏梅, 张绍武, 潘泉. 邻域种子的启发式454序列聚类方法. 软件学报, 2014, 25(5): 929-938. http://www.jos.org.cn/1000-9825/4547.htm[doi:10.13328/j.cnki.jos.004547] |
 2017, Vol. 28
2017, Vol. 28


