2. 计算智能与中文信息处理教育部重点实验室(山西大学), 山西 太原 030006
2. Key Laboratory of Computation Intelligence and Chinese Information Processing(Shanxi University), Ministry of Education, Taiyuan 030006, China
近年来, 对各种复杂网络的研究[1-3]是许多领域的研究热点之一, 如生物网络、社交网络、电子邮件网络、引文网络等已成为众多学者的主要研究对象.复杂网络中的社区发现不仅有助于深入研究整个网络的功能模块及其演化, 而且能够准确地理解并分析复杂系统的拓扑结构及动力学特性, 因此具有十分重要的理论意义和应用价值[4, 5].
目前, 针对复杂网络中的社区发现问题, 研究较多的是无向无权网络, 包括基于图划分的聚类算法[6]、基于谱分析的聚类算法[7]、基于层次的聚类算法[8]、基于密度的聚类算法[9, 10]等.其中, 基于密度的聚类算法通过搜索网络中稠密子图[11], 能够较好地发现网络中的功能模块, 在社区发现中得到了广泛应用.2003年Bader等人提出的MCODE算法[12]、2005年Palla等人[13]提出的派系过滤算法(clique percolation method, 简称CPM)、2006年Saito等人[14]提出的k-dense算法、2009年Shen等人提出的EAGLE算法[15]等将网络拓扑结构作为社区划分的依据, 对无权网络进行社区划分.
然而, 现实复杂网络中的边或顶点中往往包含有一些重要的先验信息, 如, 高通量实验所得到的蛋白质互作用网络中, 边权重往往代表实验可信度; 合作网络中的边权重通常代表合作对象的连接紧密程度.2008年, Blondel等人基于模块性最优化提出了启发式算法BGLL[16].2009年, Liu等人[17]提出的CMC算法通过迭代评分的方法给每条边赋予权重, 然后通过搜索网络中的所有极大团并计算其加权密度, 最后依据极大团的加权密度定义极大团之间的相互连接度, 进而合并极大团得到网络中的社区结构.2011年, Lee等人[18]提出的MDOS算法在加权网络中通过选取种子节点, 并依据子图函数逐步扩展得到稠密子图.Wang等人基于边聚集系数局部度量提出了适用于无权网络和加权网络的HC-PIN算法[19].2014年, Ren等人[10]也通过定义边权重将无权网络转化为加权网络, 然后结合子图的密度性和模块性定义适应度函数, 给出了基于局部适应度的LF_PIN算法.但是, 这些学者仅考虑基于拓扑结构定义网络中边的权重或者仅考虑网络中具有现实意义的边权重进行社区发现.如何将具有现实意义的边权重与网络拓扑结构相结合, 作为社区发现算法的依据, 进而发现复杂网络中的重叠社区, 是当今社区发现算法研究热点之一.
针对加权网络的社区发现问题, 提出了一种基于种子扩展策略的重叠社区发现算法(overlapping community detection on weighted networks, 简称OCDW).首先给出了一种综合考虑网络自身边信息和网络拓扑结构信息的边权重定义方式, 并基于此定义节点的加权度, 选择权重最大的节点作为社区种子节点; 进而给出了基于社区评估函数的种子扩展策略和权重更新方式, 迭代得到加权稠密子图; 最后, 将重叠率比较高的稠密子图合并为加权中心社区, 并根据节点对社区的归属度b(v, C)来度量节点和社区的连接倾向程度, 将未聚类节点分配到加权中心社区中, 从而得到最终的社区发现结果.
1 背景知识通常, 一个复杂网络可以用图G=(V, E)来表示, 其中, 节点集V={v1, v2, …, vn}, n=|V|; 边集E中每条边ei, j对应V中一对顶点(vi, vj)之间的连接关系, m=|E|.节点v的邻域NG(v)={u|(u, v)∈E}, 在不引起混淆的情况下, 简记为N(v).
N(v)中的节点称为节点v的相邻点.节点v的度记为kv.除非特别指明, 本文仅考虑简单无向图, 即kv=|N(v)|.令U⊆V(G), 用[U]G表示G的节点子集U的导出子图, 记为[U].令

|
Fig. 1 Two classical complex networks 图 1 两个典型复杂网络实例 |
为了合理地对复杂网络中的节点进行社区发现, 将顶点间的相似性作为衡量节点连接紧密程度的重要标准.Jaccard度量[20]认为:网络图中两节点间的公共邻接点越多, 其在结构上就越相似, 连接也就越紧密, 如公式(1)所示.
| $ Jaccard({{v}_{i}}, {{v}_{j}})=\frac{|N({{v}_{i}})\cap N({{v}_{j}})|}{|N({{v}_{i}})\cup N({{v}_{j}})|} $ | (1) |
Hub Promoted和Hub Depressed度量[20]考虑偏好连接对连接紧密度的影响, 如公式(2)、公式(3)所示.
| $ HP({{v}_{i}}, {{v}_{j}})=\frac{|N({{v}_{i}})\cap N({{v}_{j}})|}{\min \{|N({{v}_{i}})|, |N({{v}_{j}})|\}} $ | (2) |
| $ HD({{v}_{i}}, {{v}_{j}})=\frac{|N({{v}_{i}})\cap N({{v}_{j}})|}{\max \{|N({{v}_{i}})|, |N({{v}_{j}})|\}} $ | (3) |
由于复杂网络构建过程中实验方法的偏差会导致网络拓扑结构中有大量噪声, 如由高通量实验构建的蛋白质互作用网络中存在大量的假阳性和假阴性数据, 为了更准确地表示网络中边的连接紧密程度, 应该综合考虑网络拓扑结构和复杂网络中的边信息.
2 边权重定义现实网络中, 边的特性可以通过边权重来体现.如社会网络中, 边权重可以表示个体之间连接的紧密及强弱关系; 科学家合作网络中的边权重, 可以表示科学家之间合作的次数; 而蛋白质互作用网络中, 边权重往往表示由高通量实验所产生的蛋白质互作用边的可信度[23].在网络中进行社区发现, 不仅需要考虑网络的拓扑结构, 而且也要考虑节点间边连接的现实意义.
为了能够更加准确地度量节点之间的关联强度, 需要先对网络中边的权重进行定义, 如公式(4)所示.
| $ {w}'({{v}_{i}}, {{v}_{j}})=\alpha \cdot \frac{|N({{v}_{i}})\cap N({{v}_{j}}){{|}^{2}}}{\min {{\{|N({{v}_{i}})|, |N({{v}_{j}})|\}}^{2}}}+\beta \cdot \frac{|N({{v}_{i}})\cap N({{v}_{j}}){{|}^{2}}}{\max {{\{|N({{v}_{i}})|, |N({{v}_{j}})|\}}^{2}}}+(1-\alpha -\beta )\cdot u({{v}_{i}}, {{v}_{j}}) $ | (4) |
其中, N(vi)表示节点vi邻接节点的集合, N(vi)∩N(vj)表示节点vi和vj的公共邻接节点集合, u(vi, vj)表示节点vi和vj在真实网络中边的权重.前两项体现了节点vi和vj在网络拓扑结构方面的相似程度, 第3项体现了边的真实网络权重.将图G的边
| $ w({{v}_{i}}{{v}_{j}})=\left\{ \begin{array}{*{35}{l}} \varepsilon +\tfrac{1-\varepsilon }{{{{{w}'}}_{avg}}}\cdot {w}'({{v}_{i}}{{v}_{j}}),&{{v}_{i}}{{v}_{j}}\in E(G) \\ 0,&{{v}_{i}}{{v}_{j}}\notin E(G) \\ \end{array} \right. $ | (5) |
其中,
基于上一节给出的边权重定义, 本文给出一种基于“种子节点扩展策略”的社区发现算法OCDW, 其中包括种子节点的选取、种子扩展过程和社区合并与后处理这3个基本过程.
● 首先, 根据与节点关联的边权重计算节点权重, 选择权重较大的节点作为种子节点;
● 其次, 以种子节点为初始点, 根据节点对子图的适应度函数将种子节点扩展成局部稠密子图;
● 当所有局部稠密子图构造结束后, 将这些子图进行合并处理, 得到最终的社区发现结果.
3.1 种子节点的选取种子节点应该位于最终社区对应子图的拓扑中心位置, 通常, 这类节点在网络中重要性比较高且位于网络中相对稠密的区域中.种子节点的选择要有足够的代表性, 能够使得社区发现算法在尽可能少的迭代过程中给出网络中大多数节点的社区归属, 因此, 种子节点之间在网络结构上应该相距较远.基于这些原则, 将网络中节点vi∈V(G)的加权度作为其权重定义, 如公式(6)所示:
| $ wd({{v}_{i}})=\sum\nolimits_{{{v}_{j}}\in N({{v}_{i}})}{w({{v}_{i}}{{v}_{j}})\cdot {{k}_{j}}} $ | (6) |
其中, N(vi)表示节点vi的邻接点集合, kj表示节点vj的度.节点vi的加权度wd(vi)与其邻点的度数以及连边的权重成正相关, 反映了vi在网络中的重要程度.此处选择权重最大的节点作为第1个种子节点.
为使网络中尽可能多的节点有社区归属, 在选择下一个社区的种子节点时, 应该降低那些已经成为种子的节点选择概率.因此, 本算法在找到一个稠密子图之后, 减少该子图中的边权重, 并重新计算相关节点的加权度.若某节点的加权度变化过大, 则不应选作其他社区的种子节点.因此定义节点的加权度变化率, 如公式(7)所示.
| $ cr(v)=1-\frac{w{d}'({{v}_{i}})}{wd({{v}_{i}})} $ | (7) |
其中, wd(v)表示节点v在原始网络中的加权度, wd'(v)表示节点v在更新子图边权重之后的加权度.如果节点v的加权度变化率cr(v) > θ, 则节点v不能再次被选为种子节点, 本文设定θ=0.3.
图 2为空手道俱乐部和海豚社交网络在算法执行完之后的所有种子节点, 用黑色节点表示.可以看出, 图中种子节点选择数量适中且具有较好的代表性.
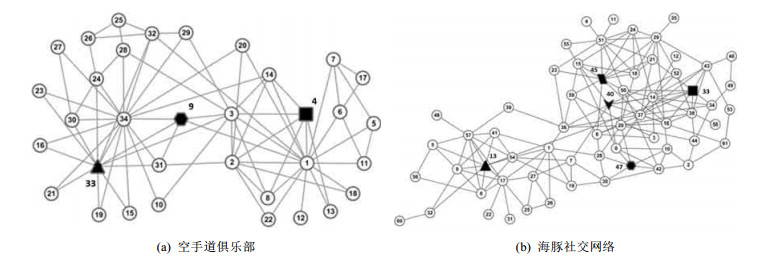
|
Fig. 2 Seed nodes in Zachary's karate club and dolphins social network obtained by OCDW 图 2 OCDW算法在Zachary空手道俱乐部和海豚社交网络中得到的种子节点 |
3.2 种子扩展策略
为了得到当前种子节点附近的一个稠密子图, 首先给出子图的适应度函数以评价其稠密程度.假设S是图G的一个连通子图, VS表示子图S的顶点集, ES表示S的边集, 令ns=|VS|, ms=|ES|.在S中添加
| $ f(S)=\sum\nolimits_{{{v}_{i}}{{v}_{j}}\in E(S)}{w({{v}_{i}}{{v}_{j}})}-\frac{1}{2|E(G)|}[{{n}_{s}}({{n}_{s}}-1)-2{{m}_{s}}]\cdot \sum\nolimits_{{{v}_{i}}{{v}_{j}}\in E(G)}{w({{v}_{i}}{{v}_{j}})} $ | (8) |
显然, f(S)越大, 子图S在给定权重意义下越稠密.根据定义可知, 若S中只有1个节点, 则f(S)=0.
对节点v∉Vs, 定义v对S的适应度函数δS(v)=f(S∪{v})-f(S).在种子节点扩展步骤, 将满足δS(v) > 0且适应度函数最大的节点扩展到当前子图S中, 此过程迭代进行, 直至找不到满足条件的节点, 从而得到一个当前稠密子图.图 3为空手道俱乐部和海豚社交网络通过算法依据子图适应度函数将种子节点扩展得到的稠密子图, 不同形状的灰色节点表示不用的稠密子图.

|
Fig. 3 Weighted dense subgraphs in Zachary's karate club and dolphins social network obtained by OCDW 图 3 OCDW算法在空手道俱乐部和海豚社交网络中得到的加权稠密子图 |
3.3 合并优化社区与未聚类节点处理
通过上述步骤之后, 我们可以找到很多稠密子图, 但是存在一些稠密子图之间重叠节点较多的情况, 需要将重叠节点较多的稠密子图进行合并, 得到最终社区发现结果.如图 3(a)所示, 节点{9, 31, 33, 34}(图中六边形节点)构成的稠密子图与由节点{24, 30, 33, 34}(图中三角形节点)构成的稠密子图之间有一半节点重合, 需要进行合并.本文中判断两个稠密子图合并的条件为两个子图间重叠节点数不小于较小稠密子图节点数一半时, 将二者合并得到中心社区, 称为加权中心社区.图 4(a)和图 4(b)分别给出了空手道俱乐部和海豚社交网络合并后所得到的加权中心社区.

|
Fig. 4 Weighted core communities in Zachary's karate club and dolphins social network obtained by OCDW 图 4 OCDW算法在空手道俱乐部和海豚社交网络中得到的加权中心社区 |
OCDW算法得到的加权中心社区包括了原始网络中的一些局部稠密子图, 但并未完全覆盖整个网络, 仍然存在一些未聚类节点(如图 4中的空白节点所示).理想的社区发现结果应该使网络中尽量多的节点有最终的社区归属, 因此, 根据节点与中心社区连接紧密程度定义未聚类对中心社区的归属度, 如公式(9)所示.
| $ b(v, C)=\gamma \cdot \frac{\sum\nolimits_{x\in N(v)\cap C}{w(x, v)}}{\sum\nolimits_{x\in C}{w(x, v)}}+(1-\gamma )\cdot \frac{\sum\nolimits_{x\in N(v)\cap C}{wd(x)}}{\sum\nolimits_{x\in C}{wd(x)}} $ | (9) |
基于公式(9)给出的节点对社区归属度的定义, 迭代地将未聚类节点划分到已有的中心社区中, 对中心社区进行扩展, 得到更加合理的聚类结果(详细的中心社区扩展策略见算法1的步骤7和步骤8).图 5给出了空手道俱乐部数据和海豚社交网络的最终聚类结果.
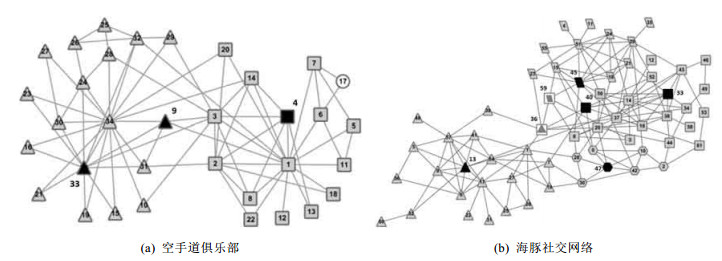
|
Fig. 5 Final communities in Zachary's karate club and dolphins social network obtained by OCDW 图 5 OCDW算法在空手道俱乐部和海豚社交网络中得到的最终聚类结果 |
3.4 算法描述
算法1.加权网络重叠社区发现算法.
输入:网络G=(V, E, U), 其中, V和E分别表示G的节点集和边集, U是G的权重矩阵.
输出:社区集合C={C1, C2, …, Ck}.
过程:
步骤1.令n=|V|, m=|E|, t=0, F=V,
步骤2.如果α < 0, 则转步骤9.令C=∅, β=0.7-α.
根据公式(4)、公式(5)计算图G中每条边的综合权重矩阵Wn×n(G).
步骤3.根据公式(6)计算V中节点的加权度wd(v), 假设wd(v1)≥wd(v2)≥…≥wd(vn).
步骤4.如果F为空集, 则转步骤5;否则, 令t=t+1, Ct=∅.
步骤4.1.选择集合F中权重最大的一个节点v, 令F=F-{v}, 且Ct=Ct∪{v}.
步骤4.2.令N(Ct)表示当前集合Ct的邻接点集合, 对N(Ct)中的每个节点u, 计算u对Ct的影响度
步骤4.3.令节点x是N(Ct)中对Ct影响度最大的节点且满足
步骤4.4.若节点x存在, 则令Ct=Ct∪{x}, 则转步骤4.3.
步骤4.5.若|Ct|≤3, 令t=t-1, 转步骤4.
步骤4.6.将Ct的导出子图[Ct]识别为稠密子图.对[Ct]中的每条边xy∈E([Ct]), 使得
步骤4.7.对每个节点x∈Ct, 令
步骤5.对每一个稠密子图Ci∈{C1, …, Ct}, 计算Ci与其他子图的重叠度.若Ci与子图Cj(i < j)节点的重叠度|Ci∩Cj|/min{|Ci|, |Cj|}≥0.5, 则令Ci=Ci∪Cj.
步骤6.对合并后的稠密子图重新编号, 得到中心社区集合为
步骤7.令集合
步骤8.令t=t+1, 对每个社区
步骤8.1.
步骤8.2.令μt=μt-1-0.1,
步骤9.结束, 输出社区集合
加权网络重叠社区发现算法OCDW给出了基于贪心搜索策略的社区发现算法, 其中, 步骤1~步骤3实现了对网络中节点和边权重赋初值, 平均时间复杂度与网络中的总边数成正比, 即O(c|E|).步骤4迭代实现了种子节点的选择与获得中心社区, 每次迭代会遍历网络中的所有边, 代价为O(|E|).中心社区扩展迭代次数与中心点的选择密切相关, 假设迭代次数为t, 则步骤4的总代价为O(t|E|).步骤5实现了中心社区合并操作, 假设有s(通常s < < |V|)个中心社区, 则时间代价为O(s2).步骤7、步骤8实现了未聚类点分配过程, 时间代价至多为O(|V|).综合以上分析, 算法OCDW的总平均时间复杂度为O(ct|E|+|V|), 其中, c和t是常数.
4 实验与结果分析在空手道俱乐部(zachary’s karate club)[24]、海豚社交网络(dolphins social network)[25]、大学生足球网络(American college football network)[26]、Polbooks(books about US politics dataset, http://www.orgnet.com/)、Adjnoun[26]和Email[27]这6个无权网络数据集以及Les Misėrables[28]、合作网络(NetScience)[28]和Hep-th[29]这3个加权网络数据集上对算法OCDW进行评测, 数据基本情况见表 1.
| Table 1 Experimental datasets 表 1 实验数据集 |
4.1 评价指标
设OCDW算法的社区发现结果为C={C1, …, Cs}, 有标签数据集上的原始划分结果为O={O1, …, Ot}.对集合Ci和Oj(1≤i≤s, 1≤j≤t), 令Ti, j=|Ci∩Oj|,
(1) 准确度(Acc)[30]用来度量两个参照集的匹配程度, 其定义如公式(10)所示.
| $ Acc=\sqrt{\frac{\sum\nolimits_{i=1}^{S}{{{\max }_{j}}\{{{T}_{i}}_{, j}\}}}{\sum\nolimits_{i=1}^{S}{{{b}_{i}}}}\cdot \frac{\sum\nolimits_{j=1}^{t}{{{\max }_{i}}\{{{T}_{i}}_{, j}\}}}{\sum\nolimits_{j=1}^{t}{|{{O}_{j}}|}}} $ | (10) |
(2) 分离度(Sep)[31]用来度量社区发现结果与原始数据划分的一致性, 其定义如公式(11)所示.
| $ Sep=\sqrt{\frac{{{\left( \sum\nolimits_{i=1}^{S}{\sum\nolimits_{j=1}^{t}{Se{{p}_{i, j}}}} \right)}^{2}}}{s\times t}} $ | (11) |
其中,
(3) 度量(F-measure)[32]通过精度(precision)和召回率(recall)的调和平均值来度量社区发现结果与原始社区的匹配程度, 其定义如公式(12)所示(此处θ=0.25).
| $ F-measure=\frac{2\cdot Precision\cdot Recall}{Precision+Recall} $ | (12) |
其中,
| $ \begin{matrix} Precision=\frac{|\{{{C}_{i}}\in C:\, \exists {{O}_{j}}\in O\wedge NA({{C}_{i}}, {{O}_{j}})\ge \theta \}|}{|C|}, \\ Recall=\frac{|\{{{C}_{j}}\in C:\, \exists {{O}_{i}}\in O\wedge NA({{C}_{i}}, {{O}_{j}})\ge \theta \}|}{|O|}, \\ NA({{C}_{i}}, {{O}_{j}})=\frac{{{({{T}_{i, j}})}^{2}}}{|{{C}_{i}}|\cdot |{{O}_{j}}|}. \\ \end{matrix} $ |
(4) 标准互信息(NMI)通过熵来度量两个数据分布的吻合程度, 此处用来度量社区发现的结果与原始社区划分的吻合程度, 其定义如公式(13)所示.
| $ NMI=\frac{2\sum\nolimits_{i=1}^{S}{\sum\nolimits_{j=1}^{t}{{{T}_{i}}_{, j}\log \frac{n{{T}_{i, j}}}{{{b}_{i}}{{d}_{j}}}}}}{-\sum\nolimits_{i=1}^{s}{{{b}_{i}}\log \frac{{{b}_{i}}}{n}}\sum\nolimits_{j=1}^{t}{{{d}_{j}}\log \frac{{{d}_{j}}}{n}}} $ | (13) |
(5) 调整兰德系数(ARI)是由Hubert and Arabie提出的, 通过统计划分正确的社区中元素的对数来度量算法社区发现结果和原始社区的一致性, 定义如公式(14)所示.
| $ ARI = \frac{{\sum\nolimits_{i = 1}^s {\sum\nolimits_{j = 1}^t {\left( \begin{array}{l} {T_{i,j}}\\ 2 \end{array} \right)} } - \frac{{\sum\nolimits_{i = 1}^s {\left( {\begin{array}{*{20}{c}} {{b_i}}\\ 2 \end{array}} \right)\sum\nolimits_{j = 1}^t {\left( \begin{array}{l} {d_j}\\ 2 \end{array} \right)} } }}{{\left( {\begin{array}{*{20}{c}} n\\ 2 \end{array}} \right)}}}}{{\frac{1}{2}\left[ {\sum\nolimits_{i = 1}^s {\left( \begin{array}{l} {b_i}\\ 2 \end{array} \right) + \sum\nolimits_{j = 1}^t {\left( \begin{array}{l} {d_j}\\ 2 \end{array} \right)} } } \right] - \frac{{\sum\nolimits_{i = 1}^s {\left( \begin{array}{l} {b_i}\\ 2 \end{array} \right)\sum\nolimits_{j = 1}^t {\left( \begin{array}{l} {d_j}\\ 2 \end{array} \right)} } }}{{\left( {\begin{array}{*{20}{c}} n\\ 2 \end{array}} \right)}}}} $ | (14) |
其中, n为网络总节点数.ARI∈[-1, 1], 其值越大, 说明社区发现结果与原始社区越吻合.
(6) 模块性(EQ).本文用Shen等人提出的重叠社区模块性[15]度量算法在无标签数据集上的社区发现质量, 如公式(15)所示.
| $ EQ=\frac{1}{2}\sum\nolimits_{i=1}^{s}{\left( \sum\nolimits_{x\in {{C}_{i}}}{\sum\nolimits_{y(\ne x)\in {{C}_{i}}}{\frac{1}{{{O}_{x}}{{O}_{y}}}\left[{{A}_{xy}}-\frac{{{k}_{x}}{{k}_{y}}}{2m} \right]}} \right)} $ | (15) |
其中, ox表示在最终社区发现结果中节点x属于的社区数, Axy是原始网络邻接矩阵, kx表示节点x的度数, m是原始网络总边数.
4.2 实验结果与分析在5个带社区标签的数据集空手道俱乐部(karate)、海豚社交网络(dolphins)、大学生足球网络(football)、Polbooks和Adjnoun上, 将本文算法OCDW与经典的社区发现算法k-dense[14], CPM[13], MCODE[12], HC-PIN[19], MDOS[18]和BGLL[16], 从F度量、准确度(ACC)、分离度(Sep)、标准互信息(NMI)、调整兰德系数ARI这些方面进行比较, 结果如表 2和图 6所示.
| Table 2 Comparison of OCDW with some classical algorithms 表 2 OCDW与一些经典算法比较结果 |

|
Fig. 6 Comparison results on F-measure, accuracy, separation, NMI and ARI on 5 real network datasets 图 6 在5个真实网络数据集上, F度量、准确度、分离度、标准互信息以及调整兰德系数实验比较结果 |
尽管k-dense算法和CPM算法在一些指标中表现突出, 然而其运行结果中存在大量的未聚类节点.本文算法OCDW能够将网络中的绝大部分节点进行社区指派, 在5个带标签数据集实验中, 各项指标均名列前茅, 表明其聚类结果与网络原始社区最为接近, 具有较高的综合性能.
在4个无标签数据集Email, Les Misérables, NetScience和Hep-th上, 将本文OCDW算法在模块性及运行时间方面与其他算法进行比较, 结果见表 3.由于算法OCDW在不影响聚类效果的前提下对种子节点的选择范围进行了控制, 因此算法的运行时间也得到了很好的改进.综合考虑模块性和时间性能, 本文算法OCDW算法表现良好.
| Table 3 Modularity (EQ) and running time of different algorithms on 4 real network datasets 表 3 在4个真实网络数据及上模块性(EQ)、运行时间指标的比较结果 |
总体而言, 本文提出的OCDW算法在社区发现过程中综合考虑了网络的拓扑结构和原始权重信息, 能够给出贴近现实网络的社区发现结果, 并且具有较好的时间性能和模块性.
5 总结本文提出了一种基于种子-扩展策略的面向复杂网络中加权稠密子图的社区发现算法OCDW, 综合考虑网络拓扑结构和现实网络权重, 对网络中的边权重进行定义; 通过搜索网络中的在加权意义下的相对稠密子图得到加权中心社区; 评估未聚类节点与加权中心社区的归属度, 对未聚类节点进行社区指派.通过与经典的社区发现算法k-dense, CPM, MCODE, HC-PIN, MDOS, BGLL等算法在9个真实网络数据集上进行分析比较, 结果表明, 本文提出的算法OCDW能够将网络中的绝大部分节点进行社区指派, 并在F度量、准确度、分离度、标准互信息、调整兰德系数、模块性及运行时间等方面均表现出较好的性能.
| [1] |
Bai L, Cheng XQ, Liang JY, Guo YK. Fast graph clustering with a new description model for community detection. Information Sciences, 2017: 388–389.
[doi:10.1016/j.ins.2017.01.026] |
| [2] |
Atay Y, Koc I, Babaoglu I, Kodaz H. Community detection from biological and social networks:A comparative analysis of metaheuristic algorithms. Applied Soft Computing, 2017, 50: 194–211.
[doi:10.1016/j.asoc.2016.11.025] |
| [3] |
Liu BY, Wang CR, Wang C, Wang JW, Wang XW, Huang M. Microblog community discovery algorithm based on dynamic topic model with multidimensional data fusion. Ruan Jian Xue Bao/Journal of Software, 2017, 28(2): 246–261(in Chinese with English abstract).
http://www.jos.org.cn/1000-9825/5116.htm [doi:10.13328/j.cnki.jos.005116] |
| [4] |
Huang FL, Yu G, Zhang JL, Li CX, Yuan CA, Lu JL. Mining topic sentiment in micro-blogging based on micro-blogger social relation. Ruan Jian Xue Bao/Journal of Software, 2017, 28(3): 694–707(in Chinese with English abstract).
http://www.jos.org.cn/1000-9825/5157.htm [doi:10.13328/j.cnki.jos.005157] |
| [5] |
Wilson SJ, Wilkins AD, Lin CH, Lua RC, Lichtarge O. Discovery of functional and disease pathway by community detection protein-protein interaction networks. Pacific Symp. on Biocomputing, 2016, 22: 336–347.
http://www.ncbi.nlm.nih.gov/pubmed/27896987 |
| [6] |
Newman MEJ. Community detection and graph partitioning. Europhysics Letters, 2013, 103(2): No.28003.
[doi:10.1209/0295-5075/103/28003] |
| [7] |
Newman MEJ. Spectral methods for community detection and graph partitioning. Physical Review E, 2013, 88(4): No.042822.
[doi:10.1103/PhysRevE.88.042822] |
| [8] |
Lin CC, Kang JR, Chen JY. An integer programming approach and visual analysis for detecting hierarchical community structures in social networks. Information Sciences, 2015, 299: 296–311.
[doi:10.1016/j.ins.2014.12.009] |
| [9] |
Bai XY, Yang PL, Shi XH. An overlapping community detection algorithm based on density peaks. Neurocomputing, 2017, 226: 7–15.
[doi:10.1016/j.neucom.2016.11.019] |
| [10] |
Ren J, Wang JX, Li M, Wang LS. Identifying protein complexes based on density and modularity in protein-protein interaction network. BMC Systems Biology, 2013, 7(Suppl 4): No.S12.
[doi:10.1186/1752-0509-7-S4-S12] |
| [11] |
Li XL, Foo CS, Ng SK. Discovering protein complexes in dense reliable neighborhoods of protein interaction networks. Computational Systems Bioinformatics, 2007, 6: 157–168.
[doi:10.1142/9781860948732_0019] |
| [12] |
Bader GD, Hogue CWV. An automated method for finding molecular complexes in large protein interaction networks. BMC Bioinformatics, 2003, 4(1): No.2.
[doi:10.1186/1471-2105-4-2] |
| [13] |
Palla G, Derényi I, Farkas I, Vicsek T. Uncovering the overlapping community structure of complex networks in nature and society. Nature, 2005, 435(7043): 814–818.
[doi:10.1038/nature03607] |
| [14] |
Saito K, Yamada T, Kazama K. Extracting communities from complex networks by the k-dense method. In:Patist JP, ed. Proc. of the 6th IEEE Int'l Conf. on Data Mining Workshops (ICDMW 2006). Piscataway:IEEE Press, 2006. 300-304.[doi:10.1109/ICDMW.2006.76] |
| [15] |
Shen HW, Cheng XQ, Cai K, Hu MB. Detect overlapping and hierarchical community structure in networks. Physica A, 2009, 388(8): 1706–1712.
[doi:10.1016/j.physa.2008.12.021] |
| [16] |
Blondel VD, Guillaume JL, Lambiotte R, Lefebvre E. Fast unfolding of communities in large networks. Journal of Statistical Mechanics:Theory and Experiment, 2008, 2008(10): 10008–100019.
[doi:10.1088/1742-5468/2008/10/P10008] |
| [17] |
Liu GM, Wong L, Chua HN. Complex discovery from weighted PPI networks. Bioinformatics, 2009, 25(15): 1891–1897.
[doi:10.1093/bioinformatics/btp311] |
| [18] |
Lee AJT, Lin MC, Hsu CM. Mining dense overlapping subgraphs in weighted protein-protein interaction networks. BioSystems, 2011, 103(3): 392–399.
[doi:10.1016/j.biosystems.2010.11.010] |
| [19] |
Wang JX, Li M, Chen JE, Pan Y. A fast hierarchical clustering algorithm for functional modules discovery in protein interaction networks. IEEE/ACM Trans. on Computational Biology and Bioinformatics, 2011, 8(3): 607–620.
[doi:10.1109/TCBB.2010.75] |
| [20] |
Lü LY, Zhou T. Link prediction in complex networks:A survey. Physica A, 2011, 390(6): 1150–1170.
[doi:10.1016/j.physa.2010.11.027] |
| [21] |
Palla G, Barabási AL, Vicsek T. Quantifying social group evolution. Nature, 2007, 446: 664–667.
[doi:10.1038/nature05670] |
| [22] |
Jeong H, Mason SP, Barabási AL, Oltvai ZN. Lethality and centrality in protein networks. Nature, 2001, 411(6833): 41–42.
[doi:10.1038/35075138] |
| [23] |
Wang J, Liang JY, Zheng WP. A graph clustering method for detecting protein complexes. Journal of Computer Research and Development, 2015, 52(8): 1784–1793(in Chinese with English abstract).
[doi:10.7544/issn1000-1239.2015.20150180] |
| [24] |
Zachary WW. An information flow model for conflict and fission in small groups. Journal of Anthropological Research, 1977, 33(4): 452–473.
[doi:10.1086/jar.33.4.3629752] |
| [25] |
Lusseau D, Newman MEJ. Identifying the role that animals play in their social networks. Proc. of the Royal Society B:Biological Sciences, 2004, 271(Suppl 6): S477–S481.
[doi:10.1098/rsbl.2004.0225] |
| [26] |
Newman MEJ. Finding community structure in networks using the eigenvectors of matrices. Physical Review E, 2006, 74(3): No. 036104.
[doi:10.1103/PhysRevE.74.036104] |
| [27] |
Guimerà R, Danon L, Diaz-Guilera A, Giralt F, Arenas A. Self-Similar community structure in a network of human interactions. Physical Review E, 2003, 68(6): No.065103.
[doi:10.1103/PhysRevE.68.065103] |
| [28] |
Newman MEJ, Girvan M. Finding and evaluating community structure in networks. Physical Review E, 2004, 69(2): No.026113.
[doi:10.1103/PhysRevE.69.026113] |
| [29] |
Newman MEJ. The structure of scientific collaboration networks. Proc. of the National Academy of Sciences of the United States of America, 2001, 98(2): 404–409.
[doi:10.1073/pnas.98.2.404] |
| [30] |
Brohée S, Helden JV. Evaluation of clustering algorithms for protein-protein interaction networks. BMC Bioinformatics, 2006, 7(1): No.488.
[doi:10.1186/1471-2105-7-488] |
| [31] |
Qin GM, Gao L. Spectral clustering for detecting protein complexes in protein-protein interaction (PPI) networks. Mathematical and Computer Modelling, 2010, 52(11): 2066–2074.
[doi:10.1016/j.mcm.2010.06.015] |
| [32] |
Li XL, Wu M, Kwoh CK, Ng SK. Computational approaches for detecting protein complexes from protein interaction networks:A survey. BMC Genomics, 2010, 11(Suppl 1): No.S3.
[doi:10.1186/1471-2164-11-S1-S3] |
| [3] |
刘冰玉, 王翠荣, 王聪, 王军伟, 王兴伟, 黄敏. 基于动态主题模型融合多维数据的微博社区发现算法. 软件学报, 2017, 28(2): 246–261.
http://www.jos.org.cn/1000-9825/5116.htm [doi:10.13328/j.cnki.jos.005116]
|
| [4] |
黄发良, 于戈, 张继连, 李超雄, 元昌安, 卢景丽. 基于社交关系的微博主题情感挖掘. 软件学报, 2017, 28(3): 694–707.
http://www.jos.org.cn/1000-9825/5157.htm [doi:10.13328/j.cnki.jos.005157]
|
| [23] |
王杰, 梁吉业, 郑文萍. 一种面向蛋白质复合体检测的图聚类方法. 计算机研究与发展, 2015, 52(8): 1784–1793.
[doi:10.7544/issn1000-1239.2015.20150180]
|
 2017, Vol. 28
2017, Vol. 28


