从1977年第一代DNA测序技术开始, 生物基因数据不断地增长.第二代测序技术大大降低了测序的成本, 并提高了测序的速度[1].而第三代测序技术在近些年又有了新的里程碑.它可以测序更长的序列, 并且具有更高的精度[2].随着基因数据的快速增长, 如何高效地存储、访问以及查询这些海量数据是目前的一个亟待解决的问题[3].
近些年的研究发现尽管这些基因数据量很大, 例如一个人类基因包括3 000 000 000个字符span>, 但是这些基因序列之间的相似度非常高, 甚至连人类和老鼠的基因都有99%的相似度.因此可以通过只存储一个基准串, 而保存其他序列与该基准序列之间的差异来减少存储代价[4].一个名为DNAzip的程序可以将一个3GB的人类基因压缩到4MB[5].进一步的研究表明, 可以在这些压缩的数据上进行直接的查询而无需对原序列进行解压缩[6-9].其中, 文献[9]中提出了基于q-gram倒排索引的基准序列索引方法以及基于二分查找树的差异索引方法来高效地支持精确和近似查询, 可以满足大多数常用的查询需求.本文在该方法的基础上, 提出了进一步的改进, 从而提高了该方法在大数据下的可伸缩性.本文的贡献主要包括以下3个方面.
首先, 本文对基准序列进一步进行了压缩, 从而减少了其空间开销, 使其可以支持更大规模的数据.
其次, 本文提出了高效的查询方法来支持在压缩后的基准序列和差异序列上的高效精确查询.与原有基于q-gram的索引方法相比, 该方法不需要预先对查询序列的长度加以了解, 可以高效地支持任意长度的序列查询.我们进一步对该方法进行了扩展, 以支持近似查询, 并通过共享计算的方法来降低冗余计算的开销, 提高查询效率.
最后, 原有方法只支持串行计算, 而目前许多高性能应用场景需要并行计算.本文对所提出索引方法进行了改进, 提出了并行索引方法, 并在此基础上提出了支持多机并行的查询方法, 从而提高了系统在并行环境下的查询效率.
1 相关工作数据压缩在计算机科学中有着悠久的历史, 本文中提到的高相似性序列压缩索引方法最早是由Wheeler在文献[4]中提到, 它利用一组高相似的序列集合中的一个序列作为基准, 将其他序列压缩为从该序列转换而需要的编辑操作.相比其他压缩方法, 该方法对于高相似性序列具有更高的压缩比, 对于人类基因数据, 该方法的压缩比可以达到1000:1[10, 11].基于该想法, 许多实用的算法被提了出来[5, 12-15].
基因数据上最常见的数据分析任务是序列的精确查询[16].经过压缩后的数据, 可以方便地保存和传输, 但对于精确查询, 传统的查询方法需要在线地对数据进行解压缩, 空间和时间开销巨大.为了提高数据的可用性, 提升系统查询性能, 一些方法提出了直接在压缩的数据上构建索引来实现精确的数据查询[3, 6-9, 17, 18].这些方法目前主要包括两大类.一类是基于整体压缩的方法来对数据进行索引, 进而支持快速查询.Mäkinen等人提出了基于压缩后缀数组的压缩索引方法来对数据进行快速查询[3].Ferrada以及Kreft等人提出了针对LZ77压缩结构的快速索引方法[6, 8].Kuruppu等人和Claude等人提出了针对Lempel-Ziv压缩结构的快速索引方法[7, 19].Huang等人提出利用BWT索引结构来对高相似性数据进行索引[20].这些方法通常需要对数据进行整体压缩索引, 即需要将所有序列进行连接之后压缩, 但是, 由于基因序列的长度一般很长, 构建索引的开销巨大.另一类是基于基准序列来构建签名索引的方法, 最为广泛采用的签名技术是q-gram, 它将一个序列中q长子序列作为签名进行索引, 通过签名匹配快速定位到所要查找的子序列.Claude等人提出了利用压缩的q-gram索引来支持基因序列上的高效查询[17].Schneeberger等人利用q-gram索引方法来进行短序列映射[18].Yang等人利用q-gram倒排索引方法来索引基准序列, 并利用二分查找树来索引差异, 从而实现高效的精确查询, 相对于其他方法, 具有更高的查询速度[9].该类方法由于只需要利用基准序列以及其他序列的差异, 因此构建索引的开销较小.但对查询的长度有一定的要求, 即查询序列长度必须不小于q.而且q的取值对查询效率有很大影响.
随着生物信息学的不断发展, 单纯支持精确查询已经无法满足应用的需求, 许多场景需要近似查询支持, 目前广泛接受的近似匹配方法被称为k近似匹配[21, 22], 即查询序列与匹配序列之间的编辑距离不超过k.文献[9, 18]通过对精确方法进行扩展, 来支持近似匹配.
文献[23, 24]研究了如何高效地计算序列之间的差异.许多基因库, 例如James Watson、炎黄等基因库都直接提供了差异序列.Wandelt等人提出利用多个序列作为基准序列的方法, 以支持在多个相似数据集合上的压缩存储和查询[25].
2 基本概念与问题定义我们用
令一个序列集合为
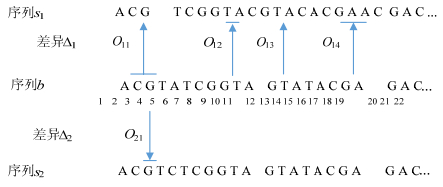
|
Fig. 1 An example of compressed genome sequence 图 1 一个压缩基因序列示例 |
下面我们给出本文研究的两个问题的形式化定义.
定义1.精确模式匹配问题:给定一个查询序列p, 找到所有S中满足的子序列, 其中, 1≤k≤n, 1≤i≤j≤|sk|.
定义2.近似模式匹配问题:给定一个查询序列p, 找到所有S中满足的子序列其中, 1≤k≤n, 1≤i≤j≤|sk|.
3 基于压缩的基准序列的查询方法 3.1 基于BWT的基准序列压缩方法我们首先研究如何对基准序列进行压缩.对基准序列压缩的方法有许多选择, 例如LZ77, LZ78, Lempel-Ziv以及BWT等, 其中, BWT(Burrows-Wheeler转换)方法是将压缩和查询结合得最好的一种方法.由于本文提出的方法既要对数据进行压缩又要兼顾高效的查询, 因此本文选择采用BWT作为基准序列的压缩方法.BWT本身不压缩数据, 而是将数据进行一个转换, 转换之后数据更容易被压缩[26, 27].压缩软件bzip2就是利用了BWT的方法来实现高压缩比.该方法对字符序列s进行转换的过程如下.首先它在s后添加一个新的字符$, 其中, $∉∑并且$的字典序小于任何∑中的字符.然后对这个新的序列计算后缀数组, 其中后缀数组中的第i位置上的元素是这个新的序列上按字典序排序的第i小的后缀的开始位置.而BWT转换之后的字符序列是|s|+1长的新序列t, 当SA[i]=1时, t[i]=$; 其他情况下, t[i]=s[SA[i]-1].BWT转换的一个具体的例子如图 2所示.对于一个字符序列ACTACGTACT来说, 进行BWT转换之后可以得到一个新的字符序列TTT$AAACCCG.不难发现, 这个字符序列中许多相同字符聚集在了一起, 相对于原序列来说可以更好地支持压缩.BWT可以高效地支持在任意位置上的逆转换, 用于整体和局部进行解压缩来恢复原序列, 关于基于BWT转换的压缩和解压缩算法的更多细节可参考文献[26, 28].
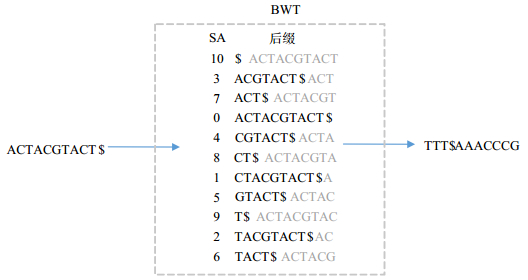
|
Fig. 2 An example of Burrows-Wheeler transform 图 2 一个BWT转换示例 |
我们对原序列进行BWT转换, 并将转换之后的数据进行游程编码压缩(run-length encoding, 简称RLE).它将连续相同的字符压缩为重复的次数和字符的形式.例如在图 2所示的例子中, TTT$AAACCCG将被压缩为3TA3CG.我们将压缩后的基准序列称为索引I.
3.2 基于压缩基准序列的精确查询方法本节我们首先给出一种基于压缩基准序列的基本精确查询方法, 然后对该方法进行改进以提高查询效率.
3.2.1 基本精确查询方法首先我们考虑如何只在基准序列上进行精确查询, 基于BWT压缩的数据具有一个很好的性质, 可以支持在压缩数据上的精确查询, 该方法被称为BWT反向查询.该方法基于一个观察:假设序列p在序列s上出现多次, 那么所有出现序列p的位置在后缀数组上是连续的一段区间.例如在图 2中, 假设查询序列p=ACT, 它出现在后缀数组中的区间[3, 4]上, 其中SA[3]=7, SA[4]=0.而BWT反向搜索方法就是从一个查询序列的最后一个字符开始, 反向遍历所有字符, 并且在遍历的过程中不断精炼匹配区间, 最终完成对匹配序列的定位.基于BWT的反向搜索算法如算法1所示.该算法首先将初始匹配区间设置为b中所有位置, 相当于在b上查询空序列, 见算法1第1行.然后它将对查询序列p的字符进行反向遍历, 在遍历过程中精炼匹配区间, 见算法1第2行~第3行.最后将p在b上所有匹配的位置添加到结果集中, 见算法1第4行~第5行.关于BWT反向查询算法的实现细节可参考文献[27].
算法1.基于BWT的反向搜索算法.
输入:索引I, 查询序列p;
输出:序列p在I中出现的所有位置P={i∈[1, |s|]|s[i, i+|p|-1]=p}.
1. [sp, ep]=[1, |b|+1]
2. FOR i←|p| TO 1
3. refine(sp, ep, p[i])
4. FOR i←sp TO ep
5. P.add(SA[i])
下面我们讨论如何在其他序列上进行查询.由于我们采用了压缩的方式存储序列, 因此对于其他序列来说, 只能查询到该序列与基准序列间的差异.接下来我们将分3种情况(插入、删除、替换)来对查询方法进行讨论.
首先考虑删除操作.由于删除操作并不会带来新的字符, 因此我们仍然可以在基准序列上进行搜索.当搜索过程中遇到一个删除操作时, 将在该位置跳过删除的字符.如图 1所示, 假设查询序列为p=CGT, 那么在反向查询该序列时, 首先读入字符串T, 它将在序列b上找到5处匹配位置.分别对应了{4, 6, 10, 13, 15}.而其中位置6的左侧为一个删除操作, 因此在该位置上跳过两个字符, 从位置3继续之前的查找.值得注意的是, 在序列b上搜索的过程只需要对该查询对应的匹配部分进行解压, 而无需全部解压.因此不会导致大量的内存开销.
接下来考虑插入操作.由于插入操作会带来新的字符, 因此有两种不同的情况.首先是当一个匹配序列结束于基准序列时, 该匹配仍然可以通过在基准序列上搜索来找到.同样考虑图 1中的例子, 假设查询序列仍然是p=CGT, 当查询反向搜索到第2个字符G时, 可以在b上找到3个匹配位置.分别对应了{3, 9, 12}, 而其中12位置的左侧有一个插入操作, 通过查看该插入操作可以发现, 该处插入了一个字符C, 匹配了p中当前字符.因此该位置是p的一个匹配.插入操作可能出现的另一种情况是, 当该匹配序列结束于插入操作中时, 该匹配将无法在基准序列上直接找到.例如, 考虑查询序列为p=AAC, 当反向搜索字符C时, 将只能找到位置{2, 7, 17, 22}, 而无法找到在O12和O14中出现的字符C.我们将在后面具体讨论如何能找到这些结束在插入操作中的匹配.
最后考虑替换操作.与插入操作相类似, 替换操作也会带来新的字符, 因此也需要分两种情况讨论.首先, 当一个匹配序列结束于基准序列时, 与插入序列一样, 我们可以直接在索引I上查找到.例如, 考虑查询序列为p=TCT, 对于最后一个字符T, 可以找到5个匹配位置, 分别对应了{4, 6, 10, 13, 15}, 其中位置6左侧对应了一个替换操作O21, 在这一情况下, 将在该替换操作中进行比对, 然后在该操作之前的位置4上继续搜索.与插入操作类似, 匹配序列结束的位置也可能出现在替换操作中.
下面我们将讨论如何找到结束在插入操作和替换操作中的匹配序列.为了找到这些位置, 我们将所有插入操作以及替换操作新引入的字符序列进行索引, 为了减少索引带来的空间开销, 我们仍然采用BWT结合游程编码的方式.首先将所有新引入的字符序列以字符$为分割进行连接, 连接之后再通过BWT对该序列进行转换, 并利用游程编码对其进行压缩, 从而得到插入和替换操作的压缩索引I’.然后通过这个压缩索引来进行搜索.例如考虑查询序列p=AAC, 当反向搜索读入字符C时, 可以在I’上找到该序列出现的位置分别为操作O12和O14, 然后读入前一个字符A, 对于操作O12来说, 该操作左侧为基准序列b上的位置11, 将在该位置上继续查询.而对于操作O14来说, 由于前一个字符仍然在O14中, 则直接在O14进行匹配.而当再读入前一个字符, 将继续在基准序列b上进行查询.同理, 结束在替换操作中的匹配序列也可以找到.
3.2.2 改进的精确查询方法在上一节, 我们给出了一种基本的基于压缩基准序列的精确查询方法, 下面我们将讨论如何进一步加速该查询.在基本的精确查询方法中, 每当反向读入一个字符时, 需要判断是否当前的匹配序列的左侧存在修改操作, 该操作需要检查所有当前匹配的序列, 时间代价很大.下面讨论如何改进该过程从而提高整体的查询效率.
该改进方法基于一个观察:只有很少一部分字符的左侧带有编辑操作.因此不需要对每个匹配位置进行线性扫描.基于该特点, 我们将这些差异序列的顺序进行重新修改, 首先将这些差异的结束位置增加1, 然后进行逆后缀数组顺序排序.考虑图 2中的示例, SA[1]=10, 那么10所对应的逆后缀数组顺序即为1, 将排在第1个位置.我们将逆后缀数组表示为SA-1.例如, SA-1[10]=1, SA-1[3]=2, 依次类推.这样当我们精炼匹配区间时, 可以通过二分查找排序之后的差异序列来直接获取哪些编辑操作在当前匹配区间内出现, 而不需要扫描所有修改位置, 从而节省了时间开销.
下面我们将给出具体的基于压缩基准序列的精确查询算法.该算法首先反向依次扫描查询序列p中的字符, 计算该查询序列在基准序列索引I上的匹配区间, 见算法2第2行.然后二分查找其前一个位置可能出现的编辑操作, 并将其保存到待验证列表, 见算法2第3行~第4行.接着在索引I’上进行反向搜索, 并将其对应的编辑操作保存到待验证列表, 见算法2第5行~第6行.然后将p的匹配位置保存到结果集, 见算法2第7行.最后对于所有待验证列表中的候选者进行验证, 并将通过验证的匹配位置添加到结果集中.当该操作出现在基准序列上时, 通过局部解压缩来验证, 当其出现在编辑操作中时, 则根据编辑操作内容进行验证, 见算法2第9行~第16行.
算法2.基于压缩基准序列的精确查询算法.
输入:基准序列索引I, 差异序列索引I’, 查询序列p;
输出:所有满足sk[i, j]=p的结果集A.
1. FOR i←|p| TO 1
2. 在基准序列索引I上反向搜索p[i], 并计算其匹配区间[sp, ep]
3. 二分查询[sp, ep]之间出现的编辑操作集合CD
4. 保存该编辑操作集合CD到待验证列表Cv
5. 在I’上反向搜索p[i], 并计算其匹配区间[sp’, ep’]
6. 保存[sp’, ep’]所对应的编辑操作集合
7.添加所有完整匹配p的序列到结果集A
8. FOR c∈Cv
9. While验证未结束
10. IF前一个字符出现在基准序列b上Then
11. 对其解压并验证
12. ELSE IF前一个字符出现在一个插入或替换操作中Then
13. 在该操作中进行字符验证
14. ELSE Then
15. 跳过删除操作
16. 添加新匹配的序列到结果集A
下面我们对该方法的时间复杂度进行分析, 该算法的查询过程主要分为两个步骤.第1步是对查询序列进行反向搜索, 并查询可能出现的编辑操作, 该过程时间复杂度为O(|p|xlognop), 其中, nop为编辑操作的总数.第2步是对出现编辑操作的候选序列进行验证, 该过程的时间复杂度为O(|p|x|Cv|), 其中, |p|为进行一次验证的最坏情况的复杂度.因此, 总的时间复杂度为O(|p|x(lognop+|Cv|)).相比较文献[9]中方法的时间复杂度为O(Nmin+Nminx lognop+Nminx|p|), 其中, Nmin表示采用了定长的q-gram的最少候选者数目, 而该数量Nmin≥Cv, 因此该方法的时间复杂度优于文献[9]中的方法.
3.3 基于压缩基准序列的近似查询方法本节我们将讨论如何高效地实现基于压缩基准序列的近似查询方法.该方法基于鸽巢原理, 即如果一个查询序列p与序列sk的某个子序列sk[i, j]的编辑距离不超过t, 那么将p分割为τ+1条分割的子序列, 至少会有一个分割子序列与序列sk[i, j]中的某个子序列精确匹配.基于该性质, 我们可以将一个近似序列的查询过程转换为多个精确序列的查询过程.然后对每个查找到的精确匹配序列再进行进一步的近似比对.例如, 考虑查询序列为p=ACGTATTCG, 假设编辑距离阈值为τ=2, 那么该查询序列将被分割为3个子序列.本文采用均匀分割的方式, 该查询序列将被分割为ACG, TAT和TCG.通过对这些子序列的查找, 可以找到一组匹配的序列.其中每个精确匹配序列都是近似匹配的一个候选, 需要通过进一步的验证来判断是否该子序列可以扩展出查询序列的一个近似匹配.
3.3.1 基本验证方法下面考虑一个基本的验证方法.假设查询序列p的一个子序列p[x, x+m]与序列sk中的一个子序列相匹配sk[y, y+m], 其中, m为这两个子序列的长度.子序列p[x, x+m]将p分为两段, 分别为p[1, x-1]和p[x+m+1, |p|].假设序列sk上存在这样一个基于sk[y, y+m]的子序列sk[i, j], 满足ed(sk[i, j], p)≤t, 其中, i≤y, j≥y+m.那么子序列sk[y, y+m]将sk[i, j]也分为两段, 分别为sk[i, y-1]和sk[y+m+1, j].并且它们一定满足ed(p[1, x-1], sk[i, y-1])+ed(p[x+m+1, |p|], sk[y+m+1, j])≤τ.
一个基本的近似验证过程如下所示.首先是对子序列sk[y, y+m]进行扩展, 然后对扩展之后的左右部分分别进行编辑距离验证.
首先考虑右扩展, 一个匹配子序列的右扩展是从该子序列的结束位置的下一个字符开始, 向右扫描字符.值得注意的是, 该子序列的结束位置可能会出现在基准序列b上, 也可能出现在序列sk的某个编辑操作(插入和替换)上.如果该位置出现在基准序列上, 则直接在基准序列上向右侧进行解压缩, 但是, 如果该序列出现在某个编辑操作上, 则需要先扫描该编辑操作上的剩余字符, 然后在基准序列上向右侧进行解压缩.如果再次遇到编辑操作, 则需扫描该编辑操作来进行扩展.扩展的长度最多为|p|-x-m+t, 因为在p的右侧一共有|p|-x-m个字符, 而两个子序列的长度差不能超过编辑距离t.如果到达了序列的边界, 则提前结束扩展.同理左扩展是从该子序列的开始位置的前一个位置开始, 向左扫描字符进行扩展.扩展的长度最多为x-1+t, 达到序列边界时, 则提前终止.
下面考虑验证过程, 在得到扩展序列之后, 可以分别在其左侧和右侧计算编辑距离, 考虑如图 3所示的例子中区域①中所对应的矩阵的最后一行上的值, 其中最小的值就是右侧的最佳匹配距离, 我们用vr来表示.同理, 左侧矩阵②中的最佳匹配距离用vl来表示, 那么整体的最小编辑距离为vl+vr, 如果该值小于编辑阈值t, 那么该子序列即为查询序列的一个近似匹配子序列.而其中左右两个矩阵中最佳编辑距离出现的位置为最佳近似匹配子序列的开始和结束位置.
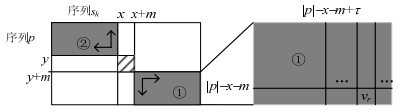
|
Fig. 3 Approximate verification method based on a pair of matching subsequences 图 3 基于一对匹配子序列的近似验证方法 |
3.3.2 优化策略
下面我们提出两个策略来对近似查询过程进行优化.首先, 多个精确序列的查找过程可以进一步优化.因为不同的精确匹配可能会有相同的后缀, 而相同的后缀在精确匹配过程中可以共享相同的计算.为了达到共享计算的目的, 我们利用这些序列共同构建一棵反向的查询树.图 4展示了对ACG, TAT和TCG构建反向查询树的结果.可以发现, ACG和TCG两个子序列共享了相同的后缀CG, 因此其查询过程可以进行合并.当基于该方法进行查询时, 不同精确查询子序列的相同后缀只需要查询1次.值得注意的是, 随着近似查询序列编辑距离阈值的增大, 分割子序列的数量也会增加, 因此共享相同后缀的概率也随之增加, 亦即会有更大的几率共享计算.

|
Fig. 4 An example of constructing a backward searching tree 图 4 构建反向查询树示例 |
其次, 多个序列的验证过程可以进一步优化.考虑一个出现在基准序列上的精确匹配序列, 该序列将对应于多个匹配的序列, 例如考虑图 1中的例子, 假设查询串ACG, 该查询串在序列b的位置1上对应了在两个序列s1和s2上的两处匹配.而对它们进行右侧扩展会找到相同的序列前缀TC.这些共享相同前缀的字符序列的编辑距离计算可以只做1次.通过编辑距离的共享计算可以减少冗余计算, 提高查询效率.值得注意的是, 重复计算并不只限制在同一个匹配位置, 例如在序列s1的操作O12位置处的子序列ACG的右侧序列同样带有字符前缀T.为了共享该部分计算, 我们采用树状结构对相同前缀上的编辑距离进行缓存.
4 基于分布式索引的并行查询方法第3节讨论了基于压缩的基准序列的索引和查询方法, 本节我们将讨论如何将该过程并行执行, 从而提高查询效率.首先, 我们讨论分布式索引方法, 然后在此基础上提出基于该索引的并行查询方法.
4.1 高效的分布式索引方法一种简单的针对压缩生物基因数据的分布式索引方法是在全局中共享一个基准序列, 其他分布式节点上保存了其他序列与该基准序列的差异, 当需要查看基准序列上的字符时, 需要通过远程通信的方式来进行访问.该方法在查询过程中由于需要大量的多机通信, 效率不高.图 5展示了基于全局共享的分布式索引结构.
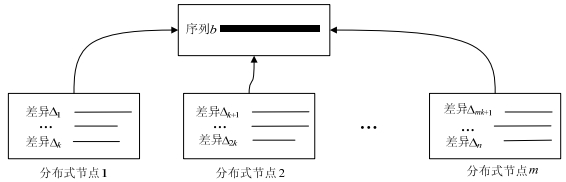
|
Fig. 5 Distributed index method with a global shared reference sequence 图 5 全局共享基准序列的分布式索引方法 |
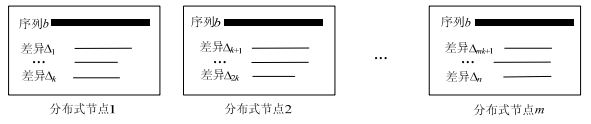
为了降低通信代价, 可以将基准序列进行多次备份, 分散到分布式环境中, 该方法相当于将一个大的系统拆分为多个平行的小系统, 其中每个小系统中都保存了一个基准序列, 这样就可以在每个基准序列上进行高效的查询, 而无需进行多机之间的通信.但是该方案存在一个问题, 亦即由于基准序列需要保存多个备份, 增加了系统的空间开销, 因此降低了可伸缩性.图 6展示了多拷贝基准序列的分布式索引结构.
|
Fig. 6 Distributed index method with multi-copy reference sequences 图 6 多拷贝基准序列的分布式索引方法 |
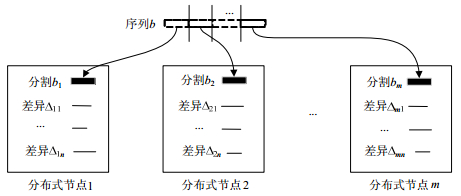
下面我们提出一种高效的分布式索引方法来实现在减少空间开销的同时保证高效的查询.本文采用的方法是将基准序列进行分割, 将分割后的序列分布到系统中的不同节点, 同时记录哪些分割序列对应了哪个节点.在添加其他序列时, 将出现在对应分割序列上的修改操作分配给对应的节点.
图 7展示了一个基于分割基准序列的分布式索引方法.
|
Fig. 7 Distributed index method with segmented reference sequence 图 7 分割基准序列的分布式索引方法 |
它首先将基准序列b进行等长分割, 并将分割之后的子序列分别保存在不同的分布式节点上, 再由每个分布式节点对其建立压缩索引.然后将对应分割子序列上的其他序列的差异序列分配给对应的节点, 再由各节点对其上的差异序列构建索引.当系统中添加了一个新的分布式节点时, 系统将对当前负载最高的节点中的分割基准子序列进一步分割, 并将分割后的基准子序列和对应其他序列上的差异序列分配给新增加的节点, 从而实现高效的扩展.该索引方法可以有效地减少空间开销, 但是查询过程中可能会存在匹配序列跨越两个甚至多个节点的情况, 直接在每个节点上进行查询可能会有漏解, 下面我们将讨论如何在保证高效查询的同时避免查询漏解.
4.2 基于分布式索引的并行查询方法首先考虑精确查询.由于我们已将基准序列和对应差异序列分配在不同节点上, 在查询的过程中可能会出现以下3种情况.以图 8为例, 情况1是匹配序列完整地出现在一个节点i中.这种情况下, 我们只需在该分布式节点上独立地进行查询即可.情况2是当一个匹配序列跨越了相邻的两个分布式节点时.在该示例中, 一个匹配序列跨越了节点i-1和节点i.在该情况下, 节点i上的查询只能找到部分匹配, 剩下的则需要在第i-1节点上进一步进行查询.第3种情况是对情况2的进一步扩展, 即当查询串很长时, 可能会跨越多个节点.在该示例中, 一个查询序列跨越了节点i、节点i+1和节点i+2, 在这种情况下, 我们将首先在i+2节点上搜索, 然后依次在节点i+1和节点i上进行查询.
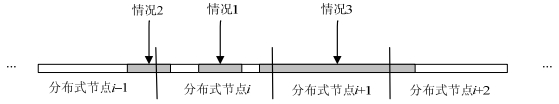
|
Fig. 8 Searching method based on distributed index with segmented reference sequence 图 8 基于分割基准序列的分布式索引的查询方法 |
下面我们将给出具体的并行查询算法, 该方法并行地在每个节点上执行以下操作.对于每个节点i, 它将首先在其上的索引上进行反向搜索, 找到一组查询序列p的整体或局部匹配序列作为候选集合, 见算法3第1行.如果该过程匹配了完整的查询序列p, 则将其保存在本地的结果集中, 否则说明该后缀跨越了当前节点i, 则将其保存在待验证列表中.然后统一发送该列表至节点i-1, 见算法3第7行.如果该节点为最后一个节点, 则发送结束信号给节点i-1并返回当前本地结果.否则, 就等待节点i+1发来的验证列表, 并对收到的列表进行验证, 并将仍无法验证的部分发送给节点i-1.当收到节点i的结束信号时, 该节点将发送结束信号给i-1节点, 并返回本地结果集, 见算法3第16行~第17行.
算法3.基于分布式索引的并行查询算法.
输入:分割后的索引IS, 查询序列p;
输出: A为p在S上的所有精确匹配子串.
在节点i上执行以下操作, 1≤i≤m
1.反向搜索得到候选集C
2. FOR候选者
3. IF c=p Then
4. 将c添加到本地结果集中
5. ELSE Then
6. 保存c到待验证列表
7.发送待验证列表到节点i-1
8. IF i=m Then
9. 发送结束信号到节点i-1
10. 返回本地结果集Ai
11 ELSE Then
12 WHILE未收到节点i+1结束信号
13. IF收到验证列表
14. 对其进行验证, 并将新的结果保存到结果集Ai中
15. 将仍未能验证的候选集发送到节点i-1
16.发送结束信号到节点i-1
17.返回本地结果集Ai
该过程可以进一步优化.可以预先通过对节点上的序列进行统计来得到节点上保存的最短序列的长度lmin.如果一个查询序列p的长度小于该长度, 那么在收到上个节点发送来的验证列表之后就可以提前结束计算过程并返回结果, 因为不会有匹配序列可以跨越两个以上的节点.进一步地, 如果一个序列p的长度满足|p|≤kxlmin, 那么我们可以在收到第k次验证列表后提前结束当前计算过程, 因为不会有匹配序列可以跨越k+1以上的节点.
近似查询与第3.3节的过程相类似, 首先将序列分割为多个精确查询的序列, 并通过上述算法查找其匹配位置, 然后在其左右两侧进行扩展验证, 与之前算法不同的是, 当该序列跨越了不同的节点时, 需要首先在右方向查询找到最佳的匹配位置, 然后发送该位置和对应最佳编辑距离给左侧节点来进一步验证, 对于跨越了多个节点的情况, 将重复该过程.最后将为满足编辑距离阈值的匹配返回其最佳匹配位置.采用并行查询方法的时间复杂度为O(|p|x(lognop+|Cv|)/m).
5 实验与分析 5.1 实验设置本文实验中的数据来自于两个公开发布的基因序列库:一个是James Watson基因库, 另一个是炎黄基因库.我们将它们保存为一个基准序列和其他序列与该基准序列的差异.由于任何两个基因序列的差异都在千分之一左右, 基准序列的选取对于压缩性能以及查询性能影响不大.本实验采用文献[9]中提到的UCSC基因库中的HG19作为基准序列.我们采用如下方法构建查询序列.首先在基准序列上随机抽取长度分别为40、200和2 000的3组子序列, 每组各100个, 然后为其添加1%~5%不等的编辑操作.
本文实验的硬件环境由20个节点构成, 使用一台Gigabit以太网交换机相互连接, 每个节点上配置一个Intel Core处理器2.93GHz, 4GB内存, 软件环境采用Linux操作系统和C++version4.7.我们主要与文献[9]中提到的基因压缩查询索引方法进行对比, 将该方法简称为GCSI, 将本文提出的高效并行的压缩查询索引方法简称为EPCSI.其中, 在GCSI方法中, gram的长度设为q=10, 查询方法采用C_Verify, 该方法为文献[9]中推荐的最佳方法.
5.2 实验结果(1) 索引空间开销对比
图 9对比了两种索引方法的空间开销.其中, 图 9(a)对比了在不同序列长度下两种索引的空间开销, 我们令查询序列个数都为50个, 由于采用改进的压缩方法, EPCSI的索引空间开销远小于GCSI, 特别是在序列长度达到109时, GCSI无法在内存中为其构建索引, 而EPCSI仍可以完成索引构建.图 9(b)对比了在不同序列数目情况下两种方法的索引大小.我们令序列长度为109.同样, 在不同序列大小的情况下EPCSI的索引空间开销也优于GCSI, 当序列数量达到50个时, 只有EPCSI可以完成索引的构建.在4GB内存的单处理机环境下, EPCSI能够最长处理2.6GB的序列.

|
Fig. 9 Comparison of index space cost 图 9 索引空间开销对比 |
(2) 精确查询性能对比
图 10对比了两种方法在查询序列长度分别为40、200和2 000时的精确查询的时间开销.在查询序列长度为40时, GCSI的性能要优于EPCSI, 但两者的差别不大.但是, 随着查询序列长度的增长以及数据集的增加, EPCSI的性能更好.出现该现象的主要原因如下:首先, 随着数据集的增加, EPCSI的索引空间占用较小, 搜索速度更快.其次, GCSI的gram长度预设为10, 随着查询长度的增长, 该方法仍需要检查长度为10的gram, 无法同时高效地支持短序列和长序列的查询.同样, 在序列长度达到109时, 由于无法构建GCSI的索引, 在该长度下我们只给出进行了EPCSI的查询时间.
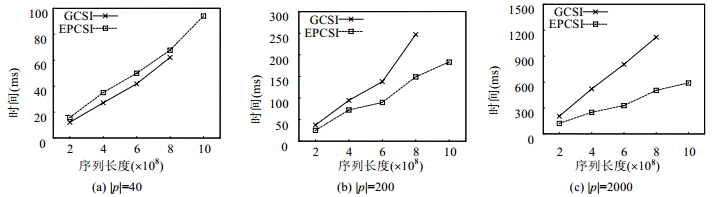
|
Fig. 10 Performance of exact query search 图 10 精确查询性能 |
(3) 近似查询性能对比
图 11对比了两种方法在查询序列长度分别为40、200和2 000时的近似查询的时间开销.我们令序列的长度为6x108.随着编辑距离的增加, 两者查询时间都有所增加, 它们的趋势基本一致.在长序列上, EPCSI的效率更好.该现象的主要原因如下:首先, 两种方法都采用了鸽巢原理来对近似序列进行划分.其次, 与精确查询相类似, EPCSI的索引空间更小, 查询的效率更高, 也更适合长序列查询.并且, EPCSI在查询过程中利用了共享后缀的方法来提高查询效率.虽然EPCSI需要在局部进行解压, 在验证时会有额外开销, 但在大多数情况下仍优于GCSI方法.当查询序列长度为40、编辑距离为4时, 每个分割子序列长度为8, 小于预设的gram长度10.在这种情况下, GCSI无法执行查询.而EPCSI没有查询长度的限制, 可以正常完成查询.
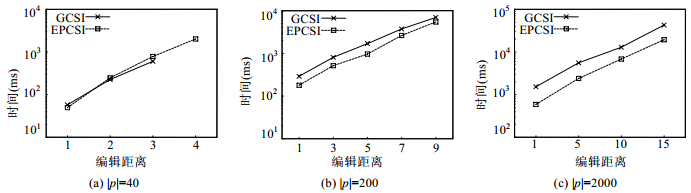
|
Fig. 11 Performance of approximate query search 图 11 近似查询性能 |
(4) 并行查询方法性能
图 12展示了并行查询方法的性能.其中, 图 12(a)比较了3种不同的并行索引方法的空间开销.随着节点数目的增加, 3种方法的空间开销都有所下降, 而其中基于分割基准序列的索引方法的空间开销最小.图 12(b)和图 12(c)分别给出了在基于分割的索引方法上进行精确和近似查询的时间开销.我们分别测试了长度分别为40、200和2 000的3组查询序列.对于近似查询, 令各组查询序列的编辑距离分别为2、5和10.可以看到, 随着节点数目的增加, 查询的时间开销有所降低.

|
Fig. 12 Performance of parallel method 图 12 并行方法的性能 |
(5) 可伸缩性
图 13展示了该方法在不同序列个数下的可伸缩性.我们在20个节点上对序列长度为3x109的数据集进行了可伸缩性实验.同样, 我们比较了查询长度分别为40、200和2 000的3组查询序列.可以看到, 随着序列个数的增加, 查询开销并没有线性地增加.该现象进一步说明了基因序列的特殊性, 即两个序列之间的差异很小, 大多数的查询可以利用共享计算来提高查询效率.
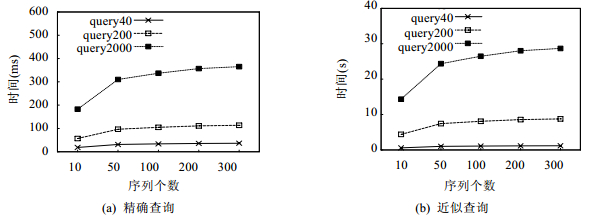
|
Fig. 13 Scalability of the method 图 13 方法的可伸缩性 |
6 总结
本文研究了在压缩的生物基因数据上高效并行的查询方法.本文首先对现有的基于基准序列的压缩索引方法进行了改进, 对基准序列进行了压缩, 进一步减少了空间开销, 从而支持更大规模的数据.然后在压缩的数据之上提出了高效的精确查询方法, 该方法不需要对查询序列长度有任何限制, 可以高效地支持任意长度序列的精确查询.在此基础上, 本文对所提出的精确查询方法进一步加以扩展, 以支持带有编辑距离的近似查询.然后, 本文将现有的串行查询方法进行扩展, 提出了空间高效的分布式的索引结构, 并且在该索引结构之上提出了并行查询方法.通过在真实的数据集进行实验证实本文提出的方法具有很高的查询效率以及很好的可伸缩性, 可以支持更大规模的数据查询.
| [1] | Mardis ER. The impact of next-generation sequencing technology on genetics. Trends in Genetics, 2008, 24 (3) :133–141. [doi:10.1016/j.tig.2007.12.007] |
| [2] | Rusk N. Cheap third-generation sequencing. Nature Methods, 2009, 6 (4) :244. [doi:10.1038/nmeth0409-244a] |
| [3] | Mäkinen V, Navarro G, Sirén J, Välimäki N. Storage and retrieval of highly repetitive sequence collections. Journal of Computational Biology, 2010, 17 (3) :281–308. [doi:10.1089/cmb.2009.0169] |
| [4] | Wheeler DA, Srinivasan M, Egholm M, Shen Y, Chen L, McGuire A, He W, Chen YJ, Makhijani V, Roth GT, Gomes X. The complete genome of an individual by massively parallel DNA sequencing. Nature, 2008, 452 (7189) :872–876. [doi:10.1038/nature06884] |
| [5] | Christley S, Lu Y, Li C, Xie X. Human genomes as email attachments. Bioinformatics, 2009, 25 (2) :274–275. [doi:10.1093/bioinformatics/btn582] |
| [6] | Ferrada H, Gagie T, Hirvola T, Puglisi SJ. Hybrid indexes for repetitive datasets. Philosophical Trans. of the Royal Society of London A:Mathematical, Physical and Engineering Sciences, 2014, 372 (2016) :130–137. [doi:10.1098/rsta.2013.0137] |
| [7] | Kuruppu S, Puglisi SJ, Zobel J. Relative Lempel-Ziv compression of genomes for large-scale storage and retrieval. In:String Processing and Information Retrieval. Berlin, Heidelberg:Springer-Verlag, 2010 :201–206. [doi:10.1007/978-3-642-16321-0_20] |
| [8] | Kreft S, Navarro G. Self-Indexing based on LZ77. Lecture Notes in Computer Science, 2011, 6661 (1) :41–54. [doi:10.1007/978-3-642-21458-5_6] |
| [9] | Yang XC, Wang B, Li C, Wang JY. Efficient direct search on compressed genomic data. In:Proc. of the Int'l Conf. on Data Engineering. IEEE, 2013 :961–972. [doi:10.1109/ICDE.2013.6544889] |
| [10] | Deorowicz S, Grabowski S. Robust relative compression of genomes with random access. Bioinformatics, 2011, 27 (21) :2979–2986. [doi:10.1093/bioinformatics/btr505] |
| [11] | Wandelt S, Leser U. FRESCO:Referential compression of highly-similar sequences. IEEE/ACM Trans. on Computational Biology & Bioinformatics, 2013, 10 (5) :1275–1288. [doi:10.1109/TCBB.2013.122] |
| [12] | Brandon MC, Wallace DC, Baldi P. Data structures and compression algorithms for genomic sequence data. Bioinformatics, 2009, 25 (14) :1731–1738. [doi:10.1093/bioinformatics/btp319] |
| [13] | Daily K, Rigor P, Christley S. Data structures and compression algorithms for high-throughput sequencing technologies. BMC Bioinformatics, 2010, 11 (19) :514–524. [doi:10.1186/1471-2105-11-514] |
| [14] | Rasko L, Hideaki S, Martin S. The sequence read archive. Nucleic Acids Research, 2011, 39 (2) :19–21. [doi:10.1093/nar/gkq1019] |
| [15] | Pinho AJ, Diogo P, Garcia SP. GReEn:A tool for efficient compression of genome resequencing data. Nucleic Acids Research, 2011, 40 (4) :27–53. [doi:10.1093/nar/gkr1124] |
| [16] | Zhu YY, Xiong Y. DNA sequence data mining technique. Ruan Jian Xue Bao/Journal of Software, 2007, 18(11):2766-2781(in Chinese with English abstract). http://www.jos.org.cn/1000-9825/18/2766.htm[doi:10.1360/jos182766] |
| [17] | Claude F, Farina A, Martínez-Prieto MA, Navarro G. Compressed q-gram indexing for highly repetitive biological sequences. In:Proc. of the BioInformatics and BioEngineering. IEEE, 2010 :86–91. [doi:10.1109/BIBE.2010.22] |
| [18] | Schneeberger K, Hagmann J, Ossowski S, Warthmann N, Gesing S, Kohlbacher O, Weigel D. Simultaneous alignment of short reads against multiple genomes. Genome Biology, 2009, 10 (9) :98–119. [doi:10.1186/gb-2009-10-9-r98] |
| [19] | Claude F, Fariña A, Martínez-Prieto MA, Navarro G. Indexes for highly repetitive document collections. In:Proc. of the 20th ACM Int'l Conf. on Information and Knowledge Management. ACM, 2011 :463–468. [doi:10.1145/2063576.2063646] |
| [20] | Huang S, Lam TW, Sung WK, Tam SL, Yiu SM. Indexing similar DNA sequences. In:Algorithmic Aspects in Information and Management. Berlin, Heidelberg:Springer-Verlag, 2010 :180–190. [doi:10.1007/978-3-642-14355-7_19] |
| [21] | Navarro G. A guided tour to approximate string matching. ACM Computing Surveys, 2001, 33 (1) :31–88. [doi:10.1145/375360.375365] |
| [22] | Lin XM, Wang W. Set and string similarity queries:A survey. Chinese Journal of Computers, 2011, 34(10):1853-1862(in Chinese with English abstract).[doi:10.3724/SP.J.1016.2011.01853] |
| [23] | Danek A, Deorowicz S, Grabowski S. Indexing large genome collections on a PC. Eprint Arxiv, 2014, 9 (10) :52–59. |
| [24] | Ferrada H, Gagie T, Hirvola T, Puglisi SJ. AliBI:An alignment-based index for genomic datasets. ArXiv, 2013, 7 (4) :32–39. |
| [25] | Wandelt S, Leser U. MRCSI:Compressing and searching string collections with multiple references. Proc. of the VLDB Endowment, 2015, 8 (5) :461–472. [doi:10.14778/2735479.2735480] |
| [26] | Burrows M. A block-sorting lossless data compression algorithm. Digital SRC Research Report, 1994, 57 (4) :425–449. |
| [27] | Navarro G, Mäkinen V. Compressed full-text indexes. ACM Computing Surveys, 2007, 39 (1) :2–53. [doi:10.1145/1216370.1216372] |
| [28] | Lam TW, Li R, Tam A, Wong S, Wu E, Yiu SM. High throughput short read alignment via bi-directional BWT. In:Proc. of the 2009 IEEE Int'l Conf. on Bioinformatics and Biomedicine. IEEE Computer Society, 2009 :31–36. [doi:10.1109/BIBM.2009.42] |
| [16] | 朱扬勇, 熊赟.DNA序列数据挖掘技术.软件学报, 2007, 18(11):2766-2781. http://www.jos.org.cn/1000-9825/18/2766.htm[doi:10.1360/jos182766] |
| [22] | 林学民, 王炜.集合和字符串的相似度查询.计算机学报, 2011, 34(10):1853-1862.[doi:10.3724/SP.J.1016.2011.01853] |
 2016, Vol. 27
2016, Vol. 27


