微阵列技术能够一次测定成千上万表达基因,为癌症等疾病诊断研究提供了全新和系统的手段,在医学基础和临床应用领域受到关注[1].然而DNA微阵列数据通常样本数较少,基因数成千上万[2, 3, 4],构成高维稀疏空间.高维稀疏特点,给基因数据集分类分析和疾病识别带来巨大挑战[5].Guyon等人研究者指出:通过微阵列技术得到的基因数据含有大量与特定疾病不相干或冗余的基因[2,5,6].冗余和无关基因的存在,使得对基因数据的分类准确率大大降低且很费时,并易于陷入维数灾难[1,2].选择具有高区别能力的基因不仅提高疾病分类识别和预测的准确率[1, 2, 3, 4, 5, 6, 7, 8],降低疾病诊断时间,减少临床诊断费用,并可促进相应药物研发[3,4].因此,特征选择成为分析高维稀疏基因数据集的一个首要且极具挑战性的问题.
特征选择依据是否独立于相应学习算法,分为Filter方法和Wrapper方法:
Filter方法与学习算法无关,直接利用所有训练数据的统计性能评估特征,进行有效区分特征选择,速度较快,但特征评估与学习算法的性能偏差较大;
Wrapper方法利用学习算法的分类准确率评估特征子集,偏差小,准确率较高,且选择的特征子集规模较小,非常有利于关键区分特征的选择,但计算量大,泛化能力较差,不适合大数据集.
因此,集Filter方法的快速与Wrapper方法的准确于一体的混合特征选择研究得到重视[1,2,5].
本文采用Filter和Wrapper相结合的方法进行有效区分基因选择,以期集成两种方法的优点,解决有效区分基因子集选择难题:
文中先分别采用Pearson相关系数[2,5]和Wilcoxon秩和检验[1]为Filter方法进行基因预选择,过滤掉对实现正确分类贡献较小的基因;然后,对剩余基因采用Wrapper方法,进行有效区分基因选择;
在Wrapper方法中:通过聚类使相关性较强的基因聚集在同一类簇,得到冗余度比较高的基因集合;训练SVM分类模型计算每个基因的权重[5,7,8],从每个类簇中选择权重最高的基因,或者采用轮盘赌思想从每个类簇中选择得票数最高的基因作为该类簇的代表基因,得到类簇数个基因构成的基因子集;再采用SVM分类器的分类性能评估选择的基因子集.
3个经典基因数据集上的详尽实验,以及不同规模预选择基因子集规模对区分基因子集的影响实验,充分验证了本文算法的有效性.
1 算法思想、实验设计与理论分析图 1描述了本文提出的有效区分基因子集选择算法的思想流程图.从图 1可见,本文算法包括5个步骤:
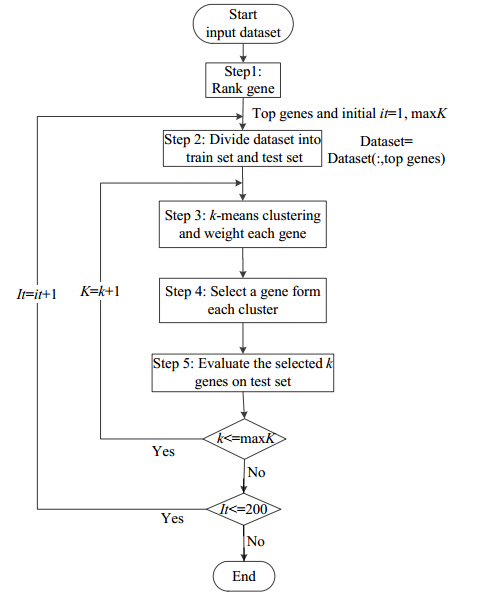 | Fig. 1 Flow chart of our gene selection algorithms图 1 本文算法思想流程图 |
第1步,在整个数据集上运用Filter方法,选择对分类贡献较大的基因构成预选择基因子集;
第2步,将数据集划分为训练集和测试集;
第3步,在训练集上,对基因进行K-means聚类,并训练SVM分类器,计算各基因的权重;
第4步,从每个类簇选取最能代表本类簇的基因,得到含有K个基因的区分基因子集;
第5步,训练SVM分类器,以其分类性能评价选择的区分基因子集的质量.
1.1 基因排序方法常用的基因排序方法有Relief-F、信息增益、c2检验等[2,9,10].Relief-F对噪声不敏感,对特征间的相互作用具有鲁棒性、不需要先验假设等优点,但其在小样本数据集上效果不佳.信息增益、c2检验适用于离散型数据,对连续型基因数据须先进行离散化处理,此操作会造成信息丢失.基因选择中广泛采用的是t统计量及其变形方法[9,11],t统计量方法的依据是t检验.
本文选用t检验的代表方法——Pearson相关系数以及不需要对原始数据集做任何假设的非参数统计方法——Wilcoxon秩和检验[11]来度量基因的类间区分能力大小,对基因进行排序,保留前10%的重要基因,剔除噪声和具有较小分类信息的基因.
1.1.1 Pearson相关系数Pearson相关系数是度量变量之间相关性的常用准则,且常用在微阵列数据分析[11,12]中,其定义见公式(1):
 (1)
(1)
其中,m是样本个数,R(i)度量第i个特征与类标的相关性,xk,i是第k个样本的第i个特征的特征值,![]() 是第i个特征的平均值,
是第i个特征的平均值,![]() 分别代表第k个样本的类标值和整个样本的类标均值.
分别代表第k个样本的类标值和整个样本的类标均值.
由公式(1)可知,R(i)的变化范围在-1和1之间:当R(i)=1时,第i个特征(基因)与类标正线性相关;当R(i)=-1时,第i个特征与类标负线性相关.随着第i个特征与类标相关程度的不同,|R(i)|的值在0~1之间变化,|R(i)|的值越大,表示第i个特征对于分类的贡献越大,越重要.因此,基因i的|R(i)|值越大,说明第i个基因越能更好地应用于分类.当第i个基因的|R(i)|=1时,表明第i个基因能够实现完全正确的分类.
1.1.2 Wilcoxon秩和检验Wilcoxon秩和检验是一种非参数统计方法,综合了t_test和阈值误判方法TNOM(threshold number of misclassification)的良好特性[1],克服了t_test对于噪音敏感和TNOM会丢失数据中信息的缺点.其计算公式见公式(2):
![]() (2)
(2)
其中,I(×)是判别函数,若![]() 成立,则I(×)取值为1;否则取0值.
成立,则I(×)取值为1;否则取0值.![]() 表示第j个样本中第g个基因(特征)的表达值(特征值),N0,N1分别代表两个类别中的样本数量.排除基因在所有样本的表现形式都相同的情况,由公式(2)可知:当基因g的Wilcoxon统计量s(g)值接近于0或N0xN1时,基因g具有非常好的分类性能.因此,基因g的类间区分能力大小定义见公式(3),q(g)值越大,基因g的分类性能越强,越能更好地用于分类.
表示第j个样本中第g个基因(特征)的表达值(特征值),N0,N1分别代表两个类别中的样本数量.排除基因在所有样本的表现形式都相同的情况,由公式(2)可知:当基因g的Wilcoxon统计量s(g)值接近于0或N0xN1时,基因g具有非常好的分类性能.因此,基因g的类间区分能力大小定义见公式(3),q(g)值越大,基因g的分类性能越强,越能更好地用于分类.
q(g)=max(s(g),N0xN1-s(g)) (3)
1.2 数据集划分方法基因数据集的特点是维数很高,样本数较少,构成高维稀疏空间.有效区分基因选择,是这类数据集分类分析的首要任务和挑战.数据集的不同划分会影响基因选择的结果,为了尽可能消除由于数据集划分不同而产生的基因子集不稳定问题,尽可能客观地反映算法本身的性能,本文采用bootstrap方法[10]得到训练集和测试集;如果数据集本身已经划分为测试集和训练集,则采用已有的划分.
1.3 基因聚类与基因权重计算通过第1.1节的Filter方法过滤掉一部分不重要基因后,预选择基因子集中的基因具有较高的分类性能,但此时,基因之间的冗余度比较大[2, 6, 13, 14, 15].剔除冗余基因的代表方法包括:基于最小冗余最大相关原则MRMR (minimum redundancy-maximum relevance),递归增加相关度高且冗余度低的基因[6];分析基因之间的相关性,剔除冗余基因的FCBF(fast correlation-based Filter)方法[14];采用遗传算法避免冗余基因影响[1]等.近年来,采用特征聚类剔除冗余特征的研究得到关注.Wang等人[9]利用层次聚类,从每个类簇中选择一个到该簇其他基因距离最小的基因作为被选择基因.Song等人[13]利用特征间的相关性作权,构造带权无向完全图,采用Prim算法构造最小生成树,然后利用特征相关性[14]剪去部分边,得到相关性很高的树(簇),选择每棵树中与类标相关性最大的特征构成特征子集.Loscalzo等人[15]提出了CGS(consensus group stable feature selection)算法,对整个数据集采用bootstrap方法构造t个训练集,每个训练集得到若干个稠密特征组;然后,由稠密特征组构造出L个一致组,选出与类标相关性比较高的K个一致组;最后,从每组中选出相关性最高的特征.然而,现有这些方法要么时间或空间需求大,要么具有错误累积缺陷等.本文采用基于划分的经典聚类算法K-means对基因进行聚类,选择每个类簇的代表基因构成有效区分基因子集,以避免层次聚类的错误累积缺陷,又克服其他算法的时间或空间需求大的问题.
1.3.1 K-means聚类算法K-means算法是MacQueen于1967年提出的基于划分思想的聚类算法[10,16],其聚类结果使相似度较高的样本聚集到同一类簇,而类簇间的样本相似度较低[10].K-means算法以其实现简单和线性时间复杂度优势在科学研究和工业应用等领域被广泛采用,并被用于大数据分析[17].
本文利用K-means对预选择基因子集中基因进行聚类,将相似的基因聚集到同一类簇,使得冗余基因集中在同一类簇,从每个类簇选择一个代表基因代表该类簇,从而剔除冗余基因,完成有效区分基因子集选择.
1.3.2 基因权重计算方法本文采用支持向量机(support vector machine,简称SVM[18,19,20]来计算基因权重.SVM提供了最小化分类错误率和最大化泛化能力的理论保障,基于SVM的特征选择,特别是基于SVM的基因选择备受关注和青睐[5,7,8].经典的基因选择算法是Guyon等人提出的SVM-RFE[7],我们针对SVM-RFE的缺陷,提出基于顺序前向选择思想的可应用于多类分类问题的基因选择方法SVM-SFS[5],并提出采用分类超平面系数绝对值的基因权重计算方法.本文的基因权重计算方法同SVM-SFS的基因权重计算方法,用于实验比较的SVM-RFE的基因权重计算方法采用原始的SVM-RFE算法中的基因权重计算方法.
需要说明的是,利用SVM分类模型计算基因权重是为了选择各类簇的代表性基因.因此在实验设计中,我们分别训练了两类SVM分类模型计算各基因的权重:一是训练一个包含全部预选择基因的SVM分类器,按照上述方法计算基因权重;二是对每个基因簇,训练一个SVM分类器,每个分类器的训练样本只包含相应簇的基因,根据上述方法计算每个基因的权重.
1.4 有效区分基因选择利用K-means聚类得到K个基因簇,各基因簇内部基因之间高度相关,而类簇之间的基因相关度较低,造成簇内基因冗余度很高.从每个类簇中选择一个代表基因,可以保证选择到的K个基因之间的冗余度很低.为选择各基因簇的代表基因,本文采用权重策略和轮盘赌策略选择各类簇的代表基因.
1.4.1 权重策略基因的权重表达了基因的类间区分能力大小,代表了基因对于分类的贡献,因此从每个基因簇中选择一个权重最大的基因作为该簇代表基因,各类簇的代表基因构成有效区分基因子集.该策略默认单个分类能力强的基因组合后依然具有较强的分类能力,然而分类能力稍弱的基因组合后的分类性能有可能更优[2].通常情况下,单个分类能力强的基因组合在一起往往能取得较好的分类效果.因此,本文从每个类簇选择具有最好分类能力的基因构成基因子集能实现有效区分基因子集的选择.
1.4.2 轮盘赌策略与权重策略不同,轮盘赌策略在保障高权重基因具有较高被选择概率的同时,也使具有次高权重的基因也有可能被选择,克服了权重选择策略在个别情况下的缺憾.本文的轮盘赌策略对每个基因簇根据轮盘赌算法选择1个基因,得到K个基因构成的基因子集;然后,根据被选择基因子集的分类性能更新其中基因的权重.重复该过程L次,并记录每次选中的基因.L次重复结束后,选择每个类簇中得票数(被选中次数)最多的基因为该类簇的代表基因.具体方法如下:
I. 初始化分类准确率Acc,每个基因的初始权重wi由SVM学习机得到,对训练集进行划分,保留9/10样本为训练子集sub_train,剩下的1/10为验证子集sub_test;
II. 用轮盘赌算法从每个类簇中选出一个基因,得到包含K个基因的基因子集;
III. 由只包含被选择基因的sub_train训练SVM,在sub_test上检验当前被选基因子集的分类性能,记分类正确率为AccNew,根据公式(4)更新被选择基因的权重,根据公式(5)更新Acc;
IV. 重复步骤II和步骤III共L次,并保存每次选中的基因;
V. 根据保存的基因,选择每个类簇中得票数最多的基因作为本类簇的代表基因.各类簇的代表基因构成规模为K的有效区分基因子集.
![]() (4)
(4)
Acc=max(AccNew,Acc) (5)
1.5 基因子集质量评估K-means算法对基因进行聚类的结果不仅依赖于初始聚类中心,而且与数据集的不同划分以及样本的先后顺序有关[21].另外,对不同划分的训练集,SVM学习机得到的基因权值也可能不同.这使得对于确定的K值,最终选择的有效区分基因子集可能不同.因此,对确定的基因子集规模K,我们重复运行算法200次,根据统计性能评价算法质量.算法每次重复运行的分类正确率计算随数据集的不同划分而不同:若数据集划分采用bootstrap方法,则用公式(6)计算当次重复的分类准确率,其中,M是当次的SVM分类模型.
Acc=0.632xAcc(M)test_set+0.368xAcc(M)train_test (6)
分类器性能评估是一个非常复杂的问题,目前还没有关于分类器性能评价的客观和全面的理论研究[22],通常采用的分类器性能评价方法是对实验结果进行的比较和判断.为了说明本文算法的性能,我们将实验结果与采用随机策略选择代表基因的基因选择算法Random、经典基因选择算法SVM-RFE以及我们前期研究提出的基因选择算法SVM-SFS在相同实验环境下的结果进行比较.
1.6 算法分析本文算法依据对预选择基因进行聚类后,选择代表基因的不同策略分为两大类,简记为Weight和Roulette Wheel,分别表示选择代表基因使用权重策略和轮盘赌策略.另外,依据计算基因权重时训练SVM模型的不同策略,将本文依据权重选择类簇代表基因的方法分为Weight和WAC Weight(weighted after clustering),将采用轮盘赌策略选择各类簇代表基因的方法分为Roulette Wheel和WAC Roulette Wheel,分别表示计算基因权重时,是训练一个包含全部预选择基因的SVM分类模型,还是训练K个只含有当前簇基因的SVM分类模型两种情况.由此得到4种混合基因选择算法Weight,WAC Weight,Roulette Wheel和WAC Roulette Wheel.
1.6.1 时间复杂度分析假设原始样本特征数为d、样本数为n,通过Filter算法过滤后的特征数为m,K-means算法的平均迭代次数为t.对基因数据集通常有关系d>>m>>nfk.在样本数为n、特征数为d的数据集,建立SVM模型的最坏时间复
杂度为O(n2xd).特征权重排序的最好时间复杂度为O(dxlog2d).因此SVM-RFE的时间复杂度为O(d2xlog2d)[23],而SVM-SFS的时间复杂度为O(dxlog2d).
本文算法的时间消耗主要来自Filter步的基因预选择,以及Wrapper步的基因聚类与代表基因选择.各步的详细时间复杂度分析如下:
· Filter步预选择基因的时间复杂度.
该步的时间复杂度来自计算基因类间区分能力的相关性分析,以及对基因依据区分能力进行的排序两部分,因此,时间复杂度为O(nxd+dxlog2d);· Warpper步基因聚类的时间复杂度.
该步采用适用于大数据聚类的K-means算法对预选择基因进行聚类.对预选择的m个基因进行K-means聚类的时间复杂度为O(tkmn)[10,21].其中,m为Filter步预选择的基因数,即待聚类的基因数;t为K-means的迭代次数;n为数据集样本数;· Wrapper步代表基因选择的时间复杂度.
该步首先计算预选择的各个基因的权重,然后采用权重策略或轮盘赌策略选择各类簇的代表基因.计算预选择的m个基因的权重通过训练SVM获得,若训练一个包含全部预选择基因的SVM分类模型,则计算预选择的m个基因的权重的时间复杂度是O(n2xm);若每个类簇训练一个SVM分类模型,则在假设每个类簇包含的基因数相等的情况下,计算预选择的m个基因的权重的时间复杂度为![]() .因此,采用K-means对预选择基因进行聚类后,为选择各类簇的代表基因的计算每个预选择基因的权重的时间复杂度为O(n2xm).
.因此,采用K-means对预选择基因进行聚类后,为选择各类簇的代表基因的计算每个预选择基因的权重的时间复杂度为O(n2xm).
选择各类簇代表基因的时间复杂度和采用的具体基因选择策略有关.权重策略选择每个类簇权值最大的基因,其时间复杂度为O(m/k)(假设各类簇的大小相同),则选择k个类簇的代表基因的时间复杂度为
![]() .
.
轮盘赌策略选择各类簇代表基因的时间复杂度主要由实现轮盘赌策略时对每个类簇进行的SVM模型训练时间决定,其值不超过O(kn2),则重复进行L次的总时间复杂度不超过O(Lkn2).
以上时间复杂度分析揭示,本文提出的混合基因选择算法Weight,WAC Weight的时间复杂度为O(nd+ dlog2d+tkmn+n2m+m).因为d>>m>>n,由渐进时间复杂度理论[24]得知,Weight,WAC Weight的时间复杂度O(nd+ dlog2d+tkmn+n2m+m)与O(nd+tkmn)同阶,因此,本文基于权重策略选择各类簇代表基因的混合基因选择算法Weight,WAC Weight的时间复杂度为O(nd+tkmn);类似地分析可得,本文基于轮盘赌策略选择各类簇代表基因的混合基因选择算法Roulette Wheel和WAC Roulette Wheel的时间复杂度为O(nd+dlog2d+tkmn+n2m+Lkn2);O(nd+tkmn).因此,本文提出的基于统计相关性与K-means的混合基因选择算法的时间复杂度是O(nd+tkmn).由d>>m>>nfk可知,本文算法的时间复杂度远小于SVM-RFE的时间复杂度O(d2xlog2d).后面的第2.2.5节各算法运行效率的实验比较,验证了这里关于算法时间复杂度的理论分析.
1.6.2 区分基因子集质量分析本文算法通过对基因进行K-means聚类,将相关性较强的基因聚集到同一类簇,相关性较弱或不相关的基因分布在不同类簇,使得每个类簇中的基因有较大的冗余性,而不同类簇间的基因冗余性较小;然后,从每个类簇中选择一个和类标相关性最强的基因构成区分基因子集,保证了选择到的区分基因子集为理想的基因子集,即:基因子集中的基因和类标高度相关,但基因之间高度不相关.
由上面关于算法时间复杂度的分析可知:当预选择的剩余基因确定时,算法的时间消耗主要来源于K-means的聚类时间,而K-means与其他聚类算法相比具有线性的时间复杂度,是一种高效的聚类算法,可被用于大数据聚类[17,21].
因此,综合以上算法分析可见:在时间允许范围内,本文算法更能找到很好的区分基因子集.后面的实验结果也验证了这里的分析.
2 实验结果与分析实验使用的3个经典基因数据集包括基因选择研究常用的数据集Leukemia[25]以及普林斯顿大学基因表达工程的2个基因数据集Colon[26]和Carcinoma[27].各基因数据集的信息描述见表 1.
| Table 1 Description of gene datasets 表 1 数据集信息描述 |
表 1的白血病数据集Leukemia是ALL/AML Leukemia,其训练集和测试集已经划分好,实验采用现有的划分.结肠癌数据集Colon[26]和恶性肿瘤Carcinoma数据集[27]采用bootstrap划分方法得到训练集和测试集.
数据预处理是特征选择的首要步骤,包括处理缺失数据和对数据进行标准化.本文对缺失数据采用均值代替,并采用公式(7)的最大最小方法对数据进行标准化.经过数据标准化,不仅消除不同量纲对实验结果的影响,而且降低算法运行时间开销:
![]() (7)
(7)
其中,gi,j表示编号为i的基因在第j个样本上的表达值,max(gi),min(gi)分别表示第i个基因的最大、最小表达值.
2.2 实验结果与分析实验使用林智仁教授等人开发的SVM工具箱Libsvm[28],并采用线性核函数,惩罚因子C取固定值20.实验代码使用Matlab R2012a实现,实验环境为Win7 32bit操作系统,4GB内存,Intel(R) Core(TM)2 Quad CPU Q9500@2.83GHz 2.83GHz.Leukemia,Carcinoma数据集经Filter算法过滤后,保留的基因个数为700,Colon数据集保留500个基因.
将本文分别采用Pearson相关系数和Wilcoxon秩和检验进行基因预选择的基因选择算法Weight,WAC Weight,Roulette Wheel和WAC Roulette Wheel分别与采用随机选择策略选择各类簇代表基因的基因选择算法Random、经典基因选择算法SVM-RFE以及我们前期研究的基因选择算法SVM-SFS进行实验比较.
实验中,被选基因数k从1逐步增加,对于每个确定的k值,重复执行基因选择过程200次,比较各算法对不同规模基因子集的平均分类正确率Accuracy、正类的平均识别率TPR(true positive rate)、负类的误识率FPR (false positive rate)、TPR随FPR针对不同基因子集规模的变化趋势,以及确定规模的被选择基因子集对应分类模型的ROC曲线及其下面积、各算法前后两次运行选择的基因子集的基因重叠率[6]、Filter算法预选择的基因子集规模对区分基因子集的影响,最后比较了各种算法的运行时间.
2.2.1 Colon数据集实验结果分析表 2展示了分别采用Pearson相关系数和Wilcoxon秩和检验进行基因预选择后,本文的Roulette Wheel,Weight,WAC Roulette Wheel和WAC Weight算法以及SVM-RFE,SVM-SFS和Random共7种基因选择算法200次重复运行选择到的区分基因子集的平均分类正确率.因为篇幅关系,其中只列出了部分基因子集的平均分类正确率.
| Table 2 Mean accuracy of some selected gene subsets on Colon dataset 表 2 Colon数据集上各算法选择的不同规模的基因子集分类正确率平均值 |
从表 2的实验结果可以看出:
1) 本文算法整体上优于SVM-RFE,SVM-SFS和Random算法.各算法的分类正确率随基因子集规模的增大而增大:在基因子集规模K较小时,SVM-RFE和SVM-SFS算法的性能最好,本文算法次之,Random算法最差;随着K值的增大,本文算法明显优于SVM-RFE,SVM-SFS和Random算法.另外,SVM-RFE,SVM-SFS和Random算法的分类正确率随基因子集规模K的增大而逐渐增大,但增大的幅度越来越小;
2) Roulette Wheel算法的平均分类正确率最高,然后依次是WAC Roulette Wheel,Weight和WAC Weight算法.可见:对于Colon数据集,轮盘赌策略更好;
3) 基因权重的不同计算方法影响有效区分基因子集的分类正确率.在K值较小时,WAC Weight优于Weight;随着K值增大,后者要优于前者.对于轮盘赌选择策略,当K值较大时,Roulette Wheel绝对优于WAC Roulette Wheel;但在K值较小时不一定.
因此,对于Colon数据集进行有效区分基因选择,基因的类间区分能力度量只需要训练一个包含所有基因的SVM分类模型即可.
图 2展示了对于确定的被选择基因子集规模,7种基因选择算法分别在Colon数据集重复执行200次,所得基因子集对应SVM分类模型的分类正确率大于93%的次数.
图 2(a)显示:当用Pearson系数作为Filter方法进行基因预选择时,本文算法优于SVM-RFE,SVM-SFS和Random算法,其中,Weight算法最好,200次实验有60次的分类正确率超过93%;其次是Roulette Wheel,200次实验最多有50次选择的基因子集的分类正确率超过93%;接着是WAC Roulette Wheel和WAC Weight,200次重复实验中分类正确率超过93%的最高次数分别为40次和30次.而SVM-RFE算法的200次实验选择的基因子集的分类正确率超过93%的次数最多只有20次,仅占实验次数的10%;SVM-SFS选择的基因子集的分类正确率最多只有10次超过93%,占200次实验的5%;Random选择到的基因子集的分类性能极少有分类正确率高于93%的情况.
图 2(b)的实验结果显示:当采用Wilcoxon秩和检验进行基因预选择时,本文Roulette Wheel算法最优,其次是Weight算法,接着是WAC Roulette Wheel,WAC Weight,SVM-RFE和SVM-SFS算法,Random算法的性能最差.
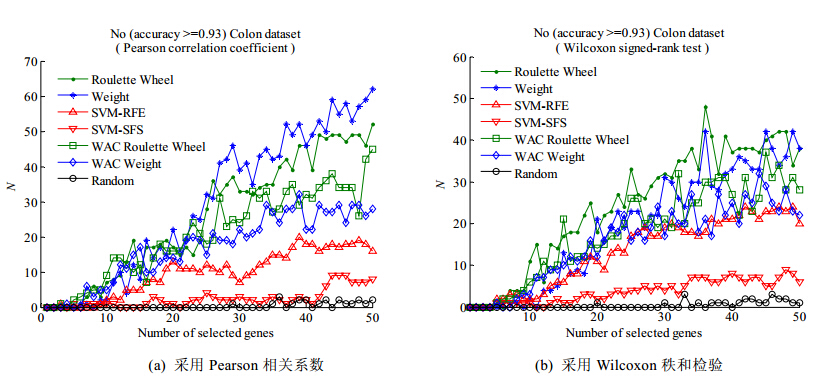 | Fig. 2 Times of the accuracy which is greater than 93% among 200 runs of 7 gene subset selection algorithms on Conlon dataset图 2 Colon数据集上,7种基因选择算法分别重复运行200次得到的基因子集的分类正确率大于93%的次数 |
以上关于图 2(a)、图 2(b)实验结果的分析是:本文算法在Colon数据集上选择到了非常好的区分基因子集;关于Colon数据集的基因权重计算方法只需要训练一个包含所有基因的SVM分类模型,与表 2的结论一致.另外,图 2(a)、图 2(b)的实验结果显示了随着基因子集规模的增加,各算法选择的基因子集的分类正确率超过93%的概率呈现上升趋势,且采用Pearson相关系数的各算法的分类正确率超过93%的概率的上升趋势更为明显.因此,对于Colon数据集,以Pearson系数为Filter方法实现有效区分基因子集选择的效果更好.
图 3展示了7种基因选择算法在Colon数据集上得到的有效区分基因子集的癌症患者识别率TPR和正常人误识率FPR分别随基因子集规模的变化情况.
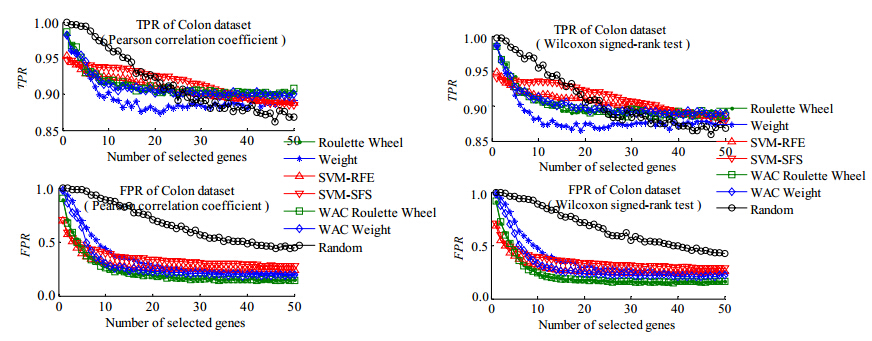 | Fig. 3 Average TPR and FPR of selected gene subsets of 7 gene subset selection algorithms on Colon dataset图 3 Colon数据集上,7种基因选择算法所得基因子集的TPR和FPR平均值随基因子集规模的变化曲线 |
从图 3(a)、图 3(b)的实验结果可以看出:随着基因子集规模K的增大,Random的TPR和FPR逐渐减小;其余6种算法的TPR和FPR先减小,后趋于稳定.
· 对正常人的误识率FPR:
Ø 当K值较小时,SVM-SFS和SVM-RFE算法较好;
Ø 随着K值的增大,本文的Roulette Wheel,Weight,WAC Roulette Wheel,WAC Weight算法较好,其中,Roulette Wheel和WAC Roulette Wheel尤为明显;
· 对癌症患者的识别率TPR:
Ø 本文算法在K值较小时,受被选基因子集规模影响较大;K值较大时趋于稳定.其中,算法Roulette Wheel和WAC Roulette Wheel的性能明显较优;
Ø Random算法在K值较小时TPR最优;但当K>15时,性能明显降低;
Ø SVM-SFS和SVM-RFE算法在K较小(<4)时的TPR不如本文算法;当4<K<35时优于本文算法;当K>35时,其TPR与本文Roulette Wheel和WAC Roulette Wheel算法的TPR相当;
Ø SVM-SFS算法的TPR优于SVM-RFE.
综合考虑各算法的癌症患者识别率TPR和对正常人的误识率FPR可见,本文基于轮盘赌选择策略的Roulette Wheel和WAC Roulette Wheel算法在Colon数据集的性能最好.
图 4给出了7种基因选择算法分别运行200次的TPR平均值随FPR平均值的在不同基因子集规模的变化趋势.图 4的实验结果显示:本文的Roulette Wheel和WAC Roulette Wheel算法可同时达到对Colon癌症患者高识别率和对正常人的低误识率;从图 4(a)、图 4(b)看出,对于Colon数据集采用Pearson系数进行基因预选择的效果更好.因为图 4(a)的Roulette Wheel和WAC Roulette Wheel算法可达到对Colon癌症患者90%的识别率,同时对正常人的误识率FPR在10%左右,优于图 4(b)的实验结果.这一结论与图 2的结论一致.
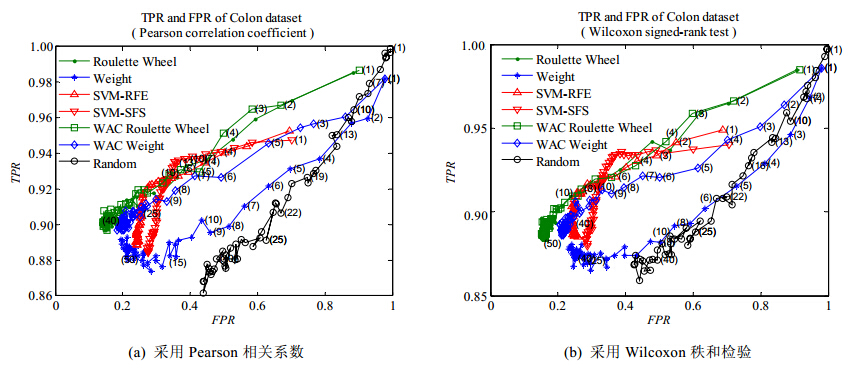 | Fig. 4 Curve of the average TPR with FPR of selected gene subsets with different size of 7 gene subset selection algorithms on Colon dataset图 4 7种基因选择算法在Colon数据集上所得不同规模基因子集的平均TPR随FPR变化曲线 |
图 5给出了采用Pearson相关系数进行基因预选择的7种基因选择算法选择的规模为15的区分基因子集的SVM分类模型的ROC曲线及其相应的AUC(area under ROC curves)值.
从图 5的ROC曲线实验结果可见:采用随机选择策略的混合基因选择算法Random的性能最差,其AUC的值仅为0.626 98;轮盘赌策略的混合基因选择算法性能最好,Roulette Wheel和WAC Roulette Wheel对应的ROC曲线下的面积AUC均为0.865 08,其中,Roulette Wheel在FPR为0的情况下对癌症患者的识别率就达到85%以上,但是随着阈值的降低,Roulette Wheel的TPR值在FPR增大到几乎与其相等时才进一步提升到93%以上;SVM-RFE算法的性能略低于轮盘赌算法,其AUC值是0.849 21;WAC Weight和SVM-SFS算法的AUC值相等,都是0.817 46,略高于Weight算法的AUC值0.801 59.以上关于规模为15的有效区分基因子集对应SVM分类模型的ROC曲线及其下面积的分析得出,本文提出的有效区分基因子集选择算法能够实现Colon数据集的有效区分基因子集选择.其中,采用轮盘赌策略的混合基因选择算法的性能最优.这与上面从不同角度的平均实验结果分析所得结论一致.
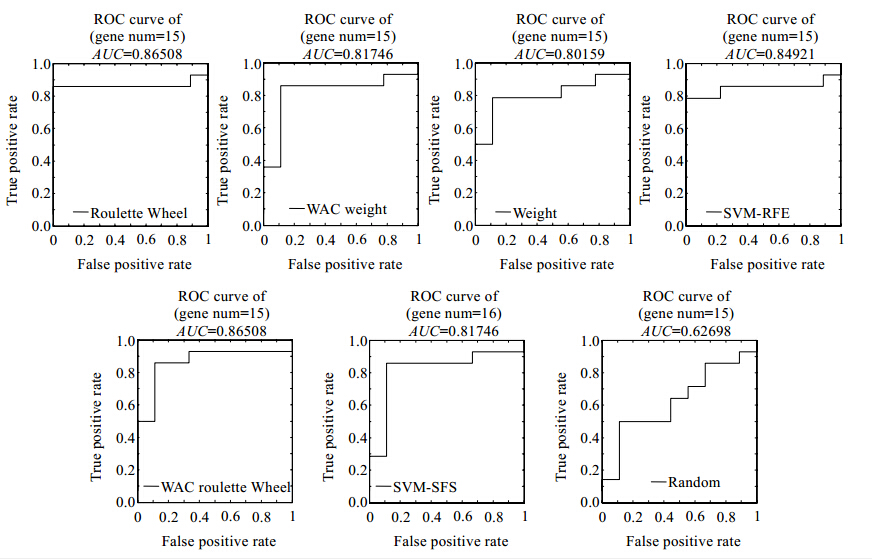 | Fig. 5 ROC curves of 7 gene selection algorithms on Colon dataset with the selected gene subset of size 15图 5 7种基因选择算法在Colon数据集的以Pearson系数为Filter方法的规模为15的基因子集的ROC曲线 |
该数据集的训练集和测试集已划分好,实验采用已有的划分进行.对于每个确定的基因子集规模K,实验重复进行200次.图 6给出了7种基因选择算法选择的有效区分基因子集的平均分类正确率曲线.
图 6(a)、图 6(b)关于各算法200次运行的平均实验结果显示:
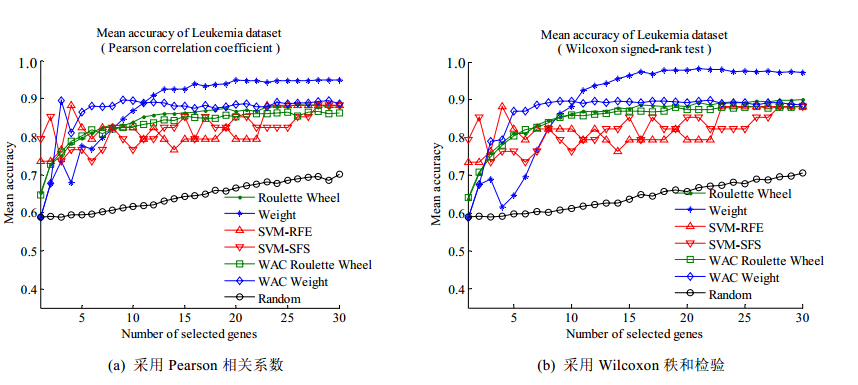 | Fig. 6 Mean accuracy of 200 runs of 7 gene subset selection algorithms on Leukemia dataset图 6 Leukemia 数据集上各基因选择算法运行200次的平均分类正确率 |
· Random算法的平均分类性能最差;
· 本文的WAC Weight算法在基因子集规模<10时,优于本文的Weight算法;但是当选择的基因子集规模>10时,本文的Weight算法最优;
· 本文的Roulette和WAC Roulette算法在ALL/AML Leukemia数据集上的分类性能差别不大,在选择的基因子集规模稍大时,Roulette的性能略优于WAC Roulette;
· SVM-RFE与SVM-SFS算法在ALL/AML Leukemia数据集的平均性能不如本文提出的4种基因选择算法.
另外,从图 6(a)、图 6(b)可见:在ALL/AML Leukemia数据集,本文的4种基因选择算法采用Wilcoxon秩和检验作为Filter算法进行基因预选择时的分类性能优于采用Pearson系数作为预选择策略时的分类性能.
Ding等人[6]用特征重叠率反映两个算法选择的特征子集的重合度,本文借用特征重叠率来统计相同基因子集规模下,同一个算法先后两次运行选择的基因子集中相同基因的比率,以此来比较各算法的稳定性.对ALL/AML Leukemia数据集,7种基因选择算法在相应基因子集规模下重复运行200次选择的基因子集的基因重叠率平均值如图 7所示.
 | Fig. 7 Average intersection of genes of selected gene subsets of 7 gene subset selection algorithms on ALL/AML Leukemia dataset图 7 Leukemia数据集上,7种基因选择算法选择的基因子集的基因重叠率平均值 |
从图 7(a)、图 7(b)的实验结果可以看出:SVM-SFS和SVM-RFE算法选择的基因子集的基因重叠率是1,而Random方法的基因重叠率是0.这是因为数据集ALL/AML Leukemia的训练集和测试集已经划分好,特征选择在完全相同的训练集上进行且样本的先后顺序不变.Weight算法当基因子集规模分别为1,2和4时,选择的基因子集的基因完全相同;随着基因子集规模的增大,基因子集中基因的重叠率先减小;当基因重叠率减小到60%时,基本不再变化.这是因为类簇数K开始增加时,K-means的聚类结果不稳定;但是当K增加到一定程度时,K-means的聚类结果的不稳定性开始减小.WAC Weight选择的基因子集的基因重叠率变化趋势与Weight算法相同,但基因的重叠率低于Weight算法的基因重叠率.这是因为Weight算法各基因的权重计算是训练一个包含所有基因的SVM分类模型得到,而WAC Weight算法的基因权重计算是训练类簇数个SVM分类模型得到,K-means聚类结果的不稳定影响类簇中的基因,进而影响WAC Weight算法的基因权值,因此,WAC Weight的基因重叠率低于Weight.Roulette Wheel和WAC Roulette Wheel选择的基因子集的基因重叠率随着基因子集规模K的增大而逐渐增大,最终趋于稳定到大约30%,低于Weight和WAC Weight算法的基因重叠率,且Roulette Wheel和WAC Roulette Wheel的基因重叠率非常接近.这是因为在备选基因子集很大而被选基因子集规模较小时,轮盘赌策略使得基因被选中的随机性很大;随着被选择基因子集规模K的增大,类簇中基因个数减少,权重较大的基因被选中的概率增加.因此,随着基因子集规模的增加,Roulette Wheel和WAC Roulette Wheel选择的基因子集的基因重叠率单调增加.假设Roulette Wheel算法在选择各类簇得票数最高的基因时,选择到相应类簇最好基因的概率为0.7(由于类簇中基因个数较多,这个概率假设已经比较高),则理想情况下,选择10个最好基因组合在一起的概率为0.710=0.0282.由此可见,轮盘赌的固有随机性,使得最好基因组合在一起的概率极大地降低.故Roulette Wheel选中的基因在很大程度上是基于权重的“次优”组合,导致其基因重叠率低于Weight和WAC Weight算法的基因重叠率.
图 8给出了200次实验中分类正确率为1的次数.从图 8(a)、图 8(b)的实验结果可见:SVM-RFE,SVM-SFS和Random的效果很差,没有选择到分类正确率为1的基因子集.
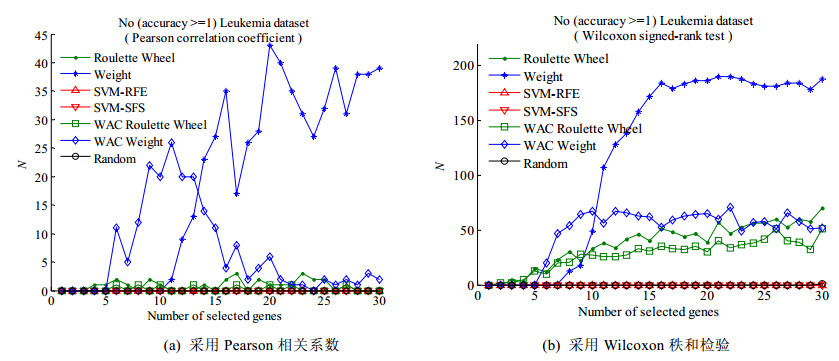 | Fig. 8 Times of the accuracy equaling 1 of 7 gene subset selection algorithms among their 200 runs on ALL/AML Leukemia dataset图 8 7种基因选择算法在Leukemia数据集的200次重复实验中分类正确率等于1的次数 |
图 8(b)的实验结果显示:本文Weight算法在选择的基因子集规模K>10时,200次实验中超过100次能选择到分类正确率等于1的区分基因子集;当基因子集规模K>15时,200次重复实验中180次的分类正确率达到1,占实验次数的90%,非常容易找到能够实现正确分类的基因子集;
本文提出的WAC Weight,Roulette Wheel和WAC Roulette Wheel算法当基因子集规模大于10时,不如本文的Weight算法,但是优于SVM-RFE,SVM-SFS和Random算法.
图 8(a)的实验结果显示:当采用Pearson相关系数为Filter方法对基因进行预选择时,本文提出的WAC Weight算法在基因规模子集为5~13之间时,其性能优于Weight算法;但是随着基因子集规模的增大,Weight的性能绝对优于WAC Weight及其他算法.原因是WAC Weight的基因权重是训练类簇数个SVM分类模型计算得到,Weight是训练一个包含所有基因的SVM分类模型计算的基因权重,随着类簇数K的增加,K-means聚类结果的不稳定对WAC Weight的影响大于其对Weight的影响,因此,WAC Weight算法的性能在基因子集规模变大时不如Weight.图 8(a)的实验结果还显示:以Pearson相关系数为Filter算法对基因进行预选择时,本文提出的Roulette Wheel和WAC Roulette Wheel算法性能很差,只略优于SVM-RFE,SVM-SFS和Random算法.
图 8(a)、图 8(b)的实验结果共同显示:对于ALL/AML Leukemia数据集,本文提出的Weight算法具有非常好的性能,能够筛选到非常好的有效区分基因子集;对该数据集,Wilcoxon秩和检验比Pearson系数作为Filter方法进行基因预选择的效果更好,与图 6的结论一致.
图 9所示为200次运行的平均TPR和平均FPR随基因子集规模的变化情况.
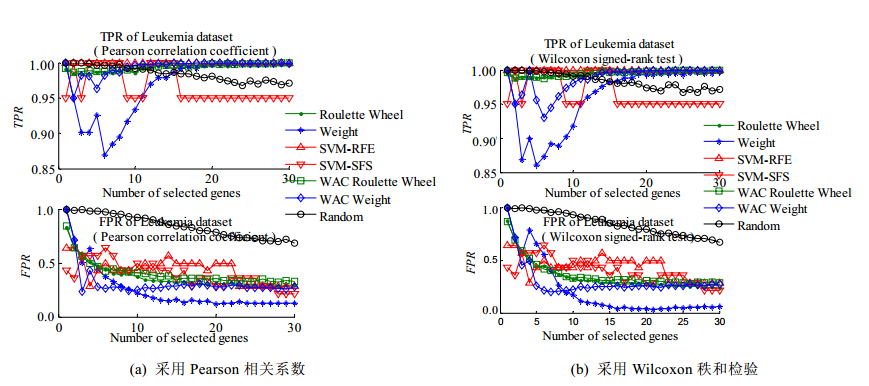 | Fig. 9 Variation of average TPF and FPR with the size of the selected gene subset of 7 gene subset selection algorithms on ALL/AML Leukemia dataset图 9 7种基因选择算法在Leukemia数据集上选择的基因子集的平均TPR和FPR随基因子集规模变化情况 |
图 10给出了针对不同规模基因子集的平均TPR随FPR的变化曲线.
 | Fig. 10Curve of the average TPR with FPR of the selected gene subset with different size of 7 gene subset selection algorithms on ALL/AML Leukemia dataset图 10 Leukemia数据集上,7种基因选择算法选择的不同规模基因子集的平均TPR随FPR变化曲线 |
从图 9(a)、图 9(b)的实验结果可以看出:Weight算法在K值较小时,对K值很敏感;当K较大(>15)时趋于稳定,此时,TPR接近于1而FPR接近0.1,说明选择的基因子集较好.图 10(a)、图 10(b)的实验结果也显示,只有Weight算法在ALL/AML Leukemia数据集上可以选择到TPR接近1而FPR接近0.1的基因子集.
表 3比较了本文算法和现有相关基因选择算法[1,6,8,13,15,29-33]在Colon和ALL/AML Leukemia数据集的实验结果.
| Table 3 The comparison between the experimental results of our gene subset selection algorithms and the available others on Colon and ALL/AML Leukemia datasets 表 3 本文基因选择算法在Colon和ALL/AML Leukemia数据集的实验结果与现有研究结果的比较 |
由表 3的实验结果可见:对于ALL/AML Leukemia数据集,本文提出的混和基因选择算法都能达到100%的识别正确率,区别在于选择的有效区分基因子集的规模大小不完全相同.当Filter算法采用Wilcoxon秩和检验时,Roulette Wheel和WAC Roulette Wheel算法使用5个基因达到完全正确的识别,Weight和WAC Weight需要9个基因达到完全正确的识别.当采用Pearson相关系数作为Filter算法时,Roulette Wheel和WAC Roulette Wheel分别需要4个和17个基因,Weight和WAC Weight分别需要11个和9个基因.与现有的基因选择算法在ALL/AML Leukemia数据集的研究结果相比,本文算法取得了很好的效果.
对于Colon数据集,当采用Wilcoxon秩和检验准则时,本文提出的混合基因选择算法Roulette Wheel在选择到5个基因时达到94.3%的分类正确率,选择到13个基因时达到97.6%的正确识别率,当基因子集规模为28时,分类正确率达到99.1%;Weight策略在选择到18个基因时的正确识别率是99.2%,基因子集规模为28时达到完全无误的分类;WAC Roulette Wheel在基因子集规模为5时的正确识别率是94.5%,略高于Roulette Wheel的94.3%,但其最高分类正确率只有97.5%,不如Roulette Wheel的最高分类正确率1.WAC Roulette Wheel和WAC Weight的性能不如Roulette Wheel和Weight,但其分类性能也优于现有的其他研究结果.与现有研究结果相比,本文的Roulette Wheel和Weight取得了非常好的效果,WAC Weight和WAC Roulette Wheel也取得了不错的效果.当采用Pearson相关系数为Filter算法时,本文算法也取得了不错的效果,分类效果优于现有的其他研究结果.
以上分析揭示:本文提出的4种混合基因选择算法能选择到非常有效的区分基因子集,建立在该区分基因子集上的SVM分类器具有非常好的分类性能.
2.2.3 Carcinoma数据集实验结果分析图 11所示本文提出的4种混合基因选择算法与Random,SVM-RFE和SVM-SFS共7种基因选择算法在Carcinoma数据集重复运行200次选择的基因子集的平均分类正确率曲线.
图 11(a)、图 11(b)各算法分别运行200次选择的平均分类正确率变化曲线揭示:Roulette Wheel和WAC Roulette Wheel性能最好,其次是Weight和WAC Weight,接着是SVM-RFE和SVM-SFS,特别是当选择的有效区分基因子集规模稍大时,这6种算法的性能越来越接近;Random算法的性能较差,远不如其他6种算法;在基因子集规模较小(<10)时,SVM-RFE的性能明显优于SVM-SFS.
 | Fig. 11Curve of the average accuracy of the 7 gene subset selection algorithms on Carcinoma dataset图 11 7种基因选择算法在Carcinoma数据集选择的基因子集的平均分类正确率变化曲线 |
图 12给出了各算法200次重复运行选择的基因子集的分类正确率为1的次数.
 | Fig. 12Times of accuracy equaling 1 of 7 gene subset selection algorithms among 200 runs on Carcinoma dataset图 12 7种基因选择算法在Carcinoma数据集上分别运行200次选择的基因子集的分类正确率为1的次数 |
图 12(a)、图 12(b)的实验结果显示:Roulette Wheel和WAC Roulette Wheel的性能最好,当选择的基因子集规模大于10时,200次运行有180次达到100%的分类正确率;其次是Weight和WAC Weight算法以及SVM-RFE与SVM-SFS算法;Random的性能最差.其中,SVM-RFE在基因子集规模小于10时,选择的基因子集达到完全正确分类的次数高于Weight和WAC Weight算法;特别是当基因子集规模小于3时,SVM-RFE选择的基因子集实现完全正确分类的概率甚至高于本文提出的Roulette Wheel和WAC Roulette Wheel算法.
图 13所示各算法选择的基因子集的平均TPR和平均FPR分别随基因子集规模的变化曲线.
 | Fig. 13Curve of average TPR and FPR with the size of the selected gene subset of 7 gene subset selection algorithms on Carcinoma dataset图 13 7种基因选择算法在Carcinoma数据集选择的基因子集的平均TPR和FPR随基因子集规模变化曲线 |
图 13(a)、图 13(b)关于7种基因选择算法选择的基因子集的SVM分类模型的平均TPR和FPR随基因子集规模的变化曲线显示:除了Random,其他6种算法的TPR随基因子集规模的增加单调上升,并趋于1;FPR随基因子集规模的增加单调下降,并趋于0.Random的TPR和FPR大体分别服从随基因子集规模增加单调上升和增加单调下降趋势,除了在个别基因子集规模上的小跳跃,但其TPR的上线没有超过0.8,FPR的下限在0.2左右.
图 14给出了各算法选择的基因子集的平均TPR随FPR在不同基因子集规模的变化曲线.
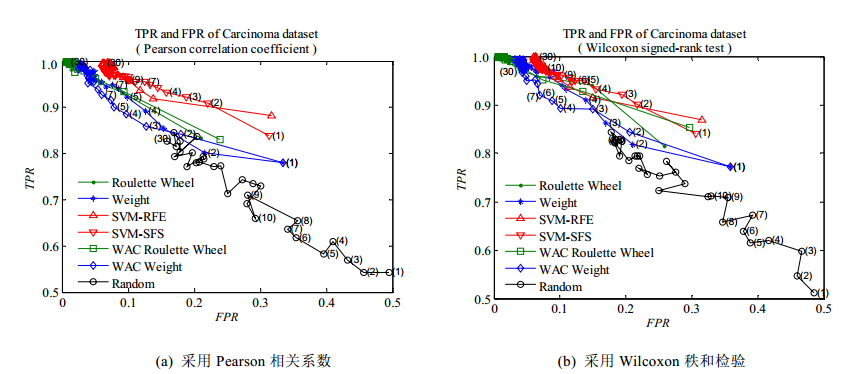 | Fig. 14 Curve of the average TPR with FPR on different size of selected gen subsets of 7 gene subset selection algorithms among their 200 runs on Carcinoma dataset图 14 Carcinoma数据集上,7种基因选择算法200次运行选择的不同规模基因子集的平均TPR随FPR变化曲线 |
图 14(a)、图 14(b)的实验结果显示:能使TPR达到1而FPR小于0.05的,只有本文提出的基于轮盘赌策略的基因选择算法Roulette Wheel和WAC Roulette Wheel以及基于权重策略的基因选择算法Weight和WAC Weight;SVM-RFE与SVM-SFS对癌症患者的识别率TPR达到1时,将正常人误判为癌症患者的概率超过了本文提出的混合基因选择算法,达到0.05和0.1之间;Random算法性能最差,对癌症患者的最高识别率不足85%,但此时误将正常人识别为患者的概率超过了0.15,即15%.
由此可见:对于Carcinoma数据集,本文提出的混合基因选择算法具有非常好的性能,能选择到非常有效的区分基因子集,实现将癌症患症从正常人群中识别出来的目的.
图 15是各算法200次重复运行中前后两次选择的基因子集的基因重叠率平均值.
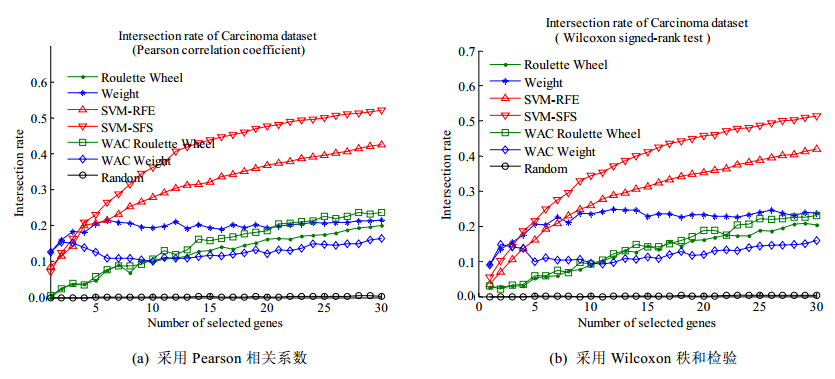 | Fig. 15Average intersection of genes of selected gene subset of 7 gene subset selection algorithms on Carcinoma dataset图 15 Carcinoma数据集上,7种基因选择算法选择的基因子集的基因重叠率平均值 |
图 15(a)、图 15(b)的实验结果显示:无论采用Pearson相关系数还是Wilcoxon秩和检验进行基因预选择,Random方法在Carcinoma数据集上选择的基因子集的重叠率都是0.SVM-RFE和SVM-SFS算法选择的基因子集的基因重叠率随着基因子集规模的增加而单调上升,其中,SVM-SFS算法选择的基因子集的基因重叠率趋向超过50%,SVM-RFE算法选择的基因子集的基因重叠率趋向高于40%,且SVM-SFS算法的基因重叠率曲线高于SVM-RFE算法的基因重叠率曲线.因此,对Carcinoma数据集,SVM-SFS选择的基因子集更稳定.Roulette Wheel和WAC Roulette Wheel算法选择的基因子集的基因重叠率也呈现随基因子集规模单调上升的趋势,但其重叠率远小于SVM-RFE和SVM-SFS.Weight和WAC Weight算法选择的基因子集的基因重叠率随基因子集规模的变化较为平稳,Weight平均大约为20%,WAC Weight仅达到15%左右;但在基因子集规模小于5时,Weight和WAC Weight选择的基因子集的基因重叠率高于其他5种基因选择算法.
从图 15(a)、图 15(b)明显可见,本文提出的混合基因选择算法在Carcinoma数据集选择的基因子集的基因重叠率最高不超过30%.这是因为该数据集上的实验采用bootstrap方法划分数据集,使得每次的训练集不同,导致基因的权重、K-means的聚类结果均不同,所以Weight,SVM-RFE和SVM-SFS方法选择的特征子集的基因重叠率低于其在固定划分的Leukemia数据集选择的基因子集的基因重叠率.Roulette Wheel,WAC Roulette Wheel和Random本身固有的随机性,使其基因重叠率受数据集不同划分方法的影响较小.图 7和图 15的基因重叠率实验结果揭示,本文的Weight算法较为稳定.
2.2.4 预选择基因子集规模对有效区分基因子集的影响本文基因权重计算方法使其权重与Filter方法预选择的基因子集规模密切相关.本节采用Colon数据集,研究Filter方法预选择的基因子集的规模对最后选择的有效区分基因子集的影响.
前面的对比实验显示,Pearson相关系数是进行基因预选择的有效准则.因此,考虑采用Pearson相关系数为Filter方法进行基因预选择,且保留的备选(预选择)基因个数分别为100,200,500,1 000,1 500,2 000;然后,从备选基因中选择1~50个基因构成有效区分基因子集.下面从各算法的运行时间、选择的有效区分基因子集的分类正确率均值及其方差以及选择的有效区分基因子集的基因重叠率几方面进行讨论.选择的区分基因子集的分类正确率计算采用第1.5节的公式(6)进行.各算法重复执行200次,计算各项指标的平均值.图 16给出了本文4种基因选择算法在不同规模的预选择基因子集上进行有效区分基因子集选择的200次运行的总时间(单位:s).
 | Fig. 16 Run time of the proposed hybrid gene subset selection algorithms' 200 runs on different size of the candidate gene subsets on Colon dataset图 16 本文提出的各算法在Colon数据集上,针对不同规模预选择基因子集重复运行200次的运行时间 |
从图 16的实验结果可见:随着预选择基因数的增加,本文算法的运行时间先减少后增多.其原因是:由第1.6.1节的算法时间复杂度分析得知,本文算法的时间复杂度为O(nd+tkmn),因此在采用相应的Filter方法进行基因预选择之后,算法的时间开销主要是K-means的聚类时间.K-means的时间复杂度为O(tmnk),t为K-means算法的迭代次数,n为数据集的样本数,m是预选择的基因数,k是类簇数.当K-means聚类的样本数目(本文是预选择基因数m)较少而类簇数k较大时,算法很难收敛到一个稳定的类簇分布,往往是达到最大的迭代阈值而停止,因此在预选择基因子集规模较小时消耗时间较多.图 16的预选择基因数100和1 000,前者是后者的1/10,但前者耗时大约是后者2倍,与以上理论分析吻合.另外,图 16的实验结果显示:在预选基因子集规模相同条件下,Weight和WAC Weight算法较Roulette Wheel和WAC Roulette Wheel算法的运行效率更好、更省时间.
图 17、图 18分别给出了各算法在不同规模的预选择基因子集上重复运行200次,选择的有效区分基因子集的分类正确率均值及其方差随选择的区分基因子集规模的变化情况.为了表示方便,以各算法在规模500的预选择基因子集上进行基因选择所得到的有效区分基因子集的分类正确率均值及其方差为基线,展示各算法在相应规模的预选择基因子集上进行基因选择得到的有效区分基因子集的平均分类正确率及其方差分别随被选择区分基因子集规模的变化情况.
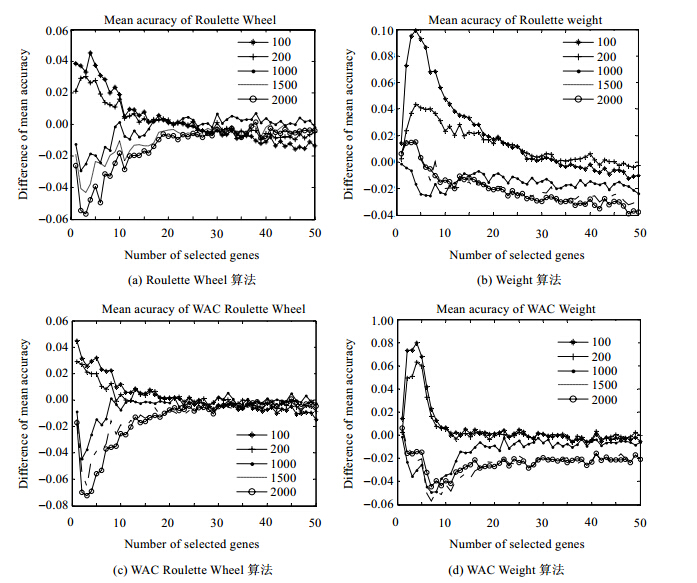 | Fig. 17Influence of the size of the pre-selected gene subsets on the accuracy of the classifiers based on the selected gene subsets by the algorithms proposed in this paper图 17 不同预选择基因子集规模对本文算法选择的区分基因子集的分类正确率的影响 |
 | Fig. 18 Accuracy variance of the classifiers on the selected gene subsets of the proposed algorithms 图 18 本文各算法在不同规模预选择基因子集选择的区分基因子集分类正确率的方差 |
从图 17(a)和图 17(c)的实验结果可见:K值(有效区分基因子集规模)较小(<15)时,预选择基因子集规模越小,本文算法Roulette Wheel和WAC Roulette Wheel选择的区分基因子集的分类正确率越高;随着被选基因子集规模K的增大,Roulette Wheel和WAC Roulette Wheel选择的区分基因子集的分类正确率均值逐渐减小,并趋于稳定.在被选择基因子集规模K较小、预选择基因子集规模较大的情况下,本文的混合基因选择算法Roulette Wheel和WAC Roulette Wheel选择的基因子集的平均分类正确率较小;随着被选择基因子集规模K的增大,Roulette Wheel和WAC Roulette Wheel选择的基因子集的平均分类正确率逐渐上升,并趋于稳定.无论预选择基因子集规模大还是小,随着被选择基因数K的增加,本文算法Roulette Wheel和WAC Roulette Wheel选择的区分基因子集的分类正确率趋于稳定与一致.这是因为轮盘赌算法在K值较小时,预选择基因子集规模越小,则随机性越小,而预选择基因子集规模越大,则随机性越大;而当被选择基因子集规模K逐渐增大时,轮盘赌方法选择的基因子集的随机性逐渐降低,并趋于稳定.
图 17(b)和图 17(d)展示的实验结果显示:在预选择因子集规模较小、被选择基因子集规模也较小时,本文的Weight和WAC Weight方法和轮盘赌选择基因子集的方法有类似的结论.但是从图 17(d)可见,尽管在预选择基因子集规模较大时,随着基因子集规模K的增大,WAC Weight选择的基因子集的分类正确率逐渐上升,并趋于稳定,但是当预选择基因子集规模超过1 000时,WAC Weight算法选择的基因子集的分类正确率始终低于预选择基因子集小于1 000时其选择的基因子集的分类正确率,只有预选择基因子集规模为1 000时,其选择的基因子集的分类正确率趋于稳定到预选择基因子集规模小于1 000时的情况.图 17(b)展示的Weight方法的实验结果显示:在预选择基因子集规模超过1 000时,随着被选择基因子集规模K的增大,选择的基因子集的分类正确率呈微小下降趋势;预选择基因子集规模为1 000时,选择的基因子集的分类正确率虽然低于基线,但是基本稳定;预选择基因子集小于1 000时,随着选择的有效区分基因数的增加,Weight算法选择的有效区分基因子集的分类正确率均值呈现下降趋势,并低于WAC Weight的相应值,原因是WAC Weight和Weight的基因权重计算依赖的SVM分类模型不一样,前者训练类簇数个SVM分类器,后者训练一个SVM分类器.
从图 18(a)和图 18(c)的实验结果来看:当预选的基因数较少时,若被选择的基因数K值较小,Roulette Wheel和WAC Roulette Wheel选择的基因子集的分类正确率方差较小,且方差随选择的基因数K的增大而逐渐增大,最终趋于稳定.当预选择的基因子集规模较大时,若选择的基因子集规模较小,则它们选择基因子集的分类正确率的方差较大,且随选择的有效区分基因子集规模的增大而下降,并趋于稳定.
图 18(b)和图 18(d)的实验结果显示:当选择的基因子集规模较小时,无论预选择的基因子集规模大还是小,Weight和WAC Weight选择的基因子集的分类正确率的方差很不稳定,在有效区分基因数K=5时达到最小,然后随选择的区分基因数K的增多,方差开始上升并趋于稳定.图 18(b)的实验结果明显看出:在预选择基因子集规模较大(>=1000)时,Weight选择的基因子集的分类正确率的方差随选择的基因数的增加趋于稳定,收敛到略高于基线;但当预选择的基因子集规模较小(<1000)时,其方差尽管也随有效区分基因子集规模的增加而上升和趋于稳定,但是最终的收敛值略低于基准线.图 18(d)的结果显示:无论预选择的基因子集规模大小,WAC Weight选择的基因子集的分类正确率方差最终都近似收敛到基准线.
造成图 18(b)、图 18(d)不同实验结果的原因是:Weight在计算基因权重时,是训练一个包含全部预选择基因的SVM分类模型,而WAC Weight是在K-means聚类基因之后训练K个SVM分类模型,分别计算各类簇基因的权重.
图 17、图 18不同规模的预选择基因子集对有效区分基因子集的分类正确率及其方差的影响实验分析揭示:当被选择基因子集规模K较小时,预选择的基因越少越好,此时,选择的区分基因子集的分类正确率均值越大,且方差越小,这意味着选择到的基因子集的性能越稳定;当被选择的基因子集规模较大时,预选择的基因多少对选择的区分基因子集性能的影响不大.另外,从选择的有效区分基因子集的性能稳定性(正确率的方差)来看,WAC Roulette Wheel最好,其次是Roulette,接着是WAC Weight和weight.
图 19展示了本文提出的混合基因选择算法重复运行200次,前后两次运行选择的基因子集的平均基因重叠率.
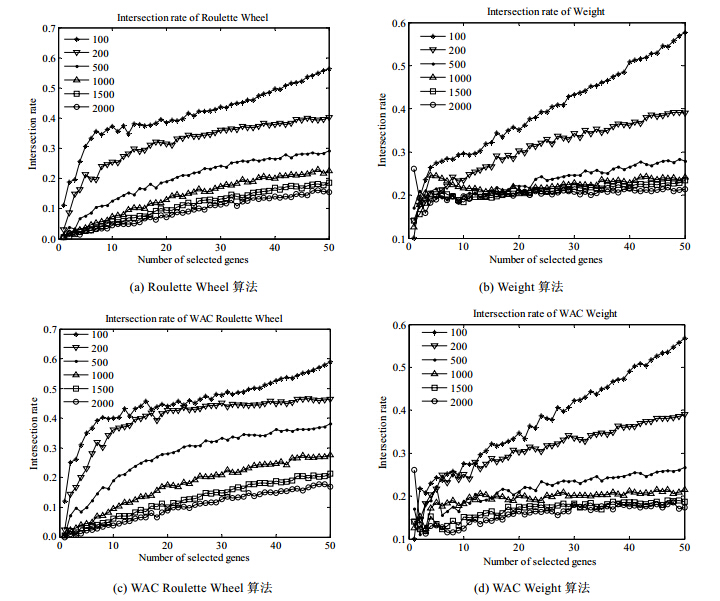 | Fig. 19 Average intersection rates of the selected genes of the proposed hybrid gene subset selection algorithms among their 200 runs图 19 本文基因选择算法在不同规模的预选基因子集上200次运行选择的基因子集的平均基因重叠率 |
从图 19的基因重叠率实验结果看出:
· 对于确定的预选择基因子集,被选择基因子集的基因重叠率随被选择基因子集规模的增大而上升;
· 相同的被选择基因子集规模,预留基因子集规模越小,被选基因子集的基因重叠率越高;
· Weight和WAC Weight在预选择的基因子集规模较大(>1000)时,选择的基因子集的基因重叠率几乎不受预选择基因子集规模的影响,再次证明了Weight算法选择的基因子集的基因稳定性.
通过以上关于预选择基因子集规模对有效区分基因子集影响的分析得知,从运行时间和选择的有效区分基因子集的基因稳定性来看,本文提出的Weight算法最优,其次是WAC Weight,接着是Roulette Wheel和WAC Roulette Wheel;从选择的有效区分基因子集的分类正确率及其方差,即从选择的有效区分基因子集的性能稳定性(正确率的方差)来看,WAC Roulette Wheel最好,其次是Roulette,接着是WAC Weight和weight.
2.2.5 7种基因选择算法的运行效率比较这里进一步比较本文算法和SVM-RFE,SVM-SFS与Random算法的运行时间,以验证第1.6.1节关于本文算法时间复杂度的理论分析.实验采用Colon数据集,Filter算法采用Pearson相关系数进行基因预选择,预留基因数为500,实验重复200次.7种基因选择算法的运行时间如图 20所示(时间单位为s).
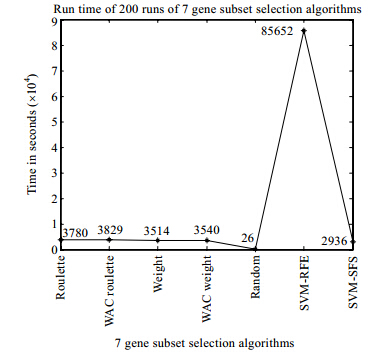 | Fig. 20Run time of 200 runs of the 7 gene subset selection algorithms图 20 7种算法在Colon数据集上重复运行200次的时间 |
从图 20所示的实验结果可知:算法200次重复运行所需的时间,Random最少;其次是我们之前提出的SVM- SFS算法;本文各算法的时间性能大致相当,均在3500s左右,Weight和WAC Weight低于Roulette Wheel和WAC Roulette Wheel,Weight稍低于WAC Weight;SVM-RFE算法所需时间最多,大概是本文算法的25倍.
这一实验结果与第1.6.1节关于本文算法的时间复杂度理论分析结论吻合,也验证了本文算法的时间消耗主要来源于K-means的聚类时间.
第2.2.1节~第2.2.5节的实验结果显示:本文提出的基于统计相关性与K-means聚类几种混合基因选择算法不仅能够选择到分类正确率非常好的区分基因子集,且时间性能接近SVM-SFS,优于经典的基因选择算法SVM-RFE;Random算法时间效率最高,但其选择的基因子集的性能最差;SVM-SFS算法的时间效率远优于SVM-RFE,且选择的基因子集性能与SVM-RFE相当,但SVM-SFS与SVM-RFE的共同缺陷是,每次迭代加入或剔除的基因数会影响基因选择的结果.
3 结论与展望本文提出了几种混合的基因选择算法,先分别使用Pearson相关系数和Wilcoxon秩和检验度量基因与类标的相关性大小,对基因进行预选择,保留相关性大的基因,剔除相关性较小(分类能力较弱)的基因;然后,采用K-means对预选择基因进行聚类,使相关性较强的基因聚集在同一类簇,相关性较弱的基因分布在不同类簇,从每个类簇中选择一个代表性基因作为本类簇基因子集的代表,各类簇的代表基因构成有效区分基因子集.其中,各类簇代表基因的选择,通过训练SVM学习机计算每个基因的权重,选择各类簇中权值最大的基因代表本类簇,或者采用轮盘赌的投票方式选择每个类簇‘得票数’最多的基因代表本类簇.
3个经典基因数据集的实验结果发现:本文算法选择的基因子集的分类性能优于Random,SVM-RFE和SVM-SFS算法选择的区分基因子集的分类性能,且运行时间远小于经典基因选择算法SVM-RFE,与我们之前提出的SVM-SFS算法的时间性能相当.不同的数据集划分方法适用本文的不同算法和不同的Filter预选择基因方法:Roulette Wheel和WAC Roulette Wheel算法适宜采用Bootstrap方法划分的数据集的有效区分基因子集选择;Weight和WAC Weight适宜采用Holdout的固定划分的数据集的有效区分基因子集选择;Wilcoxon秩和检验对训练集和测试集已经划分好的基因数据集更适用.
有效区分基因子集的基因稳定性研究发现,本文提出的4种区分基因子集选择算法的基因稳定性不如经典基因选择算法SVM-RFE和我们前期研究的基因选择算法SVM-SFS.就本文的4种基因选择算法来说,Weight算法选择的基因稳定性最好,其次是WAC Weight算法;Roulette Wheel和WAC Roulette Wheel算法的基因稳定性不相上下.因此,基因权重计算训练一个包含所有预选择基因的SVM分类模型即可.
Filter算法剔除不重要基因后的预选择基因子集规模对有效区分基因子集的影响研究发现:从运行时间和选择的有效区分基因子集的基因稳定性来看,本文提出的Weight算法最优,其次是WAC Weight,接着是Roulette Wheel和WAC Roulette Wheel;从选择的有效区分基因子集的分类正确率及其方差,即从选择的有效区分基因子集的性能稳定性(正确率的方差)来看,WAC Roulette Wheel最好,其次是Roulette,接着是WAC Weight和weight.
但是,本文算法选择的基因子集的基因重叠率,即,选择的基因子集的基因稳定性还有待进一步提升.如何找到稳定的特征子集,很多学者进行了相关研究[15,34,35],在特征子集的特征稳定性方面有了很大的提高,然而特征子集的质量(分类能力)却受到了不同程度的影响.本文算法在选择高质量的特征子集方面取得了非常好的效果,同时,在训练集和测试集划分固定的情况下,选择的基因子集的基因重叠率高达60%.如何在保证选择到高质量特征子集的情况下找到稳定的特征子集,以便给医学界提供一个非常有价值的参考,是我们未来的一个研究方向.另外,将本文方法推广于高维多样本数据集的特征选择,也是我们未来的研究方向.
| [1] | Li ST, Wu XX, Hu XY. Gene selection using genetic algorithm and support vectors machines. Soft Computing, 2008,12(7): 693-698 . |
| [2] | Guyon I, Elisseeff A. An introduction to variable and feature selection. Journal of Machine Learning Research, 2003,3: 1157-1182. |
| [3] | 张军英,Wang YJ, Khan J, Clarke R.基于类别空间的基因选择.中国科学(E辑), 2003,33(12):1125-1137. |
| [4] | Li YX, Li JG, Ruan XG. Study of inofrmative gene selection for tissue classification based on tumor gene expression profiles. Chinese Journal of Computers, 2006,29(2):324-330 (in Chinese with English abstract). |
| [5] | Xie JY, Xie WX. Several feature selection algorithms based on the discernibility of a feature subset and support vector machines. Chinese Journal of Computers, 2014,37(8):1704-1718 (in Chinese with English abstract). |
| [6] | Ding C, Peng HC. Minimum redundancy feature selection from microarray gene expression data. Journal of Bioinformatics and Computational Biology, 2005,3(2):185-205 . |
| [7] | Guyon I, Weston J, Barnhill S, Vapnik V. Gene selection for cancer classification using support vector machines. Machine Learning, 2002,46(1-3):389-422 . |
| [8] | Niijima S, Kuhara S. Recursive gene selection based on maximum margin criterion: A comparison with SVM-RFE. BMC Bioinformatics, 2006,7(1):543 . |
| [9] | Wang YH, Makedon FS, Ford JC, Pearlman J. HykGene: A hybrid approach for selecting marker genes for phenotype classification using microarray gene expression data. Bioinformatics, 2005,21(8):1530-1537 . |
| [10] | Han JW, Kamber M. Data Mining: Concepts and Techniques. 2nd ed., San Francisco: Morgan Kaufmann Publishers, 2006. 383-386. |
| [11] | Deng L, Pei J, Ma JW, Lee DL. A rank sum test method for informative gene discovery. In: Elder J, ed. Proc. of the 10th ACM SIGKDD Int’l Conf. on Knowledge Discovery and Data Mining. Seattle: ACM Press, 2004.410-419 . |
| [12] | Weston J, Elisseeff A, Schölkopf B, Tipping M. Use of the zero norm with linear models and kernel methods. The Journal of Machine Learning Research, 2003,3:1439-1461. |
| [13] | Song QB, Ni JJ, Wang GT. A fast clustering-based feature subset selection algorithm for high-dimensional data. IEEE Trans. on Knowledge and Data Engineering, 2013,25(1):1-14 . |
| [14] | Yu L, Liu H. Efficient feature selection via analysis of relevance and redundancy. Journal of Machine Learning Research, 2004, 5:1205-1224. |
| [15] | Loscalzo S, Yu L, Ding C. Consensus group stable feature selection. In: Elder J, ed. Proc. of the 15th ACM SIGKDD Int’l Conf. on Knowledge Discovery and Data Mining. Paris: ACM Press, 2009.567-576 . |
| [16] | MacQueen JB. Some methods for classification and analysis of multivariate observations. In: LeCam LM, Neyman J, eds. Proc. of the 5th Berkeley Symp. on Mathematical Statistics and Probability. Berkeley: University of California Press, 1967. 281-297. |
| [17] | Huang ZX. Extensions to the k-means algorithm for clustering large data sets with categorical values. Data Mining and Knowledge Discovery, 1998,2(3):283-304 . |
| [18] | Boser BE, Guyon IM, Vapnik VN. A training algorithm for optimal margin classifiers. In: Haussler D, ed. Proc. of the 5th Annual Workshop on Computational Learning Theory. New York: ACM Press, 1992.144-152 . |
| [19] | Vapnik VN. Statistical Learning Theory. New York : Jobn Wiley & Sons, 1998. |
| [20] | Cristianini N, Shawe-Taylor J. An Introduction to Support Vector Machines and Other Kernel-Based Learning Methods. United Kingdom: Cambridge University Press, 2000. |
| [21] | Huang JZ, Ng MK, Rong HQ, Li ZC. Automated variable weighting in k-means type clustering. IEEE Trans. on Pattern Analysis and Machine Intelligence, 2005,27(5):657-668 . |
| [22] | Witten IH, Frank E, Hall MA. Data Mining: Practical Machine Learning Tools and Techniques. 3rd ed., United States: Morgan Kaufmann Publishers, 2010. 147-187. |
| [23] | Tang YC, Zhang YQ, Huang Z. Development of two-stage SVM-RFE gene selection strategy for microarray expression data analysis. IEEE/ACM Trans. on Computational Biology and Bioinformatics, 2007,4(3):365-381 . |
| [24] | Cormen TH, Leiserson CE, Rivest RL, Stein C. Introduction to Algorithms. 3rd ed., Cambridge: MIT Press, 2009. 1-14. |
| [25] | Golub TR, Slonim DK, Tamayo P, Huard C, Gaasenbeek M, Mesirov JP, Coller H, Loh ML, Downing JR, Caligiuri MA, Bloomfield CD, Lander ES. Molecular classification of cancer: Class discovery and class prediction by gene expression monitoring. Science, 1999, 286(5439):531-537 . |
| [26] | Alon U, Barkai N, Notterman DA, Gish K, Ybarra S, Mack D, Levine AJ. Broad patterns of gene expression revealed by clustering analysis of tumor and normal colon tissues probed by oligonucleotide arrays. Proc. of the National Academy of Sciences, 1999, 96(12):6745-6750 . |
| [27] | Notterman DA, Alon U, Sierk AJ, Levine AJ. Transcriptional gene expression profiles of colorectal adenoma, adenocarcinoma, and normal tissue examined by oligonucleotide arrays. Cancer Research, 2001,61(7):3124-3130. |
| [28] | Chang CC, Lin CJ. LIBSVM: A library for support vector machines. ACM Trans. on Intelligent Systems and Technology (TIST), 2011,2(3):27. |
| [29] | Liu J, Iba H, Ishizuka M. Selecting informative genes with parallel genetic algorithms in tissue classification. In: Proc. of the Genome Informatics Series. 2001. 14-23. |
| [30] | Dettling M, Bühlmann P. Boosting for tumor classification with gene expression data. Bioinformatics, 2003,19(9):1061-1069 . |
| [31] | Krishnapuram B, Carin L, Hartemink A. Gene expression analysis: Joint feature selection and classifier design. In: Proc. of the Kernel Methods in Computational Biology. 2004. 299-317. |
| [32] | Chao S, Lihui C. Feature dimension reduction for microarray data analysis using locally linear embedding. In: Bajic VB, ed. Proc. of the 3rd Asia-Pacific Bioinformatics Conf. 2005. 211-218. |
| [33] | Model F, Adorjan P, Olek A, Piepenbrock C. Feature selection for DNA methylation based cancer classification. Bioinformatics, 2001,17(Suppl.1):S157-S164 . |
| [34] | Yu L, Han Y, Berens ME. Stable gene selection from microarray data via sample weighting. IEEE/ACM Trans. on Computational Biology and Bioinformatics (TCBB), 2012,9(1):262-272 . |
| [35] | Han Y, Yu L. A variance reduction framework for stable feature selection. In: Kotagiri R, ed. Proc. of the IEEE Int’l Conf. on Data Mining (ICDM 2010). Sydney: IEEE,2010.206-215 . |
| [4] | 李颖新,李建更,阮晓钢.肿瘤基因表达谱分类特征基因选取问题及分析方法研究.计算机学报,2006,29(2):324-330.计算机学报,2006,29(2):324-330. |
| [5] | 谢娟英,谢维信.基于特征子集区分度与支持向量机的特征选择算法. 计算机学报,2014,37(8):1704-1718. |
 2014, Vol. 25
2014, Vol. 25


